« SARS-CoV-2 » : différence entre les versions
Aucun résumé des modifications |
→Hypothèse de l'accident de laboratoire : analyse publiée, le 17 mars 2020, dans la revue Nature |
||
| Ligne 131 : | Ligne 131 : | ||
[[Luc Montagnier]], co-découvreur du [[Virus de l'immunodéficience humaine|VIH]], émet l'hypothèse selon laquelle le SARS-CoV-2 serait issu d'une manipulation génétique ayant adjoint des morceaux de génome du VIH. Il reprend ainsi à son compte une étude indienne déjugée<ref name="ThèseTrèsContestée">{{Lien web |auteur=William Audureau |titre=Le coronavirus, fabriqué à partir du virus du sida ? La thèse très contestée du professeur Montagnier |url=https://www.lemonde.fr/les-decodeurs/article/2020/04/17/le-coronavirus-fabrique-a-partir-du-virus-du-sida-la-these-tres-contestee-du-pr-luc-montagnier_6036972_4355770.html |date=17 avril 2020 |site=lemonde.fr |consulté le= 18 avril 2020}}.</ref>. Cette hypothèse est hautement improbable et marginale au sein de la communauté scientifique<ref name="ThèseTrèsContestée"/>{{,}}<ref>{{Lien web|auteur=Le point|titre=En liant coronavirus et VIH, le professeur Montagnier fait polémique|jour=17|mois=avril|année=2020|url=https://www.lepoint.fr/sante/en-liant-coronavirus-et-vih-le-professeur-montagnier-fait-polemique-17-04-2020-2371881_40.php|site=lepoint.fr|consulté le=17 avril 2020}}.</ref>. |
[[Luc Montagnier]], co-découvreur du [[Virus de l'immunodéficience humaine|VIH]], émet l'hypothèse selon laquelle le SARS-CoV-2 serait issu d'une manipulation génétique ayant adjoint des morceaux de génome du VIH. Il reprend ainsi à son compte une étude indienne déjugée<ref name="ThèseTrèsContestée">{{Lien web |auteur=William Audureau |titre=Le coronavirus, fabriqué à partir du virus du sida ? La thèse très contestée du professeur Montagnier |url=https://www.lemonde.fr/les-decodeurs/article/2020/04/17/le-coronavirus-fabrique-a-partir-du-virus-du-sida-la-these-tres-contestee-du-pr-luc-montagnier_6036972_4355770.html |date=17 avril 2020 |site=lemonde.fr |consulté le= 18 avril 2020}}.</ref>. Cette hypothèse est hautement improbable et marginale au sein de la communauté scientifique<ref name="ThèseTrèsContestée"/>{{,}}<ref>{{Lien web|auteur=Le point|titre=En liant coronavirus et VIH, le professeur Montagnier fait polémique|jour=17|mois=avril|année=2020|url=https://www.lepoint.fr/sante/en-liant-coronavirus-et-vih-le-professeur-montagnier-fait-polemique-17-04-2020-2371881_40.php|site=lepoint.fr|consulté le=17 avril 2020}}.</ref>. |
||
Selon une analyse publiée, le 17 mars 2020, dans la revue ''[[Nature (revue)|Nature]]'', par un groupe de cinq chercheurs de nationalités diverses (USA, UK, Australie), le SRAS-CoV-2 ne serait pas une construction de laboratoire ni un virus délibérément manipulé : {{citation|Bien que les preuves montrent que le SRAS-CoV-2 n'est pas un virus délibérément manipulé, il est actuellement impossible de prouver ou de réfuter les autres théories de son origine décrites ici. Cependant, puisque nous avons observé toutes les caractéristiques notables du SRAS-CoV-2, […] dans les coronavirus apparentés dans la nature, nous ne pensons pas qu'un type de scénario en laboratoire soit plausible<ref name="proximal origin">{{article|lang=en|url=https://www.nature.com/articles/s41591-020-0820-9#citeas|titre=The proximal origin of SARS-CoV-2|auteur=Kristian G. Andersen, Andrew Rambaut, W. Ian Lipkin, Edward C. Holmes et Robert F. Garry |périodique=nature medecine|numéro=26|passage=450-452|date=17 mars 2020|consulté le=18 avril 2020|citation=Although the evidence shows that SARS-CoV-2 is not a purposefully manipulated virus, it is currently impossible to prove or disprove the other theories of its origin described here. However, since we observed all notable SARS-CoV-2 features, […] in related coronaviruses in nature, we do not believe that any type of laboratory-based scenario is plausible proximal origin.}}.</ref>}}. |
|||
== Description moléculaire, structurelle et fonctionnelle du virus == |
== Description moléculaire, structurelle et fonctionnelle du virus == |
||
Version du 18 avril 2020 à 15:47
SARS-CoV-2
| Domaine | Riboviria |
|---|---|
| Ordre | Nidovirales |
| Sous-ordre | Cornidovirineae |
| Famille | Coronaviridae |
| Sous-famille | Orthocoronavirinae |
| Genre | Betacoronavirus |
| Sous-genre | Sarbecovirus |
| Espèce | SARSr-CoV |
Modèle:Arbre début
- Modèle:Arbre/Branche finale Espèce : SARSr-CoV
- SARS-CoV (humain ; SRAS)
- SARSr-CoV WIV1 (chauve-souris)
- SARSr-CoV HKU3 (chauve-souris)
- SARSr-CoV RP3 (chauve-souris)
- Modèle:Arbre/Branche finale SARS-CoV-2 (humain ; COVID-19)
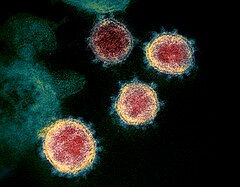
Le coronavirus 2 du syndrome respiratoire aigu sévère, désigné officiellement par le sigle SARS-CoV-2 (acronyme anglais de severe acute respiratory syndrome coronavirus 2) et parfois partiellement francisé en SRAS-CoV-2, est une nouvelle souche de l'espèce de coronavirus SARSr-CoV, découverte en décembre 2019 dans la ville de Wuhan (province de Hubei, en Chine).
C'est l'agent pathogène à l'origine d'une pneumonie atypique émergente, la maladie à coronavirus 2019 (Covid-19). La progression de cette maladie a conduit l'Organisation mondiale de la santé (OMS) à la déclarer urgence de santé publique de portée internationale le , puis pandémie le 11 mars 2020.



Le SARS-CoV-2 est apparenté au coronavirus responsable du SRAS, le SARS-CoV, et appartient comme celui-ci à l'espèce virale SARSr-CoV[2], dans le sous-genre Sarbecovirus du genre Betacoronavirus[3]. Avec ses 125 nanomètres de diamètre et un ARN très long (30 kb), c'est le plus grand des virus à ARN (« légèrement plus grand que les virus de la grippe, du SRAS et du MERS »)[4],[5]. Son ancêtre est probablement un virus de chauve-souris, qui pourrait avoir muté chez le pangolin[6].
Nom du virus et de la maladie
D'abord dénommé « coronavirus de Wuhan »[7] puis « nouveau coronavirus 2019 » (2019-nCoV), son nom officiel SARS-CoV-2[8] a été choisi le par l'International Committee on Taxonomy of Viruses (ICTV), conformément à ses recommandations générales en cas d'émergence épidémiologique[9]. La forme longue en français de l’acronyme SARS-CoV-2 est désignée par l’OMS « coronavirus 2 du syndrome respiratoire aigu sévère »[8], tandis que l’Office québécois de la langue française la désigne « coronavirus du syndrome respiratoire aigu sévère 2 »[7].
Le groupe d'étude Nidovirales de l'ICTV a proposé le nom SARS-CoV-2 au terme d'une analyse taxonomique poussée[10]. Ce travail montre notamment que le nouveau coronavirus appartient à la même espèce biologique que SARS-CoV qui avait causé l'épidémie de SRAS en 2003, même si le syndrome observé en 2019 diffère de celui-ci.
Simultanément, l'OMS donne à la maladie liée au virus le nom officiel de « maladie à coronavirus 2019 » (Covid-19[8],[11], de l'anglais coronavirus disease 2019) qui avant était informellement dénommée « pneumonie de Wuhan ». À noter que Covid-19 désigne la maladie et non le coronavirus, et s'emploie donc au féminin[12].
On trouve aussi parfois, même dans un contexte scientifique[13], l'appellation hCoV-19, pour « Human Coronavirus 2019 ».
Même si nommer différemment la maladie et l'agent qui la cause est habituel (exemple : le VIH cause le sida), et si la définition des espèces biologiques dépend d'autres éléments que ceux purement conjoncturels (exemple : Botrytis cinerea cause la pourriture grise mais aussi la pourriture noble recherchée pour la vinification des sauternes), l'apparition des deux noms le même jour a d'abord suscité quelques incompréhensions tant chez un public peu averti mais sensibilisé par une situation de crise[14], que dans la communauté scientifique[15],[16].
Selon l'historien Frédéric Vagneron, « c’est sans doute la première fois dans l’histoire que l’on a détecté un virus avant même de donner un nom à la maladie qu’il provoque »[17].
Ressources scientifiques
Les archives de prépublications, notamment bioRxiv et medRxiv, et divers forums de chercheurs, permettent une diffusion rapide – mais non formellement validée par les pairs – de l'information scientifique concernant SARS-CoV-2[18]. BioRxiv a publié le premier preprint sur SARS-CoV-2 (alors nommé 2019-nCoV) le 19 janvier[19] et medRxiv le 24 janvier[20].
Les premières publications scientifiques validées par les pairs datent du [21],[22]. Plusieurs grandes revues et grands éditeurs scientifiques, devant l'urgence de la situation, ont décidé de rendre disponibles hors abonnement un certain nombre de publications scientifiques sur le nouveau coronavirus et la pneumonie associée : c'est notamment le cas de Elsevier[23], The Lancet[24], The New England Journal of Medicine[25], Science[26], Springer-Nature[27] ou encore Wiley[28].
Symptômes chez l'humain

Les maladies que provoquent les coronavirus peuvent aller du rhume (ils en sont la seconde cause aux États-Unis[29]) à des maladies plus graves telles que le syndrome respiratoire aigu sévère (SRAS, épidémie en 2003) et le syndrome respiratoire du Moyen-Orient (MERS-CoV, épidémie en 2012).
La première description scientifique de la pathologie et de l'épidémiologie de SARS-CoV-2 date du 24 janvier 2020, faite par un consortium de médecins de Wuhan et de Pékin, et basée sur la petite cohorte des 41 premiers cas étudiés à Wuhan en décembre 2019[22]. L'âge médian des patients était de 49 ans ; aucun n'avait moins de 18 ans. Le délai médian entre l'infection et l'hospitalisation était de 7 jours (4-8 jours) ; la dyspnée s'est déclarée à 8 jours (5-13 jours) pour 51 % d'entre eux, et s'est aggravée à 9 jours (8-14 jours) pour 27 % d'entre eux ; l'admission en unité de soins intensifs est devenue nécessaire à 10,5 jours (8-17 jours) pour 39 % d'entre eux. Quelques semaines plus tard, 28 (68 %) des 41 patients étaient sortis de l'hôpital tandis que 6 (15 %) étaient décédés.
Les symptômes rapportés évoquent grossièrement ceux de la grippe saisonnière : fièvre, fatigue, toux sèche, manque de souffle, difficultés à respirer[30],[31], pneumonie, insuffisance rénale se concluant par la mort dans les cas sévères[32],[33].
On observe que 75% des premiers hospitalisés présentent une lymphopénie (manque de lymphocytes) mais des signes vitaux stables à leur admission[30]. Par ailleurs, 25% des diagnostiqués ont des symptômes sévères. Parmi les 41 premiers patients hospitalisés et diagnostiqués positifs au coronavirus 2019, la majorité étaient en bonne santé avant leur infection, un tiers étant concerné par des problèmes de santé préalables. Les trois quarts des patients toussaient, un peu plus de la moitié ressentait une gêne respiratoire, et un tiers avait des difficultés respiratoires sévères nécessitant des soins intensifs[34],[22]. Le 23 janvier 2020, l'OMS déclare que la plupart des patients décédés avaient un système immunitaire affaibli par des problèmes de santé tels que l'hypertension, un diabète ou une maladie cardiovasculaire[35].
Taux de létalité
Il reste incertain car estimer le nombre réel de cas et le nombre de décès attribuables à SARS-CoV-2 est difficile[36],[37],[38]. En outre, il peut évoluer avec des mutations du virus.
La première évaluation de létalité a été communiquée le 14 février par le Centre chinois de contrôle et de prévention des maladies (CDC chinois) d'après les 44 672 premiers cas confirmés[39] :
- il était alors estimé à 2,3 % ; la COVID-19 étant bénigne pour 81 % des patients et souvent plus dure pour les personnes âgées ou porteuses d'une autre maladie (seuls 2,2 % des cas déclarés avaient moins de 20 ans)[4], mais d'autres cas étaient symptomatiques ou non identifiés ; puisque 1,2 % des cas confirmés étaient asymptomatiques[39] ;
- l'OMS, se basant sur les chiffres fournis par les pays (infections avérées, décès...) a confirmé un taux de létalité d'environ 2 % (426 décès sur 20 588 infections)[40], taux semblant faible comparé à celui d'autres coronavirus comme le SARS-CoV ou le MERS-COV (9,8 % et 34 % respectivement[41]), mais bien plus élevée par exemple que pour la plupart des virus grippaux chez l'humain[réf. nécessaire] ;
- mais fin février, le CDC chinois a réévalué à la hausse ce taux de létalité (passé de 2,3 % à 3,46 %)[42] ;
- le taux de létalité est différent en fonction du pays et fonction de la possibilité d'accès aux services hospitaliers. Au 11 mars 2020, la létalité était de 9,26 % en Italie (5 476 décès pour 59 138 infectés[43]) contre 0,8 % en Corée du Sud (66 décès pour 7 869 infectés[44]) ;
- le taux de létalité des cas confirmés à l'échelle mondiale, communiqué par l'OMS le , est de 3,4 %[45].
Le taux de létalité est toutefois un chiffre qu'il faut interpréter avec beaucoup de précaution[46], dans la mesure où beaucoup de pays ont des taux de tests de dépistage par habitant très différents (ex. : le taux chinois est nettement plus élevé que le taux français). La proportion de contaminés asymptomatiques (et donc non décelés) est probablement supérieure à 40%, ce qui diminue beaucoup la létalité réelle par rapport à celle qui est constatée à partir du chiffre des contaminés.
Taxonomie et phylogénie (origine du virus)
Classification

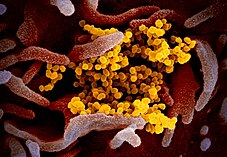
Le virus SARS-CoV-2 appartient, comme le virus du SRAS, à l'espèce SARSr-CoV (Severe acute respiratory syndrom-related Coronavirus), dans le genre Betacoronavirus et la famille Coronaviridae[10]. La morphologie des virions est typique de celle des coronavirus[31], avec notamment le halo de protubérances protéiques ("Spike") qui leur a donné leur nom ("virus à couronne").
Le nombre de génomes isolés et séquencés croît rapidement ainsi que leurs origines géographiques : ils sont 2 090 au [48] (les premiers génomes séquencés, originaires de Wuhan, l'ont été par le CDC chinois, l'Institut de biologie des agents pathogènes et l'hôpital Wuhan Jinyintan[49],[50]). Ceci a permis de rapidement montrer que SARS-CoV-2 a des similitudes avec les Betacoronavirus trouvés chez les chauves-souris[51],[52]. Il forme une souche virale génétiquement distincte des autres coronavirus humains MERS[51] ou d'autres espèces plus bénignes, mais appartenant à la même espèce biologique que le SARS-CoV[10], dans le sous-genre Sarbecovirus[53],[54],[55].
Origine
Le marché de gros de fruits de mer de Huanan, à Wuhan, a été désigné comme source initiale de l'épidémie par les autorités locales le [56]. Il a été fermé le lendemain même, le . Les premiers symptômes attribuables a posteriori à SARS-CoV-2 seraient toutefois apparus dès le chez un patient n'ayant pas fréquenté ce marché[22], de même qu'un tiers des 41 cas signalés à Wuhan en ; des experts en ont déduit que l'origine de l'épidémie pourrait être antérieure à , et ne pas se trouver sur le marché de Wuhan[57],[58]. De même, des scientifiques chinois ont affirmé, le , que l’origine de l’épidémie n’était pas à chercher dans le marché aux animaux de Huanan[59], sans toutefois fournir une alternative démontrée.
Une analyse phylogénétique et génomique précoce excluait quasiment une descendance directe de SARS-CoV-2 à partir de SARS-CoV[60]. Une origine ultime « chauve-souris » fait consensus[61],[62]. Même si le marché de Huanan vendait une grande diversité de faune sauvage avant sa fermeture[63], les chauves-souris ne semblent pas en avoir fait partie, et leur consommation n'est d'ailleurs pas traditionnelle dans la région malgré des affirmations de vidéos devenues virales sur Internet[64],[65], contribuant à une forme de xénophobie anti-asiatique développée à cette occasion. SARS-CoV-2 peut être passé d'une chauve-souris à l'humain par l'entremise d'un hôte intermédiaire de la même façon que le SARS-CoV (dont l'hôte intermédiaire aurait été la civette) ou le MERS-CoV (d'hôte intermédiaire le dromadaire). Ces voies de passage du virus vers l'espèce humaine continuent de faire débat. Une première hypothèse « serpent »[60] (bien qu'aucun coronavirus ne soit déjà connu chez les animaux à sang froid[réf. nécessaire]) a été reprise par la presse[66] mais rapidement écartée[67],[68]. Certains experts[6],[69] avancent l'hypothèse du pangolin, un mammifère en danger qui fait l'objet d'une consommation illégale en Chine et qui était en vente sur le marché de Wuhan : en effet, on trouve chez le pangolin des coronavirus quasiment identiques au SARS-CoV-2[70],[71] ; cette hypothèse ne semble toutefois pas pouvoir être retenue telle quelle puisque des virus de chauve-souris apparaissent plus proches encore[62], laissant certains observateurs supposer l'existence d'un réarrangement génétique préalable au passage chez l'humain[72].
Sur la base des séquences complètes disponibles, l'initiative Gisaid commence dès fin à distinguer une histoire évolutive de SARS-CoV-2[73] : des mutations groupées dans le temps et dans l'espace de l'épidémie, ainsi que le long du génome, si bien que fin , avec de l'ordre de 1 500 génomes comparés entre eux, on distingue assez nettement des branches asiatiques, européennes et américaines dans l'arbre de diversité du virus.
Génome
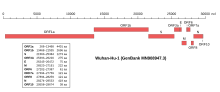
Son génome, d'abord réputé stable[74] est constitué d'un ARN simple-brin de 29 903 nucléotides. Il a été séquencé le à l'université Fudan de Shanghai en Chine[75],[76],[77],[78],[50].
En termes d'homologie, le SARS-CoV-2 est :
- à 50 % identique à celui du MERS-CoV[79] ;
- à 79,5 % identique à celui du SARS-CoV[80] ;
- à 90 % identique (99 % localement au niveau du site qui assure la fixation au récepteur cellulaire ACE2, déterminant pour la spécificité d'hôte) à un coronavirus présent chez les pangolins[81],[82],[83],[84] ;
- à 96 % identique à celui de BetaCoV/bat/Yunnan/RaTG13/2013, un coronavirus d'une chauve-souris chinoise du genre Rhinolophe : Rhinolophus affinis[51],[61],[62].
Cela laisse supposer que l'origine du virus serait chez une chauve-souris ou peut-être un pangolin[52],[68],[4],[61],[62].
Hypothèses liant l'émergence du virus aux activités humaines
Si les conditions d'émergence du SARS-CoV-2 n'ont pas été mises en évidence à ce jour, l’hypothèse d’une origine synthétique du SARS-CoV-2 est écartée. Le Monde indique que « s’il est bien possible de créer de toutes pièces des virus, tous les indices convergent ici vers une origine naturelle de ce nouvel agent infectieux. Pour acquérir cette certitude, les scientifiques ont étudié son histoire, dont une grande partie est inscrite dans ses gènes »[85].
Certains scientifiques déclarent dans les médias que l'émergence du SARS-CoV-2, comme les virus de zoonoses d'une manière générale, est probablement liée à l'anthropisation ou aux marchés d’animaux sauvages[86].
Hypothèse du lien avec l'anthropisation
Bruno Canard, directeur de recherche CNRS, relève que « les coronavirus responsables des épidémies de SRAS (syndrome respiratoire aigu sévère), du MERS (Middle East Respiratory Syndrom) et du SARS-CoV-2 proviennent de virus issus du monde animal qui ont franchi la barrière inter-espèces ». Il estime ainsi que « l’anthropisation globale du monde favorise l’émergence de virus qui étaient jusqu’à présent cachés chez les animaux et étaient maintenus dans leurs habitats naturels par une biodiversité importante ». Il souligne que « plusieurs études ont d’ailleurs démontré que la biodiversité est le meilleur rempart contre les émergences virales »[87].
Selon le microbiologiste Philippe Sansonetti, le SARS-CoV-2 appartient aux « maladies d’anthropocène : pour l’essentiel voire exclusivement, elles sont liées à la prise en main de la planète et à l’empreinte que l’homme y laisse. Ce qui est valable pour le climat, pour l’environnement, est tout aussi valable pour les maladies infectieuses, en particulier émergentes, et les trois sont liés »[88].
Selon Jean-François Guégan, chercheur en microbiologie et spécialiste de la transmission des agents infectieux, le SARS-CoV-2 est « un boomerang qui nous revient dans la figure », dont les origines de la propagation sont liées au modèle économique, à « la destruction des habitats naturels » et au « non-respect de leur biodiversité », et « n’ont rien à voir avec des causes strictement sanitaires »[89].
Serge Morand, écologiste de la santé, estime que la circulation de nouveaux pathogènes est favorisée à la fois par une grande biodiversité, une plus grande proximité entre les humains et une faune sauvage en recul, par l’augmentation du nombre d'animaux d’élevage, et plus largement par « la libéralisation complète de nos économies » et le développement des « cultures destinées au commerce international, et qui sont complètement sorties de leur aire écologique »[89]. Selon lui, « la Chine du Sud et l’Asie du Sud-Est présentent toutes [les] caractéristiques de biodiversité, d’intensification agronomique et d’élevage (qui entraîne une crise de la biodiversité) et d’intégration dans le marché international et touristique », favorables à « l’émergence d’une nouvelle maladie infectieuse à potentiel pandémique ». [90].
Hypothèse du lien avec l'exploitation d'animaux sauvages
Pour Étienne Simon-Lorière, responsable du laboratoire de génomique évolutive des virus à ARN à l'Institut Pasteur, le scénario du passage du virus à l'humain est simple ; « des animaux porteurs du virus l'ont excrété par les voies aériennes (nez, bouche), des personnes ont manipulé ces animaux les mains nues, puis se sont porté les mains au visage. On ne sait pas si cela a eu lieu une, deux ou cent fois ; mais pour que l'infection ait fonctionné, le virus a dû franchir au moins une fois l'étape de se lier de manière efficace aux cellules humaines. […] Une fois cette étape franchie, le virus avait atteint l'espèce humaine[91] ».
Didier Sicard, spécialiste des maladies infectieuses, déplore « l’indifférence aux marchés d’animaux sauvages dans le monde », qui constituent selon lui le « point de départ » de la pandémie, et appelle à la constitution d'« une sorte de tribunal sanitaire international » chargé de faire respecter l'interdiction du trafic d'animaux sauvages[92]. Il appelle également à renforcer les recherches sur les chauves-souris et à « reconstituer le parcours épidémiologique qui fait que la chauve-souris tolère des coronavirus depuis des millions d’années, mais aussi qu’elle les disperse »[92].
L'écologue Florian Kirchner, de l'UICN-France, préconise à court terme, de renforcer la lutte contre le braconnage et le trafic illégal d'animaux sauvages, notamment dans les marchés d'Asie du Sud-Est, de Chine ou d'Afrique subsaharienne, estimant que « la présence de chauves-souris sur ces marchés est une bombe à retardement »[93].
Selon Alexandre Hassanin, chercheur en biologie de l'évolution, tout porte à croire que « l’épidémie est la conséquence directe du trafic illégal de faune sauvage » car « les nombreux virus de la lignée SARS-CoV-2 identifiés chez des pangolins destinés à la vente prouvent que ces virus circulent depuis plusieurs années déjà dans les marchés des provinces du sud de la Chine et très probablement aussi dans les provinces du Nord, comme celle de Hubei ». Il juge « urgent d’interdire non pas temporairement mais définitivement le commerce de vertébrés sauvages potentiellement responsables de zoonoses »[94].
Vincent Racaniello, professeur de microbiologie et immunologie, émet l'hypothèse que l'émergence du virus pourrait être due à des agriculteurs qui récoltent les déjections de chauves-souris dans des grottes pour les utiliser en tant qu'engrais[95].
Hypothèse de l'accident de laboratoire
Selon Fox News, le virus aurait pu s'être échappé par accident de l'Institut de virologie de Wuhan. L'hypothèse est évoquée par Mike Pompeo, secrétaire d'État des États-Unis citant une enquête en cours de leur administration, ce que confirme le président Donald Trump[96]. Richard Ebright (en), expert américain en biosécurité, estime que « les preuves laissent une base pour exclure que le Covid-19 soit une construction de laboratoire, mais aucune base pour exclure un accident »[97].
Luc Montagnier, co-découvreur du VIH, émet l'hypothèse selon laquelle le SARS-CoV-2 serait issu d'une manipulation génétique ayant adjoint des morceaux de génome du VIH. Il reprend ainsi à son compte une étude indienne déjugée[98]. Cette hypothèse est hautement improbable et marginale au sein de la communauté scientifique[98],[99].
Selon une analyse publiée, le 17 mars 2020, dans la revue Nature, par un groupe de cinq chercheurs de nationalités diverses (USA, UK, Australie), le SRAS-CoV-2 ne serait pas une construction de laboratoire ni un virus délibérément manipulé : « Bien que les preuves montrent que le SRAS-CoV-2 n'est pas un virus délibérément manipulé, il est actuellement impossible de prouver ou de réfuter les autres théories de son origine décrites ici. Cependant, puisque nous avons observé toutes les caractéristiques notables du SRAS-CoV-2, […] dans les coronavirus apparentés dans la nature, nous ne pensons pas qu'un type de scénario en laboratoire soit plausible[100] ».
Description moléculaire, structurelle et fonctionnelle du virus


Chaque virion SARS-CoV-2 mesure de 50 à 200 nanomètres de diamètre, ce qui le fait classer parmi les gros virus[101].
Le premier génome entièrement séquencé est celui de la souche Wuhan-Hu-1, diffusé le 23 janvier 2020, deux semaines après la découverte de la maladie[75].
Le génome viral est un ARN monocaténaire de 29903 nucléotides, ce qui est typique d'un Betacoronavirus.
Structure protéique du virus
Certaines parties de la structure réelle ne sont pas représentées.
Sont représentés les domaines protéiques N-terminal (lettre N) à C-terminal (C) :
Domaine N-terminal (bleu) ; domaine de liaison au récepteur ACE2 (magenta) ; structure générale (cyan), hélice centrale (orange) ; le domaine du connecteur qui ancre la protéine de pointe à l'enveloppe lipidique du virus (violet). Les liaisons disulfure sont en Jaune, et les glucides en Rouge. Le gris présente la membrane lipidique du virus
Comme d'autres coronavirus, le SARS-CoV-2 possède quatre protéines structurales :
- protéines S (dites protéine spike ou protéine de pointe) : elles forment les protubérances de la « couronne », présumée essentielles pour lier le virus à un (ou plusieurs) récepteurs(s) en surface d'une cellule (après activation par une autre protéine de la membrane cellulaire)[102].
Ces protéines semblent être l'un des principaux déterminants du tropisme viral (« le tropisme d’un virus se définit comme l’ensemble des cellules cibles et permissives à ce virus. Le connaître permet de déterminer le ou les organes cibles, ainsi que la ou les espèces animales pouvant être infectées »[103] ; les virus à ARN mutent facilement ; quand une mutation permet au virus de changer son tropisme, il peut, soit franchir la barrière des espèces et infecter un nouvel hôte (humain, porc par exemple), soit cibler un autre organe (péritonite infectieuse fatale du chat et du furet par exemple, avec un autre coronavirus). La publication du génome viral a permis de la modéliser (voir ci-contre).
Elle a aussi été décrite au niveau atomique par la microscopie électronique cryogénique[104],[105].
Elle semble plus efficace que celle du SARS-CoV (affinité plus élevée pour l'ACE2 humain, un homologue de l'ACE)[104], ce qui expliquerait pourquoi la Covid-19 se répand beaucoup plus vite que le SRAS[106] Un segment de cette protéine spike semble caractéristique du SARS-CoV-2[107]. - protéine E (enveloppe) ;
- protéine M (membrane) ;
- protéine N (nucléocapside) ; c'est elle qui enveloppe et protège l'ARN viral (le code génétique du virus).
Les protéines S, E et M constituent, ensemble, l'enveloppe virale[102].
Outre un segment de la protéine S (spike), un court gène supplémentaire semble également spécifique à ce virus (segment dont la réalité biologique et le rôle éventuel étaient fin janvier encore à démontrer)[107].
Mécanisme infectieux
Il est encore incomplètement compris, mais:
- des expériences de modélisation de la structure protéique des protéines des « pointes » du virus SARS-CoV-2 ont rapidement suggéré que ces pointes ont une affinité pour l'enzyme de conversion de l'angiotensine 2 (ACE2) des cellules humaines. Le virus semble les utiliser pour se fixer sur la cellule et y entrer[108].
- Au 22 janvier 2020, un groupe chinois travaillant avec le génome complet du virus, et un groupe aux États-Unis utilisant des méthodes de génétique inverse, confirment, de manière indépendante et expérimentale, que l'ACE2 peut effectivement être récepteur du SARS-CoV-2[109],[110],[111],[112],[113],[114].
- Au 23 janvier 2020, deux groupes chinois pensent que la protéine S a une affinité suffisante avec le récepteur du SARS-CoV (enzyme de conversion de l'angiotensine 2, ACE2) pour l'utiliser comme mécanisme d'entrée cellulaire[115]. Trois autres groupes de chercheurs arrivent à la même conclusion[51],[116],[117]. Le récepteur cellulaire de MERS-CoV, DPP4, ne semble pas reconnaître la protéine Spike de SARS-CoV-2[117], ce qui est en accord avec la plus grande proximité moléculaire de SARS-CoV-2 avec SARS-CoV qu'avec MERS-CoV.

- pour permettre au virus d'entrer dans la cellule, la protéine de pointe (S) du SARS-CoV-2 doit cependant d'abord être activée (maturée). Elle l'est par une protéase transmembranaire de la surface de la cellule-hôte ; une protéase cellulaire à sérine dite TMPRSS2 ou sérine2[118].
Après qu'un virion SARS-CoV-2 s'est fixé à une cellule cible, cette protéase TMPRSS2 de la cellule va couper (cliver disent les biologistes) l'extrémité de la protéine de pointe du virus, exposant alors un « peptide de fusion » (Membrane fusion protein). La TMPRSS2 jouait un rôle identique pour le SARS-CoV précédent, ce qui pourrait aider à trouver des pistes de traitements antiviraux[117] :
- après que le virus a fusionné avec la cellule, son ARN y est libéré, utilisant le matériel intracellulaire pour produire des copies du virus. Ces copies (virions) sortent ensuite de la cellule pour se disséminer et pour infecter d'autres cellules ;
- la protéine S aurait une autre voie d'activation possible et donc un autre moyen d'entrer dans ses cellules-cibles. Le 14 mars 2020, une équipe de 30 chercheurs chinois dont six membres de l'Institut de Biotechnologies de Pékin et 24 de la Quatrième université de médecine militaire de Xi'an) a prépublié un article montrant que le SARS-CoV-2 disposerait d'une autre voie d'entrée dans ses cellules-cibles : la basigine (BSG, ou CD147, ou EMMPRIN qui a aussi d'autres synonymes). Il s'agit d'une protéine transmembranaire de la Superfamille des immunoglobulines[120]. La basigine est une protéine inductrice de métalloprotéinase, à matrice extracellulaire, aussi dénommée « cluster de différenciation 147 » (CD147), codée par le gène BSG chez l'homme[121],[122],[123]. Elle est connue comme déterminante du système de groupe sanguin Ok et impliquée dans la croissance tumorale[124],[125],[126] et joue un rôle dans certaines infections virales[127] ; on a notamment montré en 2011 qu'elle est un récepteur essentiel de la surface des globules rouges, récepteur ciblé par Plasmodium falciparum, le parasite qui cause le paludisme humain[128],[129]. Outre sa capacité d'induction de métalloprotéinases, la basigine régule des fonctions essentielle, telles que la spermatogenèse, l'expression du transporteur de monocarboxylate (un transporteur transmembranaire) et la réactivité des lymphocytes[130].
En mars 2020, son rôle dans la COVID-19 est encore mal cerné[120], mais il avait déjà été repéré 15 ans plus tôt pour le SRAS[131]. Le méplazumab (anticorps anti-CD147 humanisé) a été testé in vitro contre des virus SARS-CoV-2 infectant des cultures cellulaires[132]. (cultures de cellules Vero E6) et le méplazumab a significativement inhibé l'infection des cellules hôtes par le virus (avec une CE50 de 24,86 μg/mL et une CI50 de 15,16 μg/mL) ; la microscopie immunoélectronique, et plusieurs tests (co-immunoprécipitation, test Elisa) ont confirmé que les deux protéines se lient, avec une constante d'affinité évaluée à 1,85 × 10-7M[120]. Selon les auteurs, empêcher cette liaison pourrait être une cible pour des antiviraux spécifiques[120].
- Le SARS-CoV-2 produit au moins trois facteurs de virulence favorisant l'excrétion de nouveaux virions à partir des cellules hôtes, et inhibant la réponse immunitaire[102].
Ces caractéristiques virales causent notamment :
- une réponse inflammatoire excessive du système immunitaire inné[133] ;
- des effets neurologiques, sources de maux de tête, nausées et vomissements[134] ; un nombre croissant de preuves indique que les coronavirus ne colonisent pas que les voies respiratoires, mais aussi le système nerveux central en y faisant des dégâts neurologiques. Ainsi, le SARS-CoV a déjà été associé à des atteintes cérébrales et notamment du tronc cérébral chez des humains, comme dans le modèle animal en laboratoire. Et d'autres coronavirus peuvent se propager par des voies synaptiques vers le centre cardiorespiratoire situé dans le tronc cérébral, à partir de mécanorécepteurs et de chimiorécepteurs présent dans les poumons et reliés au cerveau et à la moelle épinière[134].
Épidémiologie
Incubation
La période d'incubation de SARS-CoV-2 est estimée entre deux et quatorze jours[135], parfois plus longue (jusqu'à 24 jours)[136].
Selon une étude publiée le 10 mars, après relecture par les pairs[137], et portant sur 181 cas documentés (avec une surreprésentation possible des cas les plus sévères d’après les auteurs), la période d'incubation médiane est estimée à 5,1 jours, tandis que 97,5 % de ceux qui développent des symptômes le font en moins de 11,5 jours ; cette étude suggère que sur 10 000 cas, 101 développent des symptômes au-delà des 14 jours de suivi actif et de confinement.
Plus de la moitié des contaminations serait le fait de porteurs asymptomatiques[138].
Durée de survie du virus hors de l'organisme
Un coronavirus — hors organisme — reste infectieux environ deux à trois heures dans l'air (en milieu sec)[139] et peut-être jusqu'à trois jours en milieu humide, ce qui exclut tout risque de contamination par colis voyageant entre continents autrement que directement en avion : les trajets les plus rapides entre la Chine et les États-Unis s'effectuent en quatre jours[140],[141].
Sur des surfaces lisses non poreuses (acier inoxydable, plastique, céramiques, verre...), des travaux antérieurs ont montré que SARS-CoV, très proche, peut rester infectieux jusqu'à neuf jours[142], sauf s'il s'agit de cuivre, laiton, bronze qui sont naturellement biocides pour de nombreux organismes ou microbes.
Pour SARS-CoV-2, la durée de demi-vie (temps nécessaire pour que la moitié des virus soient inactivés) est dans un environnement moyen d'environ treize heures sur de l'inox et seize heures sur du polypropylène[139].
Le papier/carton (non plastifié) ou la plupart des textiles de vêtements non-imperméables ne sont pas considérés comme « lisses », et de manière générale les virus de ce type y survivent bien moins longtemps que sur une surface lisse de métal ou plastique[139].
Au , selon les CDC américains, rien n'indique que le virus se propage par l'eau potable, les piscines ou les bains à remous[143].
Vulnérabilité du virus au savon et aux désinfectants
À la différence des norovirus ou d'autres virus courants protégés par une enveloppe protéique plus ou moins solide, tous les coronavirus (dont le SARS-CoV-2) sont très vulnérables au savon, aux solutions hydro-alcooliques et aux désinfectants ménagers courants, ainsi qu'aux désinfectants industriels et hospitaliers adaptés ; les coronavirus déposés (via des gouttelettes ou des aérosols) sur les surfaces intérieures sont immédiatement détruits car leur enveloppe protectrice n'est qu'une simple couche de lipides[143].
Pour le SRAS, un temps de contact d'une minute avec de l'eau de Javel diluée à 0,1 % ou avec une solution à 62–71 % d’éthanol réduisait aussi significativement l'infectivité des virus déposés sur des surfaces lisses ; mi-, on espère qu'il en va de même pour le SARS-CoV-2[142].
Historique (dispersion du virus)
Dans les premières semaines de l'épidémie de 2019-2020, le virus s'est propagé via des voyageurs vers différents pays d'Asie[144],[145],[146], le 21 janvier en Amérique du Nord[147], le 24 janvier, en Australie[148], en Europe[149],[150], le 16 février en Afrique[151] et le 26 février en Amérique du Sud[152]. Il s'agit là des premiers cas recensés: en réalité le virus est probablement arrivé plus tôt sur chacun de ces territoires, en y passant inaperçu: peut-être dès début janvier en Europe (Italie) par exemple[153],[154]. Le quotidien new-yorkais The New York Times a proposé une infographie montrant comment la dispersion du virus a pu précéder les mesures de restriction des transports pourtant les plus drastiques et les plus rapides jamais mises en œuvre pour stopper une épidémie[155].
À la date du 27 mars 2020, on compte 24 110 décès et 123 268 guérisons pour 537 042 cas confirmés[13] (le cap des 100 000 cas est franchi le 6 mars), le tout principalement dans la province du Hubei. Ces chiffres constituent probablement une large sous-estimation de l'ampleur de l'épidémie[156],[157], la maladie restant souvent discrète, notamment chez les sujets jeunes, et les tests de dépistage n'étant pas appliqués systématiquement à tous les cas suspects.
Le , la transmission interhumaine a été confirmée dans le Guangdong, en Chine, selon Zhong Nanshan, chef de l'équipe de la commission de la santé enquêtant sur l'épidémie[158]. La première description scientifiquement détaillée d'une transmission interhumaine au sein d'une famille a été publiée par une équipe de médecins de Hong Kong et de Shenzhen le 24 janvier 2020[21]. Le taux de reproduction (Ro) préliminaire de SARS-CoV-2, c’est-à-dire le nombre moyen de personnes infectées par un patient, a été estimé entre 1,4 et 2,5 par l'OMS le 23 janvier 2020[159]. Il serait comparable à celui de SARS-CoV et de la grippe de 1918, compris entre 1,4 et 3,8[160].
Situation épidémique
En décembre 2019 et janvier 2020, l'épidémie est encore principalement concentrée sur la Chine et touche surtout la province du Hubei. Le premier cas exporté hors de la Chine continentale est découvert le 13 janvier en Thaïlande[161]. La situation évoluant rapidement au niveau mondial, elle est déclarée urgence de santé publique de portée internationale par l'OMS le 30 janvier 2020[162]. C'est la sixième fois seulement que l'OMS déclenchait ce niveau d'alerte dans son histoire[réf. nécessaire].
Mi-février 2020, le plus important foyer hors de Chine était le paquebot de croisière Diamond Princess, en quarantaine dans le port de Yokohama[163]. Dans la troisième semaine de février toutefois, des foyers secondaires d'importance ont émergé en Corée du Sud, en Italie et en Iran[164], faisant craindre une installation durable de l'épidémie au niveau mondial[165]. Cette crainte se confirme début mars : le 11 mars, le directeur général de l'OMS déclare que « la COVID-19 peut être qualifiée de pandémie »[166] et le 15 mars, le nombre de cas hors de Chine dépasse celui dénombré en Chine continentale[13].
Malgré la difficulté à tenir un décompte précis dans une situation chaotique, l'Université Johns Hopkins (Baltimore, MD) rend compte quasiment en temps réel de l'évolution de l'épidémie[13]. Quelques dates marquantes :
- Le 1er février, un premier décès hors de Chine est déclaré aux Philippines : c'est un touriste venu de Wuhan où il a probablement été contaminé[167].
- Le 15 février, le premier décès hors d'Asie, survenu en France, est également un cas importé de Chine[168].
- Le 6 mars, l'Italie devenait le second pays en nombre de décès après la Chine et le 19 mars, elle comptait plus de décès que la Chine.
- Le 26 mars, les États-Unis devenaient le premier foyer en nombre de cas[169].
Traitement et vaccins
L'essentiel de la lutte contre la Covid-19 repose sur des mesures épidémiologiques : diagnostic fiable et rapide de la maladie en période d'incubation, mesures de confinement (qui peuvent aussi être un facteur de diffusion intragroupe[170]).
Traitements antiviraux
Le SARS-CoV-2 étant de nature virale, les antibiotiques et les bactériophages sont inadaptés, étant donné qu'ils ne traitent que les pathologies bactériennes. Toutefois, les bactéries étant plus susceptibles d'adhérer aux cellules infectées par un virus tel le coronavirus[171], on observe une surinfection bactérienne, en particulier par le Streptococcus pneumoniae dans 10 % des cas d'infection virale[31]. Dans cette situation, la prescription d'un antibiotique adapté à la sensibilité de la bactérie se justifie donc.
À l'image des recommandations de Santé publique France qui recommande "un traitement précoce par antiviral pour réduire la sévérité de la grippe"[172], on peut envisager l'utilisation d'antiviraux pour Sars-CoV-2, ou comme pour le Vih d'une association d'entre eux[173].
Aucun traitement antiviral spécifique n'a cependant été validé par des études largement reconnues[174], mais des antiviraux existants pourraient être repositionnés[175], y compris des inhibiteurs de protéase comme l'indinavir, le saquinavir et le lopinavir/ritonavir[176], ou encore le nelfinavir[177]. L'efficacité de l'inhibiteur de polymérase sofosbuvir, utilisé contre l'hépatite C, est également en cours d'évaluation en janvier 2020[178]. Le 2 février, des médecins thaïlandais ont annoncé avoir traité un patient avec succès en utilisant une combinaison des anti-HIV lopinavir et ritonavir ainsi que l'anti-grippal oseltamivir[179],[180]. Un analogue de nucléoside comme le remdésivir fait également l'objet de recherches à la suite d'un résultat positif[181]. Les traitements reçus par les patients concernent donc essentiellement l'atténuation des symptômes de fièvre, toux et dyspnée, afin de favoriser leur guérison spontanée[30],[13],[182]. Le Centre chinois de contrôle et de prévention des maladies a démarré dès janvier l'évaluation de l'efficacité de traitements existants contre les pneumonies[183], l'efficacité ayant été montrée in vitro[184]. Les médicaments présentant des essais thérapeutiques en phase IV (la plus avancée) sont l'umifénovir (Arbidol) et le lopinavir[185]. Le mésylate de camostat aurait une action inhibitrice de l'entrée du virus[186], en particulier s'il est utilisé avec la protéase E-64d[187]. En effet, une enzyme nécessaire à la réplication virale, la protéase à sérine TMPRSS2, est inactivée par l'utilisation d'une thérapie contre l'inflammation chronique du pancréas[188].
Le 22 mars l'Inserm lance l'étude « Discovery » portant sur 3 200 patients pour tester l'efficacité du remdésivir, lopinavir, ritonavir et de l'hydroxychloroquine, dans le cadre du consortium REACTing piloté par l'alliance Aviesan[189],[190]. On peut s'interroger sur la démarche consistant à lancer des études cliniques, qui ne soient pas précédées par des études pilotes, ou ne s'appuyant pas sur analyses moléculaires préalables. Ces dernières, en effet recommandaient de tester en priorité le pemirolast (en)[191] ou l'anisotine[192], tandis qu'une étude, clinique cette fois, avait déjà donné le 23 mars des résultats décevants pour l'association "lopinavir-ritonavir"[193]. Le même jour l'OMS annonce le lancement d'une étude sur l'efficacité de quatre molécules dont la chloroquine et son dérivé hydroxy[194]. Le 25 mars, l'Agence nationale de la recherche annonce le résultat de l'appel à projets "Flash" sur la Covid-19 dont un des axes est le traitement antiviral, et le financement immédiat de 44 projets dans ce cadre[195]. Un tel déploiement de moyens de recherche focalisés dans un délai aussi court (moins de trois mois après la première mention de la maladie, le à Wuhan) est inédit.
Chloroquine et hydroxychloroquine
Un repositionnement constituant une première piste est celui d'une molécule initialement antipaludique, la chloroquine, dont l'effet anticoronaviral in vitro avait déjà été démontré sur le SRAS[196],[197], et sa variante, l'hydroxychloroquine[198]. Le mode d'action du traitement s'expliquerait par une modification d'une protéine d'attachement sur la tête du virus, ce qui limiterait sa réplication[199] tout en contrôlant les lymphocytes NK (Natural Killer)[200].
Le 19 février, une lettre parue dans la revue BioScience Trends indique qu'à l'issue des premières études cliniques la commission nationale Chinoise de la Santé recommande[201] l'utilisation du phosphate de chloroquine dans le traitement du virus[202]. Ces données sont cependant encore limitées notamment en raison de la petite taille de l'échantillon statistique (100 patients)[203]. Un consensus d'experts chinois recommande le 20 février d'inclure le phosphate de chloroquine dans les recommandations de prise en charge des patients sans contre-indication à la chloroquine, à raison de 500 mg deux fois par jour pendant 10 jours pour les patients diagnostiqués comme des cas légers, modérés et sévères de maladie[203],[204]. Il est cependant à noter que la prise de chloroquine à haute dose peut provoquer des effets secondaires potentiellement sérieux, en particulier celui d'une intoxication aiguë susceptible de déclencher des problèmes cardiaques ou respiratoires, et que la dose recommandée par les chercheurs chinois se rapproche de cette zone de risque[203]. Une méta-analyse des études en cours souligne quant à elle l'absence d'effet secondaire notable[205].
Le 25 février, le professeur Didier Raoult recommande l'utilisation de l'hydroxychloroquine pour les patients porteurs du virus[206], prédisant même une « fin de partie » pour le virus[207],[208]. Sa déclaration est vivement critiquée par des médecins et scientifiques français sur les réseaux sociaux pour son manque de pondération[203] vis-à-vis de la limitation des données cliniques et des risques liés à la prise de chloroquine. L'Inserm tempère également et rappelle qu'aucune prise de médicament quel qu'il soit n'est anodine, et qu'il ne faut pas se faire prescrire la molécule pour l’utiliser en automédication[209],[210]. Un premier mort suite à une automédication hasardeuse est d'ailleurs rapidement signalé[211].
Le 6 mars, une étude pilote réalisée en Chine dans le cadre d'un essai randomisé ne détecte pas d'effet d'un traitement à l'hydroxychloroquine sur la durée d'hospitalisation, la baisse de fièvre ou l'état radiologique des poumons[212].
Le 17 mars, quelques jours avant la parution d'une pré-publication de l'équipe du Pr Raoult[213] et de la publication finale[214], le journal Les Échos titre : « Exclusif - Coronavirus : On sait guérir la maladie »[215], faisant état des résultats d'une étude préliminaire qui semble montrer un potentiel thérapeutique très important. Ce travail suscite naturellement un grand espoir, repris jusqu'à la tête de l'exécutif américain : le président Trump déclare contre l'avis de ses conseils « I'm a smart guy; I feel good about it » (« je suis un malin; je le sens bien »)[216]. Cet emballement médiatique ne doit pas faire oublier que l'essai clinique du professeur Raoult se base sur un échantillon de 24 personnes (selon l'annonce des Échos ; en réalité[217] seulement vingt alors que six patients initialement sous traitement sont sortis de l'étude en cours de route, dont certains suite à une aggravation de leur état allant pour l'un d'entre eux jusqu'au décès). La communauté scientifique exprime très rapidement de fortes réserves sur la qualité de ce travail[218],[219],[220],[221], au-delà même de son faible effectif imposé par l'urgence: étude non randomisée (les cas contrôles et les cas traités ne sont pas dans le même état initial), sans double aveugle ni groupe placebo, sans description de l'état clinique des patients, processus de validation éditoriale raccourci et potentiellement biaisé par un conflit d'intérêts[220].Le recrutement de patients sur la base d'un test Pcr, compte tenu des spécificité et sensibilité du test, donne un taux de faux positifs de 50%, ce qui introduit un biais de sélection, si on compare sans précaution la mortalité à celle obtenue dans d'autres études. Il faut toutefois souligner que l'étude a été réalisée dans l'urgence, plus avec le caractère d'une étude pilote, et qu'il est éthiquement difficile[222] de mener une étude en double aveugle quand la vie de patients est en jeu. Fin mars, l'IHU-Méditérannée indique que le taux de létalité des 2600 patients détectés positivement à l'infection Covid-19 et traités par l'Institut s'élève à 0,38 %[223] (la mortalité dans les hôpitaux en Ile-de-France est de 2 %[224],[225]) et met en ligne la prépublication d'une étude portant sur 80 patients[226]. La comparaison des résultats de cette étude avec l'étude Discovery de l'Inserm, devra être faite avec prudence dans la mesure où cette dernière n'utilise pas par exemple de traitements antibiotiques associés (azithromycine, ceftriaxone). En effet, l'azithromycin est connue par ailleurs pour avoir un effet antiviral en particulier dans le champ pulmonaire[227],[228] et par sa capacité à éviter[229] un choc cytokinique, (inflammation caractérisée par une augmentation de l'interleukine-6, l'interleukine-8, l'interleukine-1ß et TNF-a). L'étude Hycovid dirigée par le Chu d'Angers, dont les résultats seront connus mi-mai, permettra cependant une comparaison de ses résultats à ceux de l'IHU-Méditérannée car ils seront basés sur un protocole médical analogue[230]. En effet, à ce jour seul un médecin de la ville de New-York aurait communiqué les résultats d'un traitement semblable à celui pratiqué par l'IHU-Méditérannée[231].
Vaccins à l'étude
À plus long terme, plusieurs équipes internationales travaillent à la mise au point ou à l'adaptation d'un vaccin[232],[233],[234]. Même si l'épidémie de SRAS a finalement été maîtrisée sans vaccin[235], la proximité moléculaire de SARS-CoV-2 avec SARS-CoV est un atout puisqu'au-delà d'indéniables différences entre ces deux virus[236], elle permet de capitaliser sur les connaissances acquises alors[237] (faisant au passage regretter que les recherches sur la mise au point d'un vaccin anti-SARS-CoV aient été interrompues lorsque ce premier virus a été considéré éradiqué).
En , un vaccin est en cours d'élaboration en Russie[238],[239], et un autre est préparé par la firme pharmaceutique Johnson & Johnson, mais ne serait pas disponible avant un an[240]. Début février 2020, une équipe de recherche de l'Imperial College de Londres a fait une percée significative en réduisant une partie du temps de développement normal du vaccin « de deux à trois ans à seulement quatorze jours »[241],[242]. L'équipe de l'Imperial College était alors au stade de test du vaccin sur les animaux[241].
À la même période, en France, dans le cadre d'une recherche vaccinale concernant le MERS-CoV en cours depuis 2012, l'institut Pasteur pourrait utiliser et adapter ces vaccins pour SARS-CoV-2. Plusieurs équipes de l'Institut Pasteur travaillent sur son adaptation et seraient en mesure de produire ce vaccin en cas d'épidémie[243].
À la date du 8 avril, pas moins de 115 vaccins différents étaient en projet sur l'ensemble du globe, dont 78 activement; 16 projets étaient entrés en phase pré-clinique et 5 en Phase I[234]. Au moins 9 approches tecxhnologiques différentes sont suivies dans l'ensemble de ces projets, mais les plus suivies dans les 16+5 projets engagés dans des essais cliniques sont les protéines recombinantes (6+1 projets), l'ARN (4+1 projets) et le vecteur viral non réplicatif (2+2 projets)[234].
L'institut Robert-Koch préconise en outre, comme protection indirecte, la vaccination contre le pneumocoque des personnes âgées, en particulier autour des foyers d'infection[244]. Plusieurs études sont démarrées pour vérifier si le BCG ne pourrait pas diminuer la gravité de la maladie[245].
Action sur l'enzyme de conversion de l'angiotensine 2
Les récepteurs ACE2 sont à la fois le point d'entrée du virus et dans les cellules pulmonaires le lieu de la conversion de l'Angiotensine 2 en Angiotensine 1-7[246]. L'entrée du virus diminue le nombre de récepteurs Ace2 à la surface des cellules ce qui empêche la conversion de l'Angiotensine 2, et provoque l'inflammation locale. Cette inflammation est provoquées par la capture de l'Angiotensine 2, par les récepteurs AGTR1 des poumons qui provoquent le déclenchement des cytokines[247]. Un étude teste donc un traitement sur l'effet d'un antagoniste des récepteurs AT1 [248],[249],[250]. Un niveau élevé d'homocystéine pourrait donc être un marqueur défavorable de la réponse à l'infection, dans la mesure où il est implique un niveau d'expression élevé des récepteurs AT1[251],[252]. Il a par ailleurs plutôt moins de récepteurs Ace2 dans les poumons que dans les intestins, et pas en moyenne moins de récepteurs dans les poumons suivant l'âge [253]. Ceci montre que c'est au niveau pulmonaire que la réaction du virus est surtout pathologique. La fibrose pulmonaire dépendrait de l'équilibre en récepteur AT1, et AT2: les récepteurs AT1 seraient 5 fois plus présents en cas de fibrose, qu'à la normale [254], tandis que l'effet anti-fibrotique des recepteurs AT2[255],[256] pourrait difficement s'exprimer en cas d'age avancé [257]. Les inhibiteurs de l'enzyme de conversion ont plus ou moins d'affinité avec les récepteurs AT1, ou AT2 et un essai clinique testant ces inhibiteurs devrait plutôt porter sur ceux qui ont plus d'effet sur les récepteurs AT1 (ex:Valsartan [258]). En effet les récepteurs AT1 ont une action pro fibrotique et AT2 anti fibrotique[259].
D'après certaines études, l'injection d'ACE2, point d'attache du virus aux cellules eucaryotes[260],[261], ralentirait la pénétration du virus et protégerait les poumons. Le traitement a été essayé avec succès sur des souris [262] dans le cas d'une fibrose pulmonaire. En effet, l'enzyme Ace2, ralentit l'inflammation[263]. Le virus semble diminuer l'activité des récepteurs ACe2, ce qui accroîtrait l'action de l'ace2 circulante[264].APN01 est le non d'un médicament dont les essais débutent en avril, basé sur l'augmentation de l'Ace2 circulante[265]. Pour d'autres, au contraire, l'ACE2 circulant contribuerait à augmenter le nombre de récepteurs ACE2 dans les cellules, ce qui renforcerait l'action du virus[266],[267]. Ainsi, un risque accru d'infection par SARS-CoV-2 des patients souffrant d'hypertension ou de diabète en général a été suggéré[268], et en particulier traités par les inhibiteurs de l'enzyme de conversion[269], qui présentent un nombre accru de récepteurs de l'enzyme ACE2. Néanmoins les approches basées sur une diminution des récepteurs ACE2 cellulaires paraissent risquées car elles pourraient altérer la fonction pulmonaire[270]. Des approches thérapeutiques basées sur l'antigène CD147, s'il est confirmé qu'il est l'autre point[120] d'entrée du virus, pourraient présenter des effets indésirables moindres.
Anticorps monoclonaux et transfert adoptif de cellules
Le développement d'anticorps monoclonaux spécifiques est une autre piste[271],[272] de traitement qui a par ailleurs déjà montré son efficacité dans le cas du MERS-CoV[273],[274]. Il doit être possible, en effet, de trouver des anticorps, par la technique phage display, ou résultant de la simulation informatique[275],[276], neutralisant[277],[278] ou freinant l'action du virus. Ceci, d'autant que les anticorps produits naturellement n’empêcheraient pas les infections récidivantes chez certains patients[279],[280]. Plusieurs études sont en cours pour le tocilizumab, le sarilumab[281], et le léronlimab[282]. Un traitement conçu à partir du plasma des patients immunisés diminuerait la mortalité par deux[283]. Il est envisageable[284] par biomimétisme d'analyser le plasma des patients immunisés, en complément des anticorps déjà détectés pour la mise au point des tests sérologiques, pour la production[285] d'un cocktail thérapeutique d'anticorps[286] à administrer.
Il doit être possible comme cela a été tenté pour le virus HIV[287],[288],[289], de produire des lymphocytes B modifiés par CRISPr-Cas9 contre le virus SARS-CoV-2, dans des thérapies de type Transfert adoptif de cellules. Cette approche permet en particulier de s'affranchir du phénomène d'immunosénescence, phénomène justifiant que les vaccins contre la grippe soient moins efficaces chez les plus de 65 ans, que dans le reste de la population, tout en étant plus utiles[290]. Le recours aux thérapies basées sur l'utilisation de cellules T CAR est freiné par leurs coûts élevés, en particulier du fait des brevets déposés par les laboratoires pharmaceutiques[291]. Un faible niveau de lymphocytes T, qui pourrait aussi résulter de l'action du virus[292], est ainsi un facteur prédictif d'une mauvaise réponse à l'infection[293].
Azithromycine & zinc
Trois médecins (Jean-Jacques Erbstein, Denis Gastaldi et Olivia Van Steen Berghe) proposent un traitement basé sur l'Azithromycine, un antibiotique de la famille des macrolides, donc l'action est renforcé par un apport de zinc pour prévenir les formes graves de la maladie[294]. Ce traitement a été développé, par ces trois médecins, sur la base de leurs connaissances et d'observations empiriques. Ce type de traitement est important pour soigner les malades en attendant que le résultats des études complètes (Étude_randomisée_en_double_aveugle) qui demandent beaucoup de temps et s'inscit dans l'éthique de la médecine[295]. Ce type de traitements utilisent des médicaments éprouvés.
Notes et références
- (en) « Naming the 2019 Coronavirus », ICTV, (consulté le ).
- (en) « Virus Taxonomy: 2018b Release », ICTV, (consulté le ).
- (en) « Phylogeny of SARS-like betacoronaviruses including novel coronavirus from Wuhan using data generated by the Shanghai Public Health Clinical Center & School of Public Health, the National Institute for Viral Disease Control and Prevention, the Institute of Pathogen Biology, and the Wuhan Institute of Virology shared via GISAID », sur https://nextstrain.org/, (consulté le ).
- (en) Fisher D. et Heymann D., « Q&A: The novel coronavirus outbreak causing COVID-19 », BMC Med, (lire en ligne).
- (en) Zhengde Xie, « Pay attention to SARS-CoV-2 infection in children », PEDIATRIC INVESTIGATION, vol. 4, no 1, , p. 1–4 (ISSN 2574-2272, DOI 10.1002/ped4.12178, lire en ligne, consulté le ).
- (en) David Cyranoski, « Did pangolins spread the China coronavirus to people? », Nature, , d41586–020–00364-2 (ISSN 0028-0836 et 1476-4687, DOI 10.1038/d41586-020-00364-2, lire en ligne, consulté le ).
- « SRAS-CoV-2 », Grand Dictionnaire terminologique, Office québécois de la langue française (consulté le ).
- « Appellation de la maladie à coronavirus 2019 (COVID-19) et du virus qui la cause », sur www.who.int, Organisation mondiale de la santé, (consulté le ).
- « WHO | WHO best practices for naming of new human infectious diseases », sur WHO (consulté le ).
- (en) Alexander E. Gorbalenya, Susan C. Baker, Ralph S. Baric, Raoul J. de Groot, Christian Drosten, Anastasia A. Gulyaeva, Bart L. Haagmans, Chris Lauber, Andrey M. Leontovich, Benjamin W. Neuman, Dmitry Penzar, Stanley Perlman, Leo L.M. Poon, Dmitry Samborskiy, Igor A. Sidorov, Isabel Sola et John Ziebuhr, « Severe acute respiratory syndrome-related coronavirus – The species and its viruses, a statement of the Coronavirus Study Group », bioRxiv, (DOI 10.1101/2020.02.07.937862, lire en ligne, consulté le ).
- (en) « Novel Coronavirus (2019-nCoV) Situation Report – 22 » [PDF], sur who.int, Organisation mondiale de la santé, (consulté le ) : « Following WHO best practices for naming of new human infectious diseases, which were developed in consultation and collaboration with the World Organisation for Animal Health (OIE) and the Food and Agriculture Organization of the United Nations (FAO), WHO has named the disease COVID-19, short for “coronavirus disease 2019.” », p. 1.
- « COVID-19 », Grand Dictionnaire terminologique, Office québécois de la langue française (consulté le ).
- (en-US) « Wuhan Coronavirus (2019-nCoV) global cases (by JHU CSSE) », sur Johns Hopkins University Center for Systems Science and Engineering (consulté le ).
- « Épidémie de Covid-19 : la difficulté de bien nommer le virus et la maladie », Le Monde.fr, (lire en ligne, consulté le ).
- (en) Shibo Jiang, Zhengli Shi, Yuelong Shu et Jingdong Song, « A distinct name is needed for the new coronavirus », The Lancet, , S0140673620304190 (DOI 10.1016/S0140-6736(20)30419-0, lire en ligne, consulté le ).
- (en) Yuntao Wu, Wenzhe Ho, Yaowei Huang et Dong-Yan Jin, « SARS-CoV-2 is an appropriate name for the new coronavirus », The Lancet, , S0140673620305572 (DOI 10.1016/S0140-6736(20)30557-2, lire en ligne, consulté le ).
- Sébastien Rouet, « Covid-19, Ebola, peste noire… nos sociétés face aux maladies, avec l’historien Frédéric Vagneron », sur geo.fr, (consulté le ).
- (en) « COVID-19 SARS-CoV-2 preprints from medRxiv and bioRxiv », sur bioRxiv (consulté le ).
- (en) Tianmu Chen, Jia Rui, Qiupeng Wang et Zeyu Zhao, « A mathematical model for simulating the transmission of Wuhan novel Coronavirus », bioRxiv, Systems Biology, (DOI 10.1101/2020.01.19.911669, lire en ligne, consulté le ).
- (en) Jonathan M Read, Jessica RE Bridgen, Derek AT Cummings et Antonia Ho, « Novel coronavirus 2019-nCoV: early estimation of epidemiological parameters and epidemic predictions », medRxiv, Infectious Diseases (except HIV/AIDS), (DOI 10.1101/2020.01.23.20018549, lire en ligne, consulté le ).
- (en) Jasper Fuk-Woo Chan, Shuofeng Yuan, Kin-Hang Kok et Kelvin Kai-Wang To, « A familial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission: a study of a family cluster », The Lancet, , S0140673620301549 (DOI 10.1016/S0140-6736(20)30154-9, lire en ligne, consulté le ).
- (en) Chaolin Huang, Yeming Wang, Xingwang Li et Lili Ren, « Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China », The Lancet, , S0140673620301835 (DOI 10.1016/S0140-6736(20)30183-5, lire en ligne, consulté le ).
- (en) « Novel Coronavirus Information Center », sur Elsevier Connect (consulté le ).
- (en) « COVID-19 Resource Centre », sur thelancet.com (consulté le ).
- (en) « Coronavirus (Covid-19) — NEJM », sur New England Journal of Medicine (consulté le ).
- (en) « Coronavirus », sur Science | AAAS (consulté le ).
- « Coronavirus | Springer Nature | For Researchers | Springer Nature », sur springernature.com (consulté le ).
- (en) « Novel Coronavirus - Wiley Online Library », sur Novel Coronavirus (consulté le ).
- (en) Marie E. Killerby, Holly M. Biggs, Amber Haynes et Rebecca M. Dahl, « Human coronavirus circulation in the United States 2014–2017 », Journal of Clinical Virology, vol. 101, , p. 52–56 (DOI 10.1016/j.jcv.2018.01.019, lire en ligne, consulté le ).
- (en) Hui DS, I Azhar E, Madani TA, Ntoumi F, Kock R,, Dar O, Ippolito G, Mchugh TD, Mchugh TD, Memish ZA et al., « The continuing 2019-nCoV epidemic threat of novel coronaviruses to global health – The latest 2019 novel coronavirus outbreak in Wuhan, China », International Journal of Infectious Diseases, vol. 91, , p. 264-266 (PMID 31953166, DOI 10.1016/j.ijid.2020.01.009, lire en ligne, consulté le )
 .
.
- (en) Na Zhu, Dingyu Zhang, Wenling Wang et Xingwang Li, « A Novel Coronavirus from Patients with Pneumonia in China, 2019 », New England Journal of Medicine, , NEJMoa2001017 (ISSN 0028-4793 et 1533-4406, DOI 10.1056/NEJMoa2001017, lire en ligne, consulté le ).
- (en-CA) « Wuhan Novel Coronavirus (2019-nCoV) », sur Ontario.ca, (consulté le ).
- « Experts explain the latest bulletin of unknown cause of viral pneumonia » [archive du ], sur Wuhan Municipal Health Commission, (consulté le ).
- « Coronavirus : ce que l'on sait des premiers patients touchés par l'épidémie », sur Franceinfo, (consulté le ).
- « Déclaration du Directeur général de l’OMS sur les recommandations du Comité d’urgence du RSI sur le nouveau coronavirus », sur www.who.int, (consulté le ).
- (en) Anna Fifield, « As families tell of pneumonia-like deaths in Wuhan, some wonder if China virus count is too low », The Washington Post, (lire en ligne).
- (en-GB) Lily Kuo et Lillian Yang, « Coronavirus: Chinese hospitals not testing patients, say relatives », The Guardian, (ISSN 0261-3077, lire en ligne, consulté le ).
- (en-US) Wenxin Fan, « Relatives Wonder Why Pneumonia Deaths Not in Coronavirus Tally », The Wall Street Journal, (ISSN 0099-9660, lire en ligne, consulté le ).
- The Novel Coronavirus Pneumonia Emergency Response Epidemiology Team. The Epidemiological Characteristics of an Outbreak of 2019 Novel Coronavirus Diseases (COVID-19) — China, 2020. China CDC Weekly, 2020, 2(8): 113–122 (résumé).
- (en) « Novel Coronavirus (2019-nCoV) situation reports », sur www.who.int (consulté le ).
- « Coronavirus (SRAS-CoV et MERS-CoV) », sur Inserm - La science pour la santé (consulté le ).
- Wang Y, Wang Y, Chen Y & Qin Q (2020) Unique epidemiological and clinical features of the emerging 2019 novel coronavirus pneumonia (COVID-19) implicate special control measures ; J Med Virol. 5 mars 2020 ; Epub 2020 Mar 5 (résumé)
- (it) « Covid-19 Bollettino 11/03/2020 » [PDF], sur Ministère de la Santé (Italie) (consulté le ).
- (kr) « 코로나바이러스감염증-19(COVID-19) » (consulté le ).
- « WHO Director-General's opening remarks at the media briefing on COVID-19 - 3 March 2020 », sur who.int, Organisation mondiale de la santé, .
- (en) Mike Famulare, « 2019-nCoV: preliminary estimates of the confirmed-case-fatality-ratio and infection-fatality-ratio, and initial pandemic risk assessment », Github, Institute for disease modeling, (lire en ligne, consulté le ).
- Toutefois le SARS-CoV-2 a aussi été décrit comme mesurant approximativement de 60 nm à 140 nm, et comme étant de forme elliptique avec de nombreuses variations ; voir : Cascella M, Rajnik M, Cuomo A, et al. Features, Evaluation and Treatment Coronavirus (COVID-19), in: StatPearls [Internet], StatPearls Publishing, Treasure Island (Floride), janvier 2020, mis à jour le 8 mars 2020 lire en ligne
- (en) Trevor Bedford and Richard Neher, « Genomic epidemiology of novel coronavirus (nCoV) using data generated by Fudan University, China CDC, Chinese Academy of Medical Sciences, Chinese Academy of Sciences, Zhejiang Provincial Center for Disease Control and Prevention and the Thai National Institute of Health shared via GISAID », nextstrain.org (consulté le ).
- (en) « Initial genome release of novel coronavirus », Virological, (consulté le ).
- (en) Wu, F. et al., « Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome », sur GenBank, (consulté le ).
- (en) Shi, ZL et al., « Discovery of a novel coronavirus associated with the recent pneumonia outbreak in humans and its potential bat origin », bioRxiv, (lire en ligne).
- (en) Benvenuto D. et al., « The 2019-new Coronavirus epidemic: evidence for virus evolution », bioRxiv.org, (lire en ligne).
- (en) « Phylogeny of SARS-like betacoronaviruses », nextstrain (consulté le ).
- (en) Hui DS, I Azhar E, Madani TA, Ntoumi F, Kock R, Dar O, Ippolito G, Mchugh TD, Memish ZA, Drosten C, Zumla A, Petersen E. The continuing 2019-nCoV epidemic threat of novel coronaviruses to global health – The latest 2019 novel coronavirus outbreak in Wuhan, China. Int J Infect Dis. 2020 Jan 14;91:264–266. DOI 10.1016/j.ijid.2020.01.009
 .
.
- (en) Antonio C. P. Wong, Xin Li, Susanna K. P. Lau, Patrick C. Y. Woo: Global Epidemiology of Bat Coronaviruses, in: Viruses. 2019 Feb; 11(2): 174, doi:10.3390/v11020174.
- (zh) « Notice d'information de la Commission municipale de la santé de Wuhan sur la situation actuelle de l'épidémie de pneumonie dans notre ville [en chinois] », sur http://wjw.wuhan.gov.cn/, (consulté le ).
- (en-US) Antigone Barton, « UPDATE Wuhan coronavirus – 2019-nCoV Q&A #6: An evidence-based hypothesis », sur Science Speaks: Global ID News, (consulté le ).
- (en) Jon CohenJan. 26[Qui ?] et 2020[Qui ?], « Wuhan seafood market may not be source of novel virus spreading globally », sur Science | AAAS, (consulté le ).
- Dorian Malovic et Antoine d’Abbundo, « Coronavirus : la Chine interdit le commerce d’animaux sauvages », sur la-croix.com, (consulté le ).
- (en) Ji, Wang, Zhao et Zai, « Homologous recombination within the spike glycoprotein of the newly identified coronavirus may boost cross‐species transmission from snake to human », Journal of Medical Virology, (DOI 10.1002/jmv.25682).
- (en) Peng Zhou, Xing-Lou Yang, Xian-Guang Wang et Ben Hu, « A pneumonia outbreak associated with a new coronavirus of probable bat origin », Nature, (ISSN 0028-0836 et 1476-4687, DOI 10.1038/s41586-020-2012-7, lire en ligne, consulté le ).
- (en) Xingguang Li, Junjie Zai, Qiang Zhao et Qing Nie, « Evolutionary history, potential intermediate animal host, and cross‐species analyses of SARS‐CoV‐2 », Journal of Medical Virology, , jmv.25731 (ISSN 0146-6615 et 1096-9071, DOI 10.1002/jmv.25731, lire en ligne, consulté le ).
- (en) « Wuhan virus: a visual explainer », sur South China Morning Post (consulté le ).
- « #VraiOuFake Coronavirus : la consommation d'une soupe de chauve-souris à Wuhan est-elle à l'origine de l'épidémie ? », sur Franceinfo, (consulté le ).
- (en-US) James Palmer, « Don’t Blame Bat Soup for the Wuhan Virus », sur Foreign Policy (consulté le ).
- (en) Conversation[Qui ?], « Snakes could be the source of the Wuhan coronavirus outbreak », sur CNN (consulté le ).
- (en-US) Robertson D.L., « nCoV's relationship to bat coronaviruses & recombination signals (no snakes) », sur Virological, (consulté le ).
- (en) Callaway E. et Cyranoski D., « Why snakes probably aren’t spreading the new China virus », Nature, (ISSN 0028-0836 et 1476-4687, DOI 10.1038/d41586-020-00180-8, lire en ligne, consulté le ).
- « Coronavirus : le pangolin a-t-il pu servir d’hôte intermédiaire ? », Le Monde.fr, (lire en ligne, consulté le ).
- (en) Ping Liu, Wu Chen et Jin-Ping Chen, « Viral Metagenomics Revealed Sendai Virus and Coronavirus Infection of Malayan Pangolins (Manis javanica) », Viruses, vol. 11, no 11, , p. 979 (ISSN 1999-4915, PMID 31652964, PMCID PMC6893680, DOI 10.3390/v11110979, lire en ligne, consulté le ).
- (en) Matthew C. Wong, Sara J. Javornik Cregeen, Nadim J. Ajami et Joseph F. Petrosino, « Evidence of recombination in coronaviruses implicating pangolin origins of nCoV-2019 », bioRxiv, Microbiology, (DOI 10.1101/2020.02.07.939207, lire en ligne, consulté le ).
- (en) Alexandre Hassanin, « Covid-19 : l’analyse des génomes révèlerait une origine double du virus », sur The Conversation (consulté le ).
- (en) « GISAID - Next betaCoV App », sur www.gisaid.org (consulté le ).
- « Coronavirus chinois : les raisons pour lesquelles il ne faut pas paniquer », sur LExpress.fr, (consulté le ).
- (en) « Wuhan seafood market pneumonia virus », sur NCBI (consulté le ).
- (en) « New-type coronavirus causes pneumonia in Wuhan: expert – Xinhua | English.news.cn » (consulté le ).
- (en) « CoV2020 », platform.gisaid.org (consulté le ).
- (en) SARS-CoV, B. S. (2020). Mutation and Recombination[PDF].
- (en) Lu R, Zhao X, Li J, et al. (2020) Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding. The Lancet; 395: 565–74.
- (en) Lee E.Y, Ng M.Y & Khong P.L (2020) COVID-19 pneumonia: what has CT taught us?[PDF]. The Lancet Infectious Diseases.
- Hervé Morin, Coronavirus : le pangolin a-t-il pu servir d’hôte intermédiaire ?, Le Monde, . L’université d’agriculture de Chine du Sud a annoncé qu'un séquencage du virus trouvés chez des pangolins montrait qu'il était homologue à 99 % à celui du SARS-CoV-2. Le pangolin aurait pu servir d'espèce réservoir ou hôte entre la chauve-souris et l'homme, une hypothèse discutée par plusieurs spécialistes.
- Expert reaction to statement from South China Agricultural University that research has identified the pangolin as a possible coronavirus host, Science Media Center, 7/02/2020.
- nCoV-2019 - Spike Protein Receptor Binding Domain Shares High Amino Acid Identity With a Coronavirus Recovered from a Pangolin Viral Metagenomic Dataset, Novel 2019 coronavirus, nCoV-2019 Evolutionary History, Virological.org, 30 janvier 2020.
- (en) Kangpeng Xiao, Junqiong Zhai, Yaoyu Feng et Niu Zhou, « Isolation and Characterization of 2019-nCoV-like Coronavirus from Malayan Pangolins », bioRxiv, Microbiology, (DOI 10.1101/2020.02.17.951335, lire en ligne, consulté le ).
- Chloé Hecketsweiler, « Coronavirus : le SARS-CoV-2 est-il sorti d’un laboratoire ? », sur lemonde.fr, (consulté le ).
- Perrine Mouterde, « Coronavirus : la dégradation de la biodiversité en question », sur lemonde.fr, (consulté le ).
- Anaïs Culot, « La science fondamentale est notre meilleure assurance contre les épidémies », sur lejournal.cnrs.fr, (consulté le ).
- Philippe Sansonetti, « Covid-19, chronique d’une émergence annoncée », sur laviedesidees.fr, (consulté le ).
- Jade Lindgaard et Amélie Poinssot, « Le coronavirus, «un boomerang qui nous revient dans la figure» », sur Mediapart, (consulté le ).
- « La crise de la biodiversité favorise les maladies infectieuses émergentes », sur Charlie Hebdo.fr, (consulté le ).
- Fiorenza Gracci, « "Voici comment le coronavirus a envahi l'espèce humaine" », sur science-et-vie.com, (consulté le ).
- « Didier Sicard : "Il est urgent d'enquêter sur l'origine animale de l'épidémie de Covid-19" », sur France Culture.fr, (consulté le ).
- Marie-Adélaïde Scigacz, « "Cette crise doit nous engager à agir" : la pandémie de coronavirus va-t-elle pousser l'espèce humaine à (enfin) respecter la biodiversité ? », sur francetvinfo.fr, (consulté le ).
- Alexandre Hassanin, « Covid-19 : origine naturelle ou anthropique ? », sur The Conversation.com, (consulté le ).
- cnn.com 6 avril 2020
- L'Obs avec AFP, « L’administration Trump dit enquêter pour savoir si le coronavirus provient d’un laboratoire de Wuhan », sur nouvelobs.com, (consulté le ).
- sudinfo.be
- William Audureau, « Le coronavirus, fabriqué à partir du virus du sida ? La thèse très contestée du professeur Montagnier », sur lemonde.fr, (consulté le ).
- Le point, « En liant coronavirus et VIH, le professeur Montagnier fait polémique », sur lepoint.fr, (consulté le ).
- (en) Kristian G. Andersen, Andrew Rambaut, W. Ian Lipkin, Edward C. Holmes et Robert F. Garry, « The proximal origin of SARS-CoV-2 », nature medecine, no 26, , p. 450-452 (lire en ligne, consulté le ) :
.« Although the evidence shows that SARS-CoV-2 is not a purposefully manipulated virus, it is currently impossible to prove or disprove the other theories of its origin described here. However, since we observed all notable SARS-CoV-2 features, […] in related coronaviruses in nature, we do not believe that any type of laboratory-based scenario is plausible proximal origin. »
- (en) Nanshan Chen, Min Zhou, Xuan Dong et Jieming Qu, « Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: a descriptive study », The Lancet, vol. 395, no 10223, , p. 507–513 (DOI 10.1016/S0140-6736(20)30211-7, lire en ligne, consulté le )
- (en) Canrong Wu, Yang Liu, Yueying Yang et Peng Zhang, « Analysis of therapeutic targets for SARS-CoV-2 and discovery of potential drugs by computational methods », Acta Pharmaceutica Sinica B, , S2211383520302999 (DOI 10.1016/j.apsb.2020.02.008, lire en ligne, consulté le )
- Astrid Vabret et Fabien Miszczak, « Changement de tropisme des coronavirus », Revue Francophone des Laboratoires, tropisme des virus : de l'animal à l'homme ?, vol. 2010, no 423, , p. 63–68 (ISSN 1773-035X, DOI 10.1016/S1773-035X(10)70561-X, lire en ligne, consulté le )
- (en) Daniel Wrapp, Nianshuang Wang, Kizzmekia S. Corbett et Jory A. Goldsmith, « Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation », Science, vol. 367, no 6483, , p. 1260–1263 (ISSN 0036-8075 et 1095-9203, DOI 10.1126/science.abb2507, lire en ligne, consulté le )
- Mandelbaum RF (19 février 2020). "[Scientists Create Atomic-Level Image of the New Coronavirus's Potential Achilles Heel]". Gizmodo. consulté le 13 mars 2020
- (en) Smriti Mallapaty, « Why does the coronavirus spread so easily between people? », Nature, vol. 579, no 7798, , p. 183 (DOI 10.1038/d41586-020-00660-x).
- (en) Sandeep Chakraborty, « Ubiquitous genomic fragment in human 2019-nCoV viruses in the spike-protein, also encoding a novel 87 aa protein, completely missing in all other coronaviruses », OSF preprints, Open Science Framework, (DOI 10.31219/osf.io/6vd54, lire en ligne, consulté le ).
- (en) Xintian Xu, Ping Chen, Jingfang Wang et Jiannan Feng, « Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission », Science China Life Sciences, vol. 63, no 3, , p. 457–460 (ISSN 1674-7305 et 1869-1889, PMID 32009228, PMCID PMC7089049, DOI 10.1007/s11427-020-1637-5, lire en ligne, consulté le )
- (en) Michael Letko et Vincent Munster, « Functional assessment of cell entry and receptor usage for lineage B β-coronaviruses, including 2019-nCoV », Nature Microbiology, (DOI 10.1101/2020.01.22.915660., lire en ligne, consulté le )
- Michael Letko, Andrea Marzi et Vincent Munster, « Functional assessment of cell entry and receptor usage for SARS-CoV-2 and other lineage B betacoronaviruses », Nature Microbiology, vol. 5, no 4, , p. 562–569 (ISSN 2058-5276, PMID 32094589, DOI 10.1038/s41564-020-0688-y, lire en ligne, consulté le )
- El Sahly HM (2020) "Genomic Characterization of the 2019 Novel Coronavirus". The New England Journal of Medicine. consulté le 9 février 2020.
- (en) Lisa E. Gralinski et Vineet D. Menachery, « Return of the Coronavirus: 2019-nCoV », Viruses, vol. 12, no 2, , p. 135 (ISSN 1999-4915, DOI 10.3390/v12020135, lire en ligne, consulté le )
- Roujian Lu, Xiang Zhao, Juan Li et Peihua Niu, « Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding », The Lancet, vol. 395, no 10224, , p. 565–574 (ISSN 0140-6736, DOI 10.1016/s0140-6736(20)30251-8, lire en ligne, consulté le )
- (en) Peng Zhou, Xing-Lou Yang, Xian-Guang Wang et Ben Hu, « A pneumonia outbreak associated with a new coronavirus of probable bat origin », Nature, vol. 579, no 7798, , p. 270–273 (ISSN 0028-0836 et 1476-4687, DOI 10.1038/s41586-020-2012-7, lire en ligne, consulté le )
- (en) Xu, X. et al., « Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission », Science China - Life Sciences, (lire en ligne).
- (en) Michael C. Letko et Vincent Munster, « Functional assessment of cell entry and receptor usage for lineage B β-coronaviruses, including 2019-nCoV », bioRxiv, (DOI 10.1101/2020.01.22.915660, lire en ligne, consulté le ).
- (en) Markus Hoffmann, Hannah Kleine-Weber, Nadine Krueger et Marcel A Mueller, « The novel coronavirus 2019 (2019-nCoV) uses the SARS-coronavirus receptor ACE2 and the cellular protease TMPRSS2 for entry into target cells », bioRxiv, Molecular Biology, (DOI 10.1101/2020.01.31.929042, lire en ligne, consulté le ).
- (en) Markus Hoffmann, Hannah Kleine-Weber, Simon Schroeder et Nadine Krüger, « SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a Clinically Proven Protease Inhibitor », Cell, , S0092867420302294 (PMID 32142651, PMCID PMC7102627, DOI 10.1016/j.cell.2020.02.052, lire en ligne, consulté le )
- (en) B. Coutard, C. Valle, X. de Lamballerie et B. Canard, « The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade », Antiviral Research, vol. 176, , p. 104742 (DOI 10.1016/j.antiviral.2020.104742, lire en ligne, consulté le )
- (en) Ke Wang, Wei Chen, Yu-Sen Zhou et Jian-Qi Lian, « SARS-CoV-2 invades host cells via a novel route: CD147-spike protein », bioRxiv, Microbiology, (DOI 10.1101/2020.03.14.988345, lire en ligne, consulté le )
- W. Kasinrerk, E. Fiebiger, I. Stefanová et T. Baumruker, « Human leukocyte activation antigen M6, a member of the Ig superfamily, is the species homologue of rat OX-47, mouse basigin, and chicken HT7 molecule », Journal of Immunology (Baltimore, Md.: 1950), vol. 149, no 3, , p. 847–854 (ISSN 0022-1767, PMID 1634773, lire en ligne, consulté le )
- Vyacheslav Yurchenko, Stephanie Constant et Michael Bukrinsky, « Dealing with the family: CD147 interactions with cyclophilins », Immunology, vol. 117, no 3, , p. 301–309 (ISSN 0019-2805, PMID 16476049, PMCID 1782239, DOI 10.1111/j.1365-2567.2005.02316.x, lire en ligne, consulté le )
- (en) Teruo Miyauchi, Yasushi Masuzawa et Takashi Muramatsu, « The Basigin Group of the Immunoglobulin Superfamily: Complete Conservation of a Segment in and around Transmembrane Domains of Human and Mouse Basigin and Chicken HT7 Antigen », The Journal of Biochemistry, vol. 110, no 5, , p. 770–774 (ISSN 1756-2651 et 0021-924X, DOI 10.1093/oxfordjournals.jbchem.a123657, lire en ligne, consulté le )
- HuangQ,LiJ,XingJ,LiW,LiH,KeX,ZhangJ,RenT,ShangY,YangH,JiangJ,ChenZ. CD147 promotes reprogramming of glucose metabolism and cell proliferation in HCC cells by inhibiting the p53-dependent signaling pathway. J HEPATOL. 2014; 61: 859-866.
- (en) Pu Zhao, Wei Zhang, Shi-Jie Wang et Xiao-Ling Yu, « HAb18G/CD147 promotes cell motility by regulating annexin II-activated RhoA and Rac1 signaling pathways in hepatocellular carcinoma cells », Hepatology, vol. 54, no 6, , p. 2012–2024 (ISSN 1527-3350, PMID 21809360, DOI 10.1002/hep.24592, lire en ligne, consulté le )
- Lu M, Wu J, Hao ZW, Shang YK, Xu J, Nan G, Li X, Chen ZN, Bian H. Basolateral CD147 induces hepatocyte polarity loss by E-cadherin ubiquitination and degradation in hepatocellular carcinoma progress. HEPATOLOGY. 2018; 68: 317-332.
- Pushkarsky T, Zybarth G, Dubrovsky L, Yurchenko V, Tang H, Guo H, Toole B, Sherry B, Bukrinsky M. CD147 facilitates HIV-1 infection by interacting with virus-associated cyclophilin A. Proc Natl Acad Sci USA. 2001; 98: 6360-6365.
- Cécile Crosnier, Leyla Y. Bustamante, S. Josefin Bartholdson et Amy K. Bei, « Basigin is a receptor essential for erythrocyte invasion by Plasmodium falciparum », Nature, vol. 480, no 7378, , p. 534–537 (ISSN 1476-4687, PMID 22080952, PMCID 3245779, DOI 10.1038/nature10606, lire en ligne, consulté le )
- Zhang MY, Zhang Y, Wu XD, Zhang K, Lin P, Bian HJ, Qin MM, Huang W, Wei D, Zhang Z,Wu J, Chen R, Feng F, et al. Disrupting CD147-RAP2 interaction abrogates erythrocyte invasion by Plasmodium falciparum. BLOOD. 2018; 131: 1111-1121.
- (en) Vyacheslav Yurchenko, Stephanie Constant et Michael Bukrinsky, « Dealing with the family: CD147 interactions with cyclophilins », Immunology, vol. 117, no 3, , p. 301–309 (ISSN 0019-2805 et 1365-2567, PMID 16476049, PMCID PMC1782239, DOI 10.1111/j.1365-2567.2005.02316.x, lire en ligne, consulté le )
- ChenZ,MiL,XuJ,YuJ,WangX,JiangJ,XingJ,ShangP,QianA,LiY,ShawPX,WangJ, Duan S, et al. (2005) Function of HAb18G/CD147 in invasion of host cells by severe acute respiratory syndrome coronavirus. J INFECT DIS.; 191: 755-760.
- (en) Huijie Bian, Zhao-Hui Zheng, Ding Wei et Zheng Zhang, « Meplazumab treats COVID-19 pneumonia: an open-labelled, concurrent controlled add-on clinical trial », {{Article}} : paramètre «
périodique» manquant, Infectious Diseases (except HIV/AIDS), (DOI 10.1101/2020.03.21.20040691., lire en ligne, consulté le ) - Christopher M. Coleman, Krystal L. Matthews, Lindsay Goicochea et Matthew B. Frieman, « Wild-type and innate immune-deficient mice are not susceptible to the Middle East respiratory syndrome coronavirus », The Journal of General Virology, vol. 95, no Pt 2, , p. 408–412 (ISSN 0022-1317, PMID 24197535, PMCID 3917065, DOI 10.1099/vir.0.060640-0, lire en ligne, consulté le ).
- Li YC, Bai WZ, Hashikawa T. (2020) The neuroinvasive potential of SARS-CoV2 may be at least partially responsible for the respiratory failure of COVID-19 patients.J Med Virol. 2020 Feb 27; Epub 2020 Feb 27 (résumé)
- (en) Knvul Sheikh et Roni Caryn Rabin, « The Coronavirus: What Scientists Have Learned So Far », The New York Times, .
- (en) « Clinical characteristics of 2019 novel coronavirus infection in China », medRxiv, .
- (en) Stephen A. Lauer, Kyra H. Grantz, Qifang Bi et Forrest K. Jones, « The Incubation Period of Coronavirus Disease 2019 (COVID-19) From Publicly Reported Confirmed Cases: Estimation and Application », Annals of Internal Medicine, (ISSN 0003-4819, DOI 10.7326/M20-0504, lire en ligne, consulté le ).
- (en) Tapiwa Ganyani, Cecile Kremer, Dongxuan Chen et Andrea Torneri, « Estimating the generation interval for COVID-19 based on symptom onset data », bioRxiv, Infectious Diseases (except HIV/AIDS), (DOI 10.1101/2020.03.05.20031815, lire en ligne).
- (en) N. van Doremalen, T. Bushmaker, D. Morris, M. Holbrook, A. Gamble, B. Williamson, et J. Lloyd-Smith, « Aerosol and surface stability of HCoV-19 (SARS-CoV-2) compared to SARS-CoV-1 », medRxiv, 2020, [lire en ligne].
- (en) « VERIFY: Can you get Coronavirus from packages shipped from China? », wbir.com (consulté le ).
- « #VraiOuFake Un colis en provenance de Chine peut-il transmettre le coronavirus ? », France Info, (consulté le ).
- (en) G. Kampf, D. Todt, S. Pfaender, et E. Steinmann, « Persistence of coronaviruses on inanimate surfaces and its inactivation with biocidal agents », Journal of Hospital Infection, vol. 104, no 3, mars 2020, pp. 246-251, [lire en ligne].
- Robert F. Service (2020) Does disinfecting surfaces really prevent the spread of coronavirus? ; 12 mars 2020 Science News/HealthCoronavirus | doi:10.1126/science.abb7058
- (en) « China coronavirus: Hong Kong scraps major Lunar New Year events », sur South China Morning Post, (consulté le ).
- (en) hermesauto, « Wuhan virus: Vietnam confirms 2 cases of Sars-like coronavirus », The Straits Times, (consulté le ).
- (en) hermesauto, « Singapore confirms first case of Wuhan virus », The Straits Times, (consulté le ).
- « New Virus Spreads to U.S., Sparking Rush to Contain Outbreak », sur www.bloomberg.com, (consulté le ).
- (en) Scott Morrison, « Update on Coronavirus pic.twitter.com/Ol8DRQtVQJ », sur @ScottMorrisonMP, (consulté le ).
- « Coronavirus : un troisième cas d’infection confirmé en France », Le Monde.fr, (lire en ligne, consulté le ).
- « Coronavirus : 5 nouveaux cas "sans gravité" en France dont un enfant de 9 ans scolarisé dans 2 écoles », sur lci.fr, (consulté le ).
- APANEWS, « Coronavirus : l’Egypte enregistre le premier cas en Afrique », sur apanews.net (consulté le ).
- (en) « Brazil confirms first coronavirus case in Latin America: source », Reuters, (lire en ligne, consulté le ).
- Cereda D, Tirani M, Rovida F et Demicheli V, « The early phase of the COVID-19 outbreak in Lombardy, Italy », arXiv:2003.09320 [q-bio], (lire en ligne, consulté le ).
- huet, « Le COVID-19 en Italie dès le 1er janvier 2020 », sur {Sciences²}, (consulté le ).
- (en-US) Jin Wu, Weiyi Cai, Derek Watkins et James Glanz, « How the Virus Got Out », The New York Times, (ISSN 0362-4331, lire en ligne, consulté le ).
- (en) « Two thirds of COVID-19 cases exported from mainland China may be undetected | Imperial News | Imperial College London », sur Imperial News (consulté le ).
- « L’épidémie de coronavirus très sous-estimée dans le monde », La Croix, (ISSN 0242-6056, lire en ligne, consulté le ).
- (en) Associated Press, « China confirms human-to-human transmission of new coronavirus », sur CBC.ca, (consulté le ).
- (en) « Statement on the meeting of the International Health Regulations (2005) Emergency Committee regarding the outbreak of novel coronavirus 2019 (n-CoV) on 23 January 2020 », sur www.who.int (consulté le ).
- (en) Julien Riou et Christian L. Althaus, « Pattern of early human-to-human transmission of Wuhan 2019-nCoV », bioRxiv, , p. 2020.01.23.917351 (DOI 10.1101/2020.01.23.917351, lire en ligne, consulté le ).
- « Déclaration de l’OMS sur le nouveau coronavirus en Thaïlande », sur www.who.int (consulté le ).
- « Déclaration sur la deuxième réunion du Comité d’urgence du Règlement sanitaire international (2005) concernant la flambée de nouveau coronavirus 2019 (2019-nCoV) tenue le 30 janvier 2020 », sur www.who.int (consulté le ).
- (en) Princess Cruises, « Princess Cruises: Diamond Princess Coronavirus & Quarantine Updates - Notices & Advisories », sur www.princess.com, mon feb 17 15:21:24 pst 2020 (consulté le ).
- « Coronavirus: l'Italie, la Corée du Sud et l'Iran tentent d'endiguer l'épidémie », sur RFI, (consulté le ).
- « Coronavirus : la contamination s’accélère à travers la planète », Le Monde.fr, (lire en ligne, consulté le ).
- « Allocution liminaire du Directeur général de l’OMS lors du point presse sur la COVID-19 - 11 mars 2020 », sur www.who.int (consulté le ).
- « Coronavirus : les Philippines enregistrent le premier décès hors de Chine », sur France 24, (consulté le ).
- « Coronavirus : un premier décès sur le territoire français », sur Franceinfo, (consulté le ).
- « Les États-Unis ont le plus grand nombre de cas de Covid-19 recensés dans le monde », sur LExpress.fr, (consulté le ).
- « Coronavirus : des passagers du paquebot en quarantaine ont échappé aux contrôles », sur Europe 1 (consulté le ).
- Anna Golda, Natalia Malek, Bartosz Dudek, Slawomir Zeglen, Jacek Wojarski, Marek Ochman, Ewa Kucewicz, Marian Zembala, Jan Potempa et Krzysztof Pyrc, « Infection with human coronavirus NL63 enhances streptococcal adherence to epithelial cells », The Journal of General Virology, no Pt 6, , p. 1358–1368 (PMID 21325482, PMCID 3168281, DOI 10.1099/vir.0.028381-0, lire en ligne, consulté le ).
- « Intérêt d’un traitement précoce par antiviral pour réduire la sévérité et la mortalité par grippe A(H1N1)2009 : données issues de la surveillance des formes graves », SantePubliquefrance, (lire en ligne).
- « Sida et VIH, À quand la guérison ? », Site inserm, (lire en ligne)
- Lu H. Drug treatment options for the 2019-new coronavirus (2019-nCoV). Biosci Trends. 28 janvier 2020. DOI 10.5582/bst.2020.01020.
- (en) « WHO says new China coronavirus could spread, warns hospitals worldwide », Reuters, (lire en ligne, consulté le ).
- « China Testing HIV Drug as Treatment for New Coronavirus, AbbVie Says », The New York Times, (lire en ligne).
- (en) Zhijian Xu, Cheng Peng, Yulong Shi et Zhengdan Zhu, « Nelfinavir was predicted to be a potential inhibitor of 2019 nCov main protease by an integrative approach combining homology modelling, molecular docking and binding free energy calculation », bioRxiv, Pharmacology and Toxicology, (DOI 10.1101/2020.01.27.921627, lire en ligne, consulté le ).
- Abdo Elfiky, « Sofosbuvir Can Inhibit the Newly Emerged Coronavirus (2019-nCoV) in Wuhan, China », (Preprints with the Lancet, Powered by SSRN), .
- « Thai doctors have been using a cocktail of flu and HIV drugs to treat coronavirus cases », Business Insider, (lire en ligne, consulté le ).
- « Coronavirus: Thailand has apparent treatment success with antiviral drug cocktail », South China Morning Post, (lire en ligne, consulté le ).
- (en) Michelle L. Holshue, Chas DeBolt, Scott Lindquist, Kathy H. Lofy, John Wiesman, Hollianne Bruce, Christopher Spitters, Keith Ericson, Sara Wilkerson, Ahmet Tural, George Diaz, Amanda Cohn, LeAnne Fox, Anita Patel, Susan I. Gerber, Lindsay Kim, Suxiang Tong, Xiaoyan Lu, Steve Lindstrom, Mark A. Pallansch, William C. Weldon, Holly M. Biggs, Timothy M. Uyeki, et Satish K. Pillai, « First Case of 2019 Novel Coronavirus in the United States », The New England Journal of Medicine, (PMID 32004427, DOI 10.1056/NEJMoa2001191, lire en ligne, consulté le ).
- (en) « Q&A on coronaviruses » [archive du ], sur Organisation mondiale de la santé (WHO) (consulté le ).
- (en) « China CDC developing novel coronavirus vaccine » [archive du ], sur Xinhua, (consulté le ).
- "Nature, 4/02/20: Remdesivir and chloroquine effectively inhibit the recently emerged novel coronavirus (2019-nCoV) in vitro".
- « DrugVirus.info », sur drugvirus.info (consulté le ).
- (en) "Cell, 5/3/20:SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a Clinically Proven Protease Inhibitor"
- « SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a Clinically Proven Protease Inhibitor », ScienceDirect=, (lire en ligne).
- (en) "5/3/20, cell:SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a Clinically Proven Protease Inhibitor "
- « Lancement d’un essai clinique européen contre le Covid-19 », SiteInserm, (lire en ligne).
- « COVID-19 funding opportunities », sur Reacting, (consulté le ).
- (en) Micholas Smith, Jeremy C. Smith, « Repurposing Therapeutics for COVID-19: Supercomputer-Based Docking to the SARS-CoV-2 Viral Spike Protein and Viral Spike Protein-Human ACE2 Interface, p11/31 Table3 », ChemRxiv, (lire en ligne)
- (en) Abhrajit Bag, Arijit Bag, « Treatment of COVID-19 patients: Justicia adhatoda leaves extract is a strong remedy for COVID-19 – Case report analysis and docking based study, p6/18 », ChemRxiv, (lire en ligne)
- (en) RÈNE DROGOU, « Un premier essai décevant pour le lopinavir-ritonavir », Le quotidien du medecin, (lire en ligne)
- Kai Kupferschmidt, « WHO launches global megatrial of the four most promising coronavirus treatments », Science, (ISSN 0036-8075 et 1095-9203, DOI 10.1126/science.abb8497, lire en ligne, consulté le ).
- « Appel flash Covid-19 : Premier financement immédiat pour le démarrage de 44 projets urgents », sur Agence nationale de la recherche (consulté le ).
- (en) Els Keyaerts, Leen Vijgen, Piet Maes et Johan Neyts, « In vitro inhibition of severe acute respiratory syndrome coronavirus by chloroquine », Biochemical and Biophysical Research Communications, vol. 323, no 1, , p. 264–268 (PMID 15351731, PMCID PMC7092815, DOI 10.1016/j.bbrc.2004.08.085, lire en ligne, consulté le ).
- (en) Martin J Vincent, Eric Bergeron, Suzanne Benjannet et Bobbie R Erickson, « Chloroquine is a potent inhibitor of SARS coronavirus infection and spread », Virology Journal, vol. 2, no 1, , p. 69 (DOI 10.1186/1743-422X-2-69, lire en ligne, consulté le ).
- (en) Christophe Biot, Wassim Daher, Natascha Chavain et Thierry Fandeur, « Design and Synthesis of Hydroxyferroquine Derivatives with Antimalarial and Antiviral Activities », Journal of Medicinal Chemistry, vol. 49, no 9, , p. 2845–2849 (ISSN 0022-2623 et 1520-4804, DOI 10.1021/jm0601856, lire en ligne, consulté le ).
- (en) « Chloroquine is a potent inhibitor of SARS coronavirus infection and spread », SpingerLink, (lire en ligne).
- (en) Meijuan Zheng, Yong Gao, Gang Wang et Guobin Song, « Functional exhaustion of antiviral lymphocytes in COVID-19 patients », Cellular & Molecular Immunology, (ISSN 1672-7681 et 2042-0226, PMCID PMC7091858, DOI 10.1038/s41423-020-0402-2, lire en ligne, consulté le ).
- (en) « Diagnosis and Treatment Protocol for Novel Coronavirus Pneumonia, p8/17 », National Health Commission & State Administration of Traditional Chinese Medicine, (lire en ligne).
- « Breakthrough: Chloroquine phosphate has shown apparent efficacy in treatment of COVID-19 associated pneumonia in clinical studie », BioScienceTrends, (lire en ligne).
- « La chloroquine est-elle un bon traitement contre le coronavirus ? », sur Sciences et Avenir (consulté le ).
- (en) Multicenter collaboration group of Department of Science and Technology of Guangdong Province and Health Commission of Guangdong Province for chloroquine in the treatment of novel coronavirus pneumonia, « [Expert consensus on chloroquine phosphate for the treatment of novel coronavirus pneumonia] », Zhonghua Jie He He Hu Xi Za Zhi = Zhonghua Jiehe He Huxi Zazhi = Chinese Journal of Tuberculosis and Respiratory Diseases, vol. 43, no 0, , E019 (ISSN 1001-0939, PMID 32075365, DOI 10.3760/cma.j.issn.1001-0939.2020.0019, lire en ligne, consulté le ).
- (en) « A systematic review on the efficacy and safety of chloroquine for the treatment of COVID-19 », ScienceDirect, (lire en ligne).
- (en) Philippe Colson, Jean-Marc Rolain et Didier Raoult, « Chloroquine for the 2019 novel coronavirus SARS-CoV-2 », International Journal of Antimicrobial Agents, , p. 105923 (DOI 10.1016/j.ijantimicag.2020.105923, lire en ligne, consulté le ).
- « Coronavirus : le professeur Raoult, spécialiste des maladies infectieuses, valide un traitement contre le Covid-19 », sur France 3 Provence-Alpes-Côte d'Azur (consulté le ).
- IHU Méditerranée-Infection, « Coronavirus : diagnostiquons et traitons ! Premiers résultats pour la chloroquine », .
- « ”Fake news” et désinformation autour du coronavirus SARS-CoV2 », Communiqué de presse, Inserm, (consulté le ).
- Pauline Moullot, « La chloroquine, un médicament contre le paludisme, est-elle efficace contre le Covid-19 ? », Libération, (consulté le ).
- « Après une mort, l'enthousiasme de Trump pour la chloroquine questionné », Paris Match (consulté le ).
- (en + zh) CHEN Jun, LIU Danping, LIU Li, LIU Ping, XU Qingnian, XIA Lu, LING Yun, HUANG Dan, SONG Shuli, ZHANG Dandan, QIAN Zhiping, LI Tao, SHEN Yinzhong & LU Hongzhou, « A pilot study of hydroxychloroquine in treatment of patients with common coronavirus disease-19 (COVID-19) », Journal of ZheJiang University (Medical Sciences), (lire en ligne).
- (en) Philippe Gautret, Jean Christophe Lagier, Philippe Parola et Van Thuan Hoang, « Hydroxychloroquine and Azithromycin as a treatment of COVID-19: preliminary results of an open-label non-randomized clinical trial », medRxiv, , p. 2020.03.16.20037135 (DOI 10.1101/2020.03.16.20037135, lire en ligne, consulté le ).
- (en) Philippe Gautret, Jean-Christophe Lagier, Philippe Parola et Van Thuan Hoang, « Hydroxychloroquine and azithromycin as a treatment of COVID-19: results of an open-label non-randomized clinical trial », International Journal of Antimicrobial Agents, , p. 105949 (DOI 10.1016/j.ijantimicag.2020.105949, lire en ligne, consulté le ).
- « Exclusif - Coronavirus : « On sait guérir la maladie », affirme l'infectiologue Didier Raoult », Les Échos, (consulté le ).
- (en) Katie Thomas et Denise Grady, « Trump’s Embrace of Unproven Drugs to Treat Coronavirus Defies Science », The New York Times, (consulté le ).
- (en) Philippe Gautret, Jean-Christophe Lagier, Philippe Parola et Van Thuan Hoang, « Hydroxychloroquine and azithromycin as a treatment of COVID-19: results of an open-label non-randomized clinical trial », International Journal of Antimicrobial Agents, , p. 105949 (DOI 10.1016/j.ijantimicag.2020.105949, lire en ligne, consulté le ).
- « Chloroquine : un remède français contre le Covid-19 ? », France Culture, (consulté le ).
- (en) Dahly D., Gates S. & Morris T., « Statistical review of Hydroxychloroquine and azithromycin as a treatment of COVID-19: results of an open-label non-randomized clinical trial », Zenodo preprints, (lire en ligne).
- (en) Author eliesbik, « Thoughts on the Gautret et al. paper about Hydroxychloroquine and Azithromycin treatment of COVID-19 infections », sur Science Integrity Digest, (consulté le ).
- (en) « Hydroxychloroquine and Azithromycin as a treatment of COVID-19: preliminary results of an open-label non-randomized clinical trial », sur PubPeer.
- « L’éthique de la recherche et l’usage du placebo : un état de la question au Canada », Site Medecinessciences.org, (lire en ligne).
- « Données Ihu méditérannée, covid 19 », Ihu méditérannée, (lire en ligne)
- « Coronavirus : situation toujours grave en France, masques, respirateurs », journaldesfemme, (lire en ligne)
- « Coronavirus nombre de cas à Paris et en région parisienne », Site eficiens, (lire en ligne)
- « Clinical and microbiological effect of a combination of hydroxychloroquine and azithromycin in 80 COVID-19 patients with at least a six-day follow up: an observational study », Site mediterranee-infection, (lire en ligne).
- (en) « Azithromycin induces anti-viral responses in bronchial epithelial cells », European Respiratory Journal 2010, (lire en ligne)
- (en) « Energetics Based Modeling of Hydroxychloroquine and Azithromycin Binding to the SARS-CoV-2 Spike (S)Protein - ACE2 Complex », ChemRxIv, (lire en ligne)
- (en) « Azithromycin Reduces Exaggerated Cytokine Production by M1 Alveolar Macrophages in Cystic Fibrosis », ATSjournal, (lire en ligne)
- « Etude Hycovid – Foire Aux Questions (FAQ) », Chu Angers, (lire en ligne)
- « Décryptages » Covid-19 et chloroquine : et si l'on écoutait le Dr Vladimir Zelenko nous parler de sa propre expérience ? », Atlantico, (lire en ligne)
- « DIRECT. Le gouvernement américain tente de développer un vaccin contre le coronavirus 2019-nCoV », sur Franceinfo, (consulté le ).
- « Coronavirus : « La France travaille à un vaccin » », La Croix, (ISSN 0242-6056, lire en ligne, consulté le ).
- (en) Tung Thanh Le, Zacharias Andreadakis, Arun Kumar et Raúl Gómez Román, « The COVID-19 vaccine development landscape », Nature Reviews Drug Discovery, , d41573–020–00073-5 (ISSN 1474-1776 et 1474-1784, DOI 10.1038/d41573-020-00073-5, lire en ligne, consulté le )
- Pierre Sautreuil, « Coronavirus : mais au fait, c'était quoi le Sras ? », sur Le Figaro.fr, (consulté le ).
- « COVID-19 : S’inspirer du SRAS pour développer le vaccin », sur santé log, (consulté le ).
- (en) Xiaolong Tian, Cheng Li, Ailing Huang et Shuai Xia, « Potent binding of 2019 novel coronavirus spike protein by a SARS coronavirus-specific human monoclonal antibody », bioRxiv, Microbiology, (DOI 10.1101/2020.01.28.923011, lire en ligne, consulté le ).
- « Coronavirus: un vaccin en cours d'élaboration en Russie », sur Le Figaro.fr, (consulté le ).
- (en) « Coronavirus 'may become global emergency' as 18 deaths confirmed », sur Sky News (consulté le ).
- (en) Haroon Siddique, Ben Quinn, Stephanie Convery et Libby Brooks, « China coronavirus: 14 test negative in UK as military doctors sent to Wuhan – as it happened », The Guardian, (ISSN 0261-3077, lire en ligne, consulté le ).
- (en) « Coronavirus: 'Significant breakthrough' in race for vaccine made by UK scientists », sur Sky News (consulté le ).
- (en) « Imperial researchers in race to develop a coronavirus vaccine | Imperial News | Imperial College London », sur Imperial News (consulté le ).
- « Coronavirus : « La France travaille à un vaccin » », La Croix, (ISSN 0242-6056, lire en ligne, consulté le ).
- (de) « Gesundheitssenatorin begrüßt Ausweitung des Risikogebiets durch Robert-Koch-Institut », sur www.berlin.de, (consulté le ).
- Jop Vrieze, « Can a century-old TB vaccine steel the immune system against the new coronavirus? », Science, (ISSN 0036-8075 et 1095-9203, DOI 10.1126/science.abb8297, lire en ligne, consulté le ).
- (en) « COVID-19 and Angiotensin II, The Plot Thickens, 8 min », Youtube, (lire en ligne)
- (en) « The Role of Angiotensin II in the Process of Atherosclerosis, 1min17 », Youtube, (lire en ligne)
- (en) « COVID-19: The role of Angiotensin-2 Receptor in Transmission Process », Journal of Regenerative Biology and Medicine, (lire en ligne)
- (en) « Angiotensin-converting enzyme 2 (ACE2) mediates influenza H7N9 virus-induced acute lung injury », Nature, (lire en ligne)
- (en) « Renin-angiotensin system inhibitors improve the clinical outcomes of COVID-19 patients with hypertension », Emerging Microbes & Infections, (lire en ligne)
- (en) « Homocysteine directly interacts and activates the angiotensin II type I receptor to aggravate vascular injury », Nature, (lire en ligne)
- (en) « Predictors for imaging progression on chest CT from coronavirus disease 2019 (COVID-19) patients. », {{Article}} : paramètre «
périodique» manquant, (lire en ligne) - (en) Harlan Barker and Seppo Parkkila, « Bioinformatic characterization of angiotensin-converting enzyme 2,the entry receptor for SARS-CoV-2 », Journal of Regenerative Biology and Medicine, (lire en ligne)
- (en) Harlan Barker and Seppo Parkkila, « Distribution of type-1 and type-2 angiotensin receptors in the normal human lung and in lungs from patients with chronic obstructive pulmonary disease. », Nbci, (lire en ligne)
- (en) Harlan Barker and Seppo Parkkila, « Distribution of type-1 and type-2 angiotensin receptors in the normal human lung and in lungs from patients with chronic obstructive pulmonary disease. », Nbci, (lire en ligne)
- (en) Harlan Barker and Seppo Parkkila, « The Angiotensin II Receptor 2 Is Expressed and MediatesAngiotensin II Signaling in Lung Fibrosis », Resp Cell Mol Biol, (lire en ligne)
- (en) Harlan Barker and Seppo Parkkila, « Senescence of Alveolar Stem Cells Drives Progressive Pulmonary Fibrosis », {{Article}} : paramètre «
périodique» manquant, (lire en ligne) - (en) Harlan Barker and Seppo Parkkila, « Protective Effect of Valsartan on Beleomycine-Induced Fibrosis », JUNDISHAPUR JOURNAL OF NATURAL PHARMACEUTICAL PRODUCTS, (lire en ligne)
- (en) Harlan Barker and Seppo Parkkila, « Les récepteurs AT1 et AT2 de l’angiotensine II », {{Article}} : paramètre «
périodique» manquant, (lire en ligne) - (en) Haibo Zhang et all, « Angiotensin-converting enzyme 2 (ACE2) as a SARS-CoV-2 receptor: molecular mechanisms and potential therapeutic target », SpringerLink, (lire en ligne)
- (en) « ESR Connect with Josef Penninger: ACE2 – a rational frontline therapy for COVID-19 », Youtube, (lire en ligne).
- (en) « Angiotensin 1-7 a comprehensive review, p137 », SpringerLink, (lire en ligne)
- (en) « The Anti-Inflammatory Potential of ACE2/Angiotensin-(1-7)/Mas Receptor Axis: Evidence from Basic and Clinical Research. », Ncbi, (lire en ligne)
- (en) « Angiotensin-Converting Enzyme 2: The First Decade, p7/13 », International Journal of Hypertension, (lire en ligne)
- (en) « Experimental drug APN01 prevents COVID-19 infection in the lab », News Medical, (lire en ligne)
- (en) « Expression of Human ACE2 in Lactobacillus and Beneficial Effects in Diabetic Retinopathy in Mice », Cell review, (lire en ligne)
- (en) « Circulating ACE2 activity is increased in patients with type 1 diabetes and vascular complications », Review of Hypertension, (lire en ligne)
- « Les autorités clarifient la liste des personnes à risque face au coronavirus », Le Figaro, (lire en ligne).
- (en) « Are patients with hypertension and diabetes mellitus at increased risk for COVID-19 infection? », Lancet, (lire en ligne).
- (en) « Two Promising SARS-CoV-2 Blocking Compounds Undergo Testing », Genegnews, (lire en ligne)
- (en) Lin Li, Ting Sun, Yufei He et Wendong Li, « Epitope-based peptide vaccine design and target site characterization against novel coronavirus disease caused by SARS-CoV-2 », bioRxiv, , p. 2020.02.25.965434 (DOI 10.1101/2020.02.25.965434, lire en ligne, consulté le ).
- (en) « Vir Biotechnology discovers two antibodies against new coronavirus » (consulté le ).
- (en) Anurodh Shankar Agrawal, Tianlei Ying, Xinrong Tao et Tania Garron, « Passive Transfer of A Germline-like Neutralizing Human Monoclonal Antibody Protects Transgenic Mice Against Lethal Middle East Respiratory Syndrome Coronavirus Infection », Scientific Reports, vol. 6, no 1, , p. 1–8 (ISSN 2045-2322, DOI 10.1038/srep31629, lire en ligne, consulté le ).
- (en) Jia Liu, Xin Zheng, Qiaoxia Tong et Wei Li, « Overlapping and discrete aspects of the pathology and pathogenesis of the emerging human pathogenic coronaviruses SARS-CoV, MERS-CoV, and 2019-nCoV », Journal of Medical Virology, (ISSN 1096-9071, DOI 10.1002/jmv.25709, lire en ligne, consulté le ).
- (en) « COVID-19 Docking Server: An interactive server for docking small molecules, peptides and antibodies against potential targets of COVID-19 », Cornell University, (lire en ligne)
- (en) « Cloud Immunity: Analyzing Billions of Antibodies for Next Generation Monoclonal », Youtube, (lire en ligne)
- (en) « Virus neutralization by antibodies », Virology.ws, (lire en ligne)
- (en) « A human monoclonal antibody blocking SARS-CoV-2 infection », biorxiv.org, (lire en ligne)
- (en) « People can get the coronavirus more than once, experts warn — recovering does not necessarily make you immune », sur Business Insider France (consulté le ).
- (en) Lan Lan, Dan Xu, Guangming Ye et Chen Xia, « Positive RT-PCR Test Results in Patients Recovered From COVID-19 », JAMA, (DOI 10.1001/jama.2020.2783, lire en ligne, consulté le ).
- (en) « Choc cytokinique : les essais thérapeutiques en cours dans la COVID-19 », Vidal, (lire en ligne)
- (en) « Un effet sur l'orage cytokinique:Le léronlimab », Le Quotidien du medecin, (lire en ligne)
- (en) « Treating coronavirus: How blood plasma from recovered patients may help », sur statnews.com, (consulté le ).
- « Coronavirus : Des chercheurs chinois ont identifié des anticorps prometteurs », LaTribune, (lire en ligne)
- (en) « Monoclonal Antibody Production [HD Animation] », Youtube, (lire en ligne)
- (en) « Analysis of a Therapeutic Antibody Cocktail Reveals Determinants for Cooperative and Broad Ebolavirus Neutralization. », Nbci, (lire en ligne)
- Hugo Mouquet, « Ingénierie de lymphocytes B humains produisant des anticorps neutralisant le virus VIH-1 par édition génique CRISPR-Cas9 », huffingtonpost, (lire en ligne).
- (en) « Antigen-specific humoral immune responses by CRISPR/Cas9-edited B cells », biorxiv, (lire en ligne).
- (en) « CRISPR/Cas9-Based Antiviral Strategy: Current Status and the Potential Challenge », Nbci, (lire en ligne).
- (en) « Efficacité de la vaccination contre la grippe saisonnière chez les personnes âgées et les professionnels de santé, p17/40 », Haut conseil de la santé publique, (lire en ligne).
- Roseup association, « Médecins du Monde s’oppose au brevet sur les CART-cells », Roseup assocation, (lire en ligne).
- (en) « Functional exhaustion of antiviral lymphocytes in COVID-19 patients », Nature, (lire en ligne).
- (en) « Decreased Counts of T Lymphocyte Subsets Predict Prognosis in SARS-CoV-2-Infected Pneumonia in Wuhan, China: a Retrospective Study », researchsquare, (lire en ligne).
- « Un médecin mosellan constate l’efficacité d’un protocole à base d’azithromycine », sur www.estrepublicain.fr (consulté le ).
- « L’éthique du traitement contre l’éthique de la recherche », sur www.lequotidiendumedecin.fr (consulté le ).
Voir aussi
Bibliographie
- Wang Y, Wang Y, Chen Y & Qin Q (2020) Unique epidemiological and clinical features of the emerging 2019 novel coronavirus pneumonia (COVID-19) implicate special control measures ; J Med Virol. 5 mars 2020 ; Epub 2020 Mar 5 (résumé).
Articles connexes
- Virologie
- Pandémie de maladie à coronavirus de 2019-2020
- Maladie à coronavirus 2019
- SARSr-CoV (espèce)
- SARS-CoV (coronavirus du SRAS)
- Syndrome respiratoire aigu sévère (SRAS)
- Coronavirus du syndrome respiratoire du Moyen-Orient
- Enzyme de conversion de l'angiotensine 2 (ACE2), porte d'entrée du virus
- Test diagnostique du SARS-CoV-2
Liens externes
- CDC chinois (Chinese Center for Disease Control and Prevention)
- Institut de Biologie des Agents pathogènes (Institute of Pathogen Biology)
- Carte de l'épidémie actualisée en temps réel par le Center for Systems Science and Engineering de la Johns Hopkins University de Baltimore
- (en-US) Gardner L., « Mapping the Wuhan Coronavirus (2019-nCoV) », sur systems.jhu.edu, (consulté le )
- Questions fréquentes sur les nouveaux coronavirus et Rapports quotidiens de situation sur le nouveau Coronavirus (2019-nCoV) [en anglais] par l'Organisation Mondiale de la Santé
- Fiche-maladie régulièrement actualisée par l'Institut Pasteur
- « Coronavirus 2019-nCoV : le point sur la situation en France et dans le monde », sur le site du Dictionnaire Vidal (consulté le ).
- "Analyses bioinformatiques du coronavirus 2019-nCoV : pourquoi et comment ?", article rédigé par deux bioinformaticiens sur bioinfo-fr.net, 05 février 2020
- "Conseil scientifique COVID-19", Avis du Conseil scientifique COVID-19 (.fr)
- "Infection au nouveau Coronavirus (SARS-CoV-2), COVID-19, France et Monde", Chiffres officiels français, des derniers jours.