« Protéine » : différence entre les versions
m remplacement modèle {{lien}}, article existant sur fr |
Compléments. |
||
| Ligne 470 : | Ligne 470 : | ||
Les protéines remplissent ainsi des fonctions très diverses au sein de la cellule et de l'organisme<ref>{{Biologie moléculaire de la cellule}}</ref>: |
Les protéines remplissent ainsi des fonctions très diverses au sein de la cellule et de l'organisme<ref>{{Biologie moléculaire de la cellule}}</ref>: |
||
* les ''protéines |
* les '''protéines structurelles''', qui permettent à la cellule de maintenir son organisation dans l'espace, et qui sont les constituants du [[cytosquelette|cytosquelette]] ; |
||
* les ''protéines de transport'', qui assurent le transfert des différentes molécules dans et en dehors des cellules |
* les '''protéines de transport''', qui assurent le transfert des différentes molécules dans et en dehors des cellules ; |
||
* les ''protéines régulatrices'', qui modulent l'activité d'autres protéines ou qui contrôlent l'expression des gènes |
* les '''protéines régulatrices''', qui modulent l'activité d'autres protéines ou qui contrôlent l'[[Expression génétique|expression des gènes]] ; |
||
* les ''protéines de signalisation'', qui captent les signaux extérieurs, et assurent leur transmission dans la cellule ou l'organisme ; il en existe plusieurs sortes |
* les '''protéines de signalisation''', qui captent les signaux extérieurs, et assurent leur transmission dans la cellule ou l'organisme ; il en existe plusieurs sortes, par exemple les '''protéines hormonales''', qui contribuent à coordonner les activités d'un organisme en agissant comme des signaux entre les cellules ; |
||
* les '''protéines réceptrices''', qui détectent les molécules messagères et les autres signaux pour que la cellule agisse en conséquence : |
|||
** les ''protéines |
** les '''protéines sensorielles''' détectent les signaux environnementaux (ex. : lumière) et répondent en émettant des signaux dans la cellule ; |
||
** les '' |
** les '''récepteurs d'hormone''' détectent les [[hormone]]s et envoient des signaux à la cellule pour qu'elle agisse en conséquence (ex. : l'[[insuline]] est une hormone qui, lorsqu'elle est captée, signale à la cellule d'absorber et d'utiliser le [[glucose]]) ; |
||
| ⚫ | |||
** les '' récepteurs d'hormone '' détectent les hormones et envoient des signaux à la cellule pour qu'elle agisse en conséquence (ex. : l'insuline est une hormone qui lorsqu'elle va être captée, va signaler à la cellule d'absorber et d'utiliser le sucre) ; |
|||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
== Méthodes d'étude == |
== Méthodes d'étude == |
||
La structure et les fonctions des protéines peuvent être étudiées ''[[in vivo]]'', ''[[in vitro]]'' et ''[[in silico]]''. Les études ''in vivo'' permettent d'explorer le rôle [[Physiologie|physiologique]] d'une protéine au sein d'une [[Cellule (biologie)|cellule vivante]] ou même au sein d'un [[Organisme (physiologie)|organisme]] dans son ensemble. Les études ''in vitro'' de protéines purifiées dans des environnements contrôlés sont utiles pour comprendre la façon dont une protéine fonctionne ''in vivo'' : par exemple, l'étude de la [[Cinétique enzymatique|cinétique]] d'une[[enzyme]] permet d'analyser le [[Mécanisme réactionnel|mécanisme chimique]] de son activité [[Catalyse|catalytique]] et de son [[Affinité (biochimie)|affinité]] relative vis-à-vis de différents [[Substrat enzymatique|substrats]]. Les études '' |
La structure et les fonctions des protéines peuvent être étudiées ''[[in vivo]]'', ''[[in vitro]]'' et ''[[in silico]]''. Les études ''in vivo'' permettent d'explorer le rôle [[Physiologie|physiologique]] d'une protéine au sein d'une [[Cellule (biologie)|cellule vivante]] ou même au sein d'un [[Organisme (physiologie)|organisme]] dans son ensemble. Les études ''in vitro'' de protéines purifiées dans des environnements contrôlés sont utiles pour comprendre la façon dont une protéine fonctionne ''in vivo'' : par exemple, l'étude de la [[Cinétique enzymatique|cinétique]] d'une [[enzyme]] permet d'analyser le [[Mécanisme réactionnel|mécanisme chimique]] de son activité [[Catalyse|catalytique]] et de son [[Affinité (biochimie)|affinité]] relative vis-à-vis de différents [[Substrat enzymatique|substrats]]. Les études ''in silico'' utilisent des [[algorithme]]s [[informatique]]s pour [[Modèle mathématique|modéliser]] des protéines. |
||
=== Purification des protéines === |
=== Purification des protéines === |
||
| Ligne 491 : | Ligne 490 : | ||
{{Article détaillé|purification de protéines}} |
{{Article détaillé|purification de protéines}} |
||
Pour pouvoir être analysée ''[[in vitro]]'', une protéine doit préalablement avoir été purifiée des autres constituants chimiques de la cellule. Ceci commence généralement par la [[Lyse (biologie)|lyse]] de la cellule, au cours de laquelle la [[membrane plasmique]] est rompue afin d'en libérer le contenu dans une solution pour donner un lysat. Ce mélange peut être purifié par [[ultracentrifugation]], ce qui permet d'en séparer les constituants en fractions contenant respectivement les protéines solubles, les [[lipide]]s et [[Protéine membranaire|protéines membranaires]], les [[organite]]s cellulaires, et les [[Acide nucléique|acides nucléiques]]. La [[Précipité|précipitation]] des protéines par [[relargage]] permet de les concentrer à partir de ce lysat. Il est alors possible d'utiliser plusieurs types de [[chromatographie]] pour isoler les protéines que l'on souhaite étudier en fonction de leurs propriétés [[Physico-chimie|physico-chimiques]] telles que leur [[masse molaire]], leur [[charge électrique]], ou encore leur affinité de liaison. Le degré de purification peut être suivi à l'aide de plusieurs types d'[[électrophorèse sur gel]] si la masse moléculaire et le [[point isoélectrique]] des protéines étudiées sont connus, par [[spectroscopie]] si la protéine présente des caractéristiques spectroscopiques identifiables, ou par {{Lien|trad=Enzyme assay|lang=en|fr=dosage enzymatique}} si la protéine est porteuse d'une activité enzymatique. Par ailleurs |
Pour pouvoir être analysée ''[[in vitro]]'', une protéine doit préalablement avoir été purifiée des autres constituants chimiques de la cellule. Ceci commence généralement par la [[Lyse (biologie)|lyse]] de la cellule, au cours de laquelle la [[membrane plasmique]] est rompue afin d'en libérer le contenu dans une solution pour donner un lysat. Ce mélange peut être purifié par [[ultracentrifugation]], ce qui permet d'en séparer les constituants en fractions contenant respectivement les protéines solubles, les [[lipide]]s et [[Protéine membranaire|protéines membranaires]], les [[organite]]s cellulaires, et les [[Acide nucléique|acides nucléiques]]. La [[Précipité|précipitation]] des protéines par [[relargage]] permet de les concentrer à partir de ce lysat. Il est alors possible d'utiliser plusieurs types de [[chromatographie]] pour isoler les protéines que l'on souhaite étudier en fonction de leurs propriétés [[Physico-chimie|physico-chimiques]] telles que leur [[masse molaire]], leur [[charge électrique]], ou encore leur affinité de liaison. Le degré de purification peut être suivi à l'aide de plusieurs types d'[[électrophorèse sur gel]] si la masse moléculaire et le [[point isoélectrique]] des protéines étudiées sont connus, par [[spectroscopie]] si la protéine présente des caractéristiques spectroscopiques identifiables, ou par {{Lien|trad=Enzyme assay|lang=en|fr=dosage enzymatique}} si la protéine est porteuse d'une activité enzymatique. Par ailleurs, les protéines peuvent être isolées en fonction de leur charge électrique par {{Lien|trad=Isoelectric focusing|lang=en|fr=électrofocalisation}}<ref name="10.1007/978-1-60327-064-9_19"> |
||
{{Article |
{{Article |
||
| langue = en |
| langue = en |
||
| Ligne 527 : | Ligne 526 : | ||
| pmid = 12536251 |
| pmid = 12536251 |
||
| pmc = |
| pmc = |
||
}}</ref>. |
|||
=== Localisation cellulaire === |
|||
[[Fichier:Localisations02eng.jpg|redresse=1.5|vignette|{{en}} Localisation de protéines marquées à la [[protéine fluorescente verte]], apparaissant en blanc, dans différents {{Lien|trad=Cellular compartment|lang=en|fr=Compartiment cellulaire|texte=compartiments cellulaires}}.]] |
|||
L'étude ''[[in vivo]]'' des protéines implique souvent de savoir précisement où elles sont synthétisées et où elles se trouvent dans les cellules. Bien que la plupart des protéines intracellulaires soient produites dans le [[cytoplasme]] et que la plupart des protéines membranaires ou sécrétées dans le milieux extracellulaire sont produites dans le [[réticulum endoplasmique]], il est rare qu'on comprenne précisément comment les protéines ciblent spécifiquement certaines structures cellulaires ou certains [[organite]]s. Le [[génie génétique]] offre des outils utiles pour se faire une idée de la localisation de certaines protéines, par exemple en liant la protéine étudiée à une protéine permettant de la repérer, c'est-à-dire en réalisant une [[protéine de fusion]] entre la protéine étudiée et une protéine utilisée comme marqueur, telle que la [[protéine fluorescente verte]]<ref name="10.2174/138920308785132668"> |
|||
{{Article |
|||
| langue = en |
|||
| nom1 = Olesya V. Stepanenko, Vladislav V. Verkhusha, Irina M. Kuznetsova, Vladimir N. Uversky and K. K. Turoverov |
|||
| titre = Fluorescent Proteins as Biomarkers and Biosensors: Throwing Color Lights on Molecular and Cellular Processes |
|||
| périodique = Current Protein & Peptide Science |
|||
| volume = 9 |
|||
| numéro = 4 |
|||
| jour = |
|||
| mois = août |
|||
| année = 2008 |
|||
| pages = 338-369 |
|||
| url texte = http://www.eurekaselect.com/83028/article |
|||
| consulté le = 15 mai 2015 |
|||
| doi = 10.2174/138920308785132668 |
|||
| pmid = 18691124 |
|||
| pmc = 2904242 |
|||
}}</ref>. La localisation intracellulaire de la protéine de fusion résultante peut être facilement et efficacement visualisée par [[microscopie]]<ref name="10.1038/nmeth1205-902"> |
|||
{{Article |
|||
| langue = en |
|||
| nom1 = Rafael Yuste |
|||
| titre = Fluorescence microscopy today |
|||
| périodique = Nature Methods |
|||
| volume = 2 |
|||
| numéro = 12 |
|||
| jour = |
|||
| mois = décembre |
|||
| année = 2005 |
|||
| pages = 902-904 |
|||
| url texte = http://www.nature.com/nmeth/journal/v2/n12/full/nmeth1205-902.html |
|||
| consulté le = 15 mai 2015 |
|||
| doi = 10.1038/nmeth1205-902 |
|||
| pmid = 16299474 |
|||
| pmc = |
|||
}}</ref>. |
|||
D'autres méthodes de localisation intracellulaire des protéines impliquent l'utilisation de marqueurs connus pour certains {{Lien|trad=Cellular compartment|lang=en|fr=Compartiment cellulaire|texte=compartiments cellulaires}} tels que le [[réticulum endoplasmique]], l'[[appareil de Golgi]], les [[lysosome]]s, les [[mitochondrie]]s, les [[chloroplaste]]s, la [[membrane plasmique]], etc. Il est par exemple possible de localiser des protéines marquées avec une étiquette fluorescente ou ciblées avec des [[anticorps]] contre ces marqueurs. Les techniques d'[[immunofluorescence]] permettent ainsi de localiser des protéines spécifiques. Des pigments fluorescents sont également utilisés pour marquer des compartiments cellulaires dans un but similaire<ref name="10.1006/meth.1999.0906"> |
|||
{{Article |
|||
| langue = en |
|||
| nom1 = William Margolin |
|||
| titre = Green Fluorescent Protein as a Reporter for Macromolecular Localization in Bacterial Cells |
|||
| périodique = Methods |
|||
| volume = 20 |
|||
| numéro = 1 |
|||
| jour = |
|||
| mois = janvier |
|||
| année = 2000 |
|||
| pages = 62-72 |
|||
| url texte = http://www.sciencedirect.com/science/article/pii/S1046202399909064 |
|||
| consulté le = 15 mai 2015 |
|||
| doi = 10.1006/meth.1999.0906 |
|||
| pmid = 10610805 |
|||
| pmc = |
|||
}}</ref>. |
|||
L'[[immunohistochimie]] utilise généralement un anticorps ciblant une ou plusieurs protéines étudiées qui sont conjugués à des [[enzyme]]s émettant des signaux luminescents ou [[chromogène]]s pouvant être comparés à divers échantillons, ce qui permet d'en déduire des informations sur la localisation des protéines étudiées. Il est également possible d'utiliser des techniques de cofractionnement dans un gradient de [[saccharose]] (ou d'une autre substance) à l'aide d'une centrifugation isopycnique. |
|||
La microscopie immunoélectronique combine l'utilisation d'une [[microscopie électronique]] classique à l'utilisation d'un anticorps dirigé contre la protéine étudiée, cet anticorps étant préalablement conjugué à un matériau à forte densité électronique telle que l'[[or]]. Ceci permet de localiser des détails ultrastructurels ainsi que la protéine étudiée<ref name="10.1007/s00418-008-0451-6"> |
|||
{{Article |
|||
| langue = en |
|||
| nom1 = Terry M. Mayhew et John M. Lucocq |
|||
| titre = Developments in cell biology for quantitative immunoelectron microscopy based on thin sections: a review |
|||
| périodique = Histochemistry and Cell Biology |
|||
| volume = 130 |
|||
| numéro = 2 |
|||
| jour = |
|||
| mois = août |
|||
| année = 2008 |
|||
| pages = 299-313 |
|||
| url texte = http://link.springer.com/article/10.1007%2Fs00418-008-0451-6 |
|||
| consulté le = 15 mai 2015 |
|||
| doi = 10.1007/s00418-008-0451-6 |
|||
| pmid = 18553098 |
|||
| pmc = 2491712 |
|||
}}</ref>. |
}}</ref>. |
||
Version du 15 mai 2015 à 23:10


Une protéine est une macromolécule biologique formée d'une ou de plusieurs chaînes polypeptidiques. Chacune de ces chaînes est constituée de résidus d'acides aminés liés entre eux par des liaisons peptidiques. On parle généralement de protéine au-delà d'une cinquantaine de résidus dans la molécule[1], et de peptide jusqu'à quelques dizaines de résidus. Les protéines sont codées par des gènes, qui spécifient 22 acides aminés, dits protéinogènes, qui sont incorporés directement par les ribosomes lors de la biosynthèse des protéines. La succession des acides aminés le long de la chaîne polypeptidique est appelée séquence du polypeptide. La séquence peptidique d'une protéine est directement liée à la séquence nucléotidique de l'ADN des gènes qui codent cette protéine ; la séquence peptidique dérive de la séquence nucléotidique à travers le code génétique. Des modifications post-traductionnelles peuvent cependant altérer significativement les résidus d'acides aminés une fois la protéine synthétisée, ce qui a pour effet d'en modifier les propriétés physico-chimiques. Il est également fréquent que des molécules non protéiques, appelées groupes prosthétiques, interagissent avec des protéines de façon déterminante pour leur fonction biologique : c'est par exemple le cas de l'hème dans l'hémoglobine, sans lequel cette protéine ne pourrait pas transporter l'oxygène dans le sang.
La nature des protéines est déterminée avant tout par leur séquence en acides aminés, qui constitue leur structure primaire. Les acides aminés ayant des propriétés chimiques fort diverses, leur disposition le long de la chaîne polypeptidique détermine leur arrangement spatial. Celui-ci est décrit localement par leur structure secondaire, stabilisée par des liaisons hydrogène entre résidus d'acides aminés voisins, et globalement par leur structure tertiaire, stabilisée par l'ensemble des interactions entre les résidus — parfois très éloignés sur la séquence peptidique mais mis en contact spatialement par le repliement de la protéine — ainsi qu'entre la protéine elle-même et son environnement ; la réticulation de plusieurs chaînes peptidiques entre elles par des ponts disulfure entre résidus de cystéine est également décrite au niveau de la structure tertiaire de la protéine. Enfin, l'assemblage de plusieurs sous-unités protéiques pour former un complexe fonctionnel est décrit par la structure quaternaire de cet ensemble.
Les protéines assurent une multitude de fonctions au sein de la cellule vivante et dans les tissus : rôle structurel (actine, collagène), dans la mobilité (myosine), dans le conditionnement de l'ADN (histones), dans la régulation de l'expression génétique (facteurs de transcription), dans la signalisation cellulaire (récepteurs membranaires) ou encore comme catalyseurs (enzymes).
Au laboratoire, elles peuvent être séparées des autres constituants cellulaires à l'aide de diverses techniques telles que l'ultracentrifugation, la précipitation, l'électrophorèse et la chromatographie. Le génie génétique a introduit un grand nombre de méthodes permettant de faciliter la purification des protéines. Leur structure peut être étudiée par immunohistochimie, par mutagenèse dirigée, par cristallographie aux rayons X, par résonance magnétique nucléaire et par spectrométrie de masse.
Biochimie
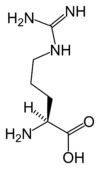
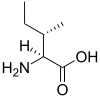
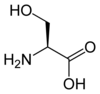
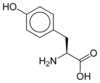
Les protéines sont formées d'une ou plusieurs chaînes polypeptidiques, qui sont des biopolymères linéaires éventuellement assez longs constitués d'une vingtaine d'acides L-α-aminés différents. Tous les acides aminés protéinogènes — à l'exception de la proline — partagent une structure commune, constituée d'une fonction acide carboxylique, d'une amine primaire sur le carbone α, et d'une chaîne latérale. Cette dernière présente une très grande variété de structures chimiques, et c'est l'effet combiné de toutes ces chaînes latérales d'une chaîne polypeptidique qui détermine sa structure tridimensionnelle ainsi que ses propriétés chimiques[2]. La planche ci-dessous présente la structure chimique des 22 acides aminés protéinogènes :
| Structure des 22 acides aminés protéinogènes. La pyrrolysine et la sélénocystéine (ci-dessus grisées) sont spécifiques à certaines protéines : - la pyrrolysine ne se rencontre que chez certaines archées méthanogènes, - la sélénocystéine est présente également chez les eucaryotes mais a priori dans quelques dizaines d'enzymes de la famille des oxydoréductases. Les 20 autres acides aminés, dits standards, sont en revanche universellement distribués chez tous les êtres vivants connus. |
Les acides aminés d'une chaîne polypeptidique sont liés entre eux par des liaisons peptidiques qui s'établissent entre le carboxyle –COOH d'un premier acide aminé et l'amine primaire –NH2 d'un second :

La liaison peptidique présente deux formes de résonance lui conférant en partie les propriétés d'une double liaison, ce qui limite les rotations autour de son axe, de sorte que les carbones α sont à peu près coplanaires. Les deux autres angles dièdres de la liaison peptidique déterminent le géométrie locale adoptée par le squelette constitué de la succession des liaisons peptidiques de la protéine. L'extrémité de la chaîne polypeptidique côté carboxyle est appelée extrémité C-terminale, tandis que celle côté amine est appelée extrémité N-terminale. Les mots protéine, polypeptide et peptide sont assez ambigus et leur sens peut se recouvrir. On parle généralement de protéine en référence à la molécule biologique complète dotée d'une conformation stable, tandis qu'un peptide désigne généralement une molécule plus courte dépourvue de structure tridimensionnelle stable. La limite entre les deux est très imprécise et se situe autour de quelques dizaines de résidus d'acides aminés[3].
Structure
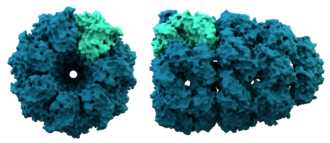

À gauche : représentation de tous les atomes et les leurs liaisons, chaque élément chimique étant représenté par une couleur différente.
Au centre : représentation de la conformation du squelette polypeptidique coloréé par structure secondaire.
À droite : surface moléculaire en contact avec le solvant colorée par type de résidu (acide en rouge, basique en bleu, polaire en vert, apolaire en blanc).
La plupart des protéines adoptent une conformation tridimensionnelle unique. La forme naturelle d'une protéine in vivo est son état natif, qui correspond à la forme qu'elle prend pour être biologiquement active et fonctionnelle. De nombreuses protéines prennent par elles-mêmes leur forme biologiquement active sous l'effet de la distribution spatiale des résidus d'acides aminés qui les constituent, d'autres ont besoin d'être assistées pour ce faire par des protéines chaperonnes pour être repliées selon leur état natif.
En biochimie, on distingue généralement quatre niveaux d'organisation pour décrire la structure des protéines :
- La structure primaire correspond à la séquence en acides aminés.
- La structure secondaire décrit l'arrangement des résidus d'acides aminés observable à l'échelle atomique. Stabilisées par des liaisons hydrogène, ces arrangements locaux sont par exemple les hélices α, les feuillets β, les tonneaux β, ou les coudes. Il en existe plusieurs variétés, et il est courant qu'une protéine possède globalement plusieurs types de structures secondaires.
- La structure tertiaire correspond à la forme générale de la protéine observable à l'échelle de la molécule toute entière. Elle décrit les interactions entre les différents éléments de la structure secondaire. Elle est stabilisée par tout un ensemble d'interactions conduisant le plus souvent à la formation d'un cœur hydrophobe, avec éventuellement des liaisons salines, des liaisons hydrogène, des ponts disulfure, voire des modifications post-traductionnelles. On désigne souvent par structure tertiaire le repliement d'une protéine.
- La structure quaternaire décrit le complexe résultant de l'assemblage de plusieurs molécules de protéines (plusieurs chaînes polypeptidiques), appelées dans ce cas sous-unités protéiques, pour former un complexe protéique unique. Toutes les protéines ne sont pas nécessairement constituées de plusieurs sous-unités et ne possèdent par conséquent pas toujours de structure quaternaire.
Les protéines ne sont pas des molécules entièrement rigides. Elles sont susceptible d'adopter plusieurs conformations apparentées en réalisant leurs fonctions biologiques. La transition d'une de ces conformations à une autre est appelée changement conformationnel. Dans le cas d'une enzyme par exemple, de tels changements conformationnels peuvent être induits par l'interaction avec le substrat au site actif. En solution, les protéines subissent également de nombreux changements conformationnels en raison de la vibration thermique de la collision avec d'autres molécules.

On peut distinguer trois grands groupes de protéines en fonction de leur structure tertiaire : les protéines globulaires, les protéines fibreuses et les protéines membranaires. Presque toutes les protéines globulaires sont solubles et ce sont souvent des enzymes. Les protéines fibreuses jouent souvent un rôle structurel, à l'instar du collagène, constituant principal des tissus conjonctifs, ou de la kératine, constituant protéique des poils et des ongles. Les protéines membranaires sont souvent des récepteurs ou des canaux permettant aux molécules polaires ou électriquement chargées de traverser la membrane.
Synthèse
Les acides α-aminés protéinogènes sont assemblés en polypeptides au sein des cellules par les ribosomes à partir de l'information génétique transmise par les ARN messagers depuis l'ADN constituant les gènes. C'est la séquence nucléotidique de l'ADN, transcrite à l'identique dans l'ARN messager, qui porte l'information lue par les ribosomes pour produire les protéines selon la séquence peptidique spécifié par les gènes. La correspondance entre la séquence nucléotidique de l'ADN et de l'ARN messager d'une part et la séquence peptidique des protéines synthétisées d'autre part est déterminée par le code génétique, qui est essentiellement le même pour tous les être vivants connus hormis un certain nombre de variantes assez limitées.
Code génétique

Le code génétique établit la correspondance entre un triplet de bases azotées, appelé codon, sur l'ARN messager et un acide α-aminé protéinogène. Cette correspondance est réalisée in vivo par les ARN de transfert, qui sont des ARN comptant une centaine de nucléotides tout au plus et portant un acide aminé esterifiant leur extrémité 3’-OH. Chacun des acides aminés est lié à des ARN de transfert spécifiques, portant des codons eux-aussi spécifiques, de sorte que chacun des 64 codons possibles ne peut coder qu'un seul acide aminé. En revanche, chacun des 22 acides aminés protéinogènes peut être codé par plusieurs codons différents. Ce sont en fait les enzymes réalisant l'estérification des ARN messagers avec les acides aminés — les aminoacyl-ARNt synthétases — qui maintiennent le code génétique : en effet, ces enzymes se lient spécifiquement à la fois à un ARN de transfert donné et à un acide aminé donné, de sorte que chaque type d'ARN de transfert n'est estérifié que par un acide aminé spécifique.
Le cas de la sélénocystéine et de la pyrrolysine est quelque peu différent en ce que ces acides aminés particuliers ne sont pas codés directement par des codons spécifiques mais par recodage traductionnel de codons stop en présence de séquences d'insertions particulières appelées respectivement élément SECIS et élément PYLIS, qui recodent les codons stop UGA (Opale) et UAG (Ambre) respectivement en sélénocystéine et en pyrrolysine. De surcroît, la sélénocystéine n'est pas liée telle quelle à son ARN de transfert, car elle est trop réactive pour exister librement dans la cellule sans l'endommager ; c'est en fait la sérine qui est liée à un ARN de transfert de sélénocystéine ARNtSec par la sérine-ARNt ligase. Le séryl-ARNtSec ne peut être utilisé par les ribosomes car il n'est pas reconnu par les facteurs d'élongation intervenant au cours de la biosynthèse des protéines, de sorte que la sérine ne peut être incorporée dans les sélénoprotéines à la place de la sélénocystéine. En revanche, le séryl-ARNtSec est un substrat de la sélénocystéine synthase, qui le convertit en sélénocystéinyl-ARNtSec.
Les gènes codés dans l'ADN sont tout d'abord transcrits en ARN pré-messager par des enzymes telles que les ARN polymérases. La plupart des êtres vivants modifient cet ARN pré-messager à travers un ensemble de processus appelés modifications post-transcriptionnelles conduisant à l'ARN messager mature. Ce dernier est alors utilisable par les ribosomes pour servir de modèle lors de la biosynthèse des protéines. Chez les procaryotes, l'ARN messager peut être utilisé dès qu'il est synthétisé ou être traduit en protéines après avoir quitté le nucléoïde. En revanche, chez les eucaryotes, l'ARN messager est produit dans le noyau de la cellule tandis que les protéines sont synthétisées dans le cytoplasme, de sorte que l'ARN messager doit traverser la membrane nucléaire.
Biosynthèse

La biosynthèse d'une protéine à partir d'un ARN messager est la traduction de cet ARNm. L'ARN messager se lie au ribosome, qui le lit séquentiellement à raison de trois nucléotides à chaque étape de la synthèse. Chaque triplet de trois nucléotides constitue un codon sur l'ARN messager, auquel peut se lier l'anticodon d'un ARN de transfert apportant l'acide aminé correspondant. L'appariement entre le codon et l'anticodon repose sur la complémentarité de leurs séquences respectives. C'est cette complémentarité qui assure la reconnaissance entre l'ARN de transfert et le codon de l'ARN messager. L'acide aminé apporté par l'ARN de transfert sur le ribosome établit une liaison peptidique avec l'extrémité C-terminale de la chaîne naissante, ce qui permet de l'allonger d'un résidu d'acide aminé. Le ribosome se déplace alors de trois nucléotides sur l'ARN messager pour faire face à un nouveau codon, qui suit exactement le codon précédent. Ce processus se répète jusqu'à ce que le ribosome soit en face d'un codon stop, auquel cas la traduction s'arrête.
La biosynthèse d'une protéine s'effectue ainsi résidu après résidu, de l'extrémité N-terminale vers l'extrémité C-terminale. Une fois synthétisée, la protéine peut subir diverses modifications post-traductionnelles telles que clivage, phosphorylation, acétylation, amidation, méthylation, glycosylation, lipidation, voire la formation de ponts disulfure. La taille des protéines ainsi synthétisées est très variable. Cette taille peut être exprimée en nombre de résidus d'acides aminés constituant ces protéines, ainsi qu'en daltons (symbole Da), qui correspondent en biologie moléculaire à l'unité de masse atomique. Les protéines étant souvent des molécules assez grosses, leur masse est souvent exprimée en kilodaltons (symbole kDa). À titre d'exemple, les protéines de levure ont une longueur moyenne de 466 résidus d'acides aminés, pour une masse de 53 kDa. Les plus grosses protéines connues sont les titines des sarcomères formant les myofibrilles des muscles striés[10] : la titine de souris contient quelque 35 213 résidus d'acides aminés formés de 551 739 atomes pour une masse de plus de 3 900 kDa et une longueur de l'ordre de 1 µm[11].
Synthèse chimique
Les petites protéines peuvent également être synthétisées par un ensemble de méthodes appelées synthèse peptidique, qui reposent sur des techniques de synthèse organique telles que la ligature chimique (en) pour produire efficacement des peptides[12]. La synthèse chimique permet d'introduire des acides aminés non naturels dans la chaîne polypeptidique, en posant par exemple des sondes fluorescentes sur la chaîne latérale de certains d'entre eux[13]. Ces méthodes sont utiles au laboratoire en biochimie et en biologie cellulaire mais ne sont généralement pas employées pour des applications commerciales. La synthèse chimique n'est pas efficace pour synthétiser des peptides de plus de 300 résidus d'acides aminés environ, et les protéines ainsi produites peuvent ne pas adopter facilement leur structure tertiaire native. La plupart des méthodes de synthèse chimique des protéines procèdent de l'extrémité C-terminale vers l'extrémité N-terminale, c'est-à-dire dans le sens inverse de la biosynthèse des protéines par les ribosomes[14].
Fonctions

Parmi tous les constituants de la cellule, les protéines sont les éléments les plus actifs. Hormis certains ARN, la plupart des autres molécules biologiques sont chimiquement assez peu réactives et ce sont les protéines qui agissent sur elles. Les protéines constituent environ la moitié de la matière sèche d'une cellule d'E. coli tandis que l'ARN et l'ADN en constituent respectivement un cinquième et 3 %. L'ensemble des protéines exprimées dans une cellule constitue son protéome.
La caractéristique principale des protéines qui leur permet de réaliser leurs fonctions biologiques est leur faculté de se lier à d'autres molécules de façon à la fois très spécifique et très étroite. La région d'une protéine permettant de se lier à une autre molécule est son site de liaison, qui forme souvent une dépression, une cavité, ou « poche », dans la surface de la molécule. C'est la structure tertiaire de la protéine et la nature chimique des chaînes latérales des résidus d'acides aminés du site de liaison qui déterminent la spécificité de cette interaction. Les sites de liaison peuvent conduire à des liaisons particulièrement spécifiques et étroites : ainsi, l'inhibiteur de la ribonucléase (en) se lie à l'angiogénine humaine avec une constante de dissociation sub-femtomolaire (< 10−15 mol L−1) mais ne se lie pas du tout à la ranpirnase, homologue d'amphibien de cette protéine (constante supérieure à 1 mol L−1). Une légère modification chimique peut radicalement modifier la faculté d'une molécule à interagir avec une protéine donnée. Ainsi l'aminoacyl-ARNt synthétase spécifique de la valine se lie à cette dernière sans interagir avec l'isoleucine, qui lui est pourtant structurellement très proche[15].
Les protéines peuvent se lier selon les cas à d'autres protéines ou à de petites molécules comme substrats. Lorsqu'elles se lient spécifiquement à d'autres protéines identiques à elles-mêmes, elles peuvent polymériser pour former des fibrilles. Ceci est fréquent pour les protéines structurelles, formées de monomères globulaires qui s'auto-assemblent pour former des fibres rigides. Des interactions protéine-protéine régulent également leur activité enzymatique, l'avancement du cycle cellulaire et l'assemblage de grands complexes protéiques réalisant des réactions étroitement apparentées partageant une fonction biologique commune. Les protéines peuvent également se lier à la surface des membranes cellulaires et même fréquemment en faire partie intégrante. La capacité de certaines protéines à changer de conformation lorsqu'elles se lient à des molécules spécifiques permet de construire des réseaux de signalisation cellulaire extrêmement complexes. D'une manière générale, l'étude des interactions entre protéines spécifiques est un élément clé de notre compréhension du fonctionnement des cellules et de leur faculté à échanger de l'information[16],[17].
Enzymes
Le rôle le plus visible des protéines dans la cellule est celui d'enzyme, c'est-à-dire de biomolécule catalysant des réactions chimiques. Les enzymes sont généralement très spécifiques et n'accélèrent qu'une ou quelques réactions chimiques. La très grande majorité des réactions chimiques du métabolisme sont réalisées par des enzymes. Outre le métabolisme, ces dernières interviennent également dans l'expression génétique, la réplication de l'ADN, la réparation de l'ADN, la transcription de l'ADN en ARN, et la traduction de l'ARN messager en protéines. Certaines enzymes agissent sur d'autres protéines pour y lier ou en cliver certains groupes fonctionnels et des résidus d'autres biomolécules, selon un processus appelé modification post-traductionnelle. Les enzymes catalysent plus de 5 000 réactions chimiques différentes[18]. Comme tous les catalyseurs, elles ne modifient pas les équilibres chimiques mais accélèrent les réactions, parfois dans des proportions considérables ; ainsi, l'orotidine-5'-phosphate décarboxylase catalyse en quelques millisecondes une réaction qui prendrait sinon plusieurs millions d'années[19],[20].
Les molécules qui se lient aux enzymes et sont modifiées chimiques par elles sont appelées substrats. Bien que les enzymes soient parfois constituées de plusieurs centaines de résidus d'acides aminés, seuls quelques uns d'entre eux entrent en contact avec le ou les substrats de l'enzyme, et un très petit nombre — généralement trois ou quatre — sont impliqués directement dans la catalyse. On appelle site actif la région d'une enzyme impliquée dans la réaction chimique catalysée par cette protéine : elle regroupe les résidus qui se lient au substrat ou contribuent à son positionnement, ainsi que les résidus qui catalysent véritablement la réaction.
Signalisation cellulaire et liaison de ligands

De nombreuses protéines sont impliquées dans les mécanismes de signalisation cellulaire et de transduction de signal. Certaines protéines telles que l'insuline appartiennent au milieu extracellulaire et transmettent un signal de la cellule où elles sont synthétisées vers d'autre cellules parfois situées dans des tissus éloignés. D'autres sont des protéines membranaires qui agissent comme récepteurs dont la fonction principale est de se lier aux molécules porteuses de signaux et d'induire une réponse biochimique dans la cellule cible. De nombreux récepteurs membranaires ont un site de liaison exposé à l'extérieur de la cellule et un domaine effecteur en contact avec le milieu intracellulaire. Ce domaine effecteur peut être porteur d'une activité enzymatique ou peut subir des changements conformationnels agissant sur d'autres protéines intracellulaires.
Les anticorps sont les constituants protéiques du système immunitaire dont la fonction principale est de se lier aux antigènes ou aux xénobiotiques afin de les marquer pour élimination par l'organisme. Les anticorps peuvent être sécrétés dans le milieu extracellulaire ou bien ancrés dans la membrane plasmique de lymphocytes B spécialisés appelés plasmocytes. Là où les enzymes sont très spécifiques de leurs substrats afin d'accélérer des réactions chimiques très précises, les anticorps n'ont pas cette contrainte ; en revanche, leur affinité pour leur cible est extrêmement élevée.
De nombreuses protéines transporteuses de ligands se lient spécifiquement à de petites molécules et les transportent à destination à travers les cellules et les tissus des organismes multicellulaires. Ces protéines doivent posséder une forte affinité pour leur ligand lorsque la concentration de celui-ci est élevée, mais doivent également pouvoir le libérer lorsque sa concentration est faible dans les tissus cibles. L'exemple canonique de la protéine porteuse de ligand est l'hémoglobine, qui transporte l'oxygène des poumons vers les autres organes et tissus chez tous les vertébrés et a des homologues apparentés dans tous les règnes du vivant. Les lectines sont des protéines qui se lient réversiblement à certains glucides avec une très grande spécificité. Elles jouent un rôle dans les phénomènes de reconnaissance biologique impliquant cellules et protéines[21].
Les protéines transmembranaires peuvent également jouer le rôle de protéines transporteuses de ligands susceptibles de modifier la perméabilité de la membrane plasmique aux petites molécules polaires et aux ions. La membrane elle-même possède un cœur hydrophobe à travers lequel les molécules polaires ou électriquement chargées ne peuvent pas diffuser. Les protéines membranaires peuvent ainsi contenir un ou plusieurs canaux à travers la membrane cellulaire et permettant à ces molécules et à ces ions de la traverser. De nombreux canaux ioniques sont très spécifiques de l'ion dont ils permettent la circulation. Ainsi, les canaux potassiques et les canaux sodiques sont souvent spécifiques de l'un des deux ions potassium et sodium à l'exclusion de l'autre.
Protéines structurelles
Les protéines structurelles confèrent raideur et rigidité à des constituants biologiques qui, sans elles, seraient fluides. La plupart des protéines structurelles sont fibreuses. C'est par exemple le cas du collagène et de l'élastine qui sont des constituants essentiels de tissus conjonctifs tels que le cartilage, et de la kératine présente dans les structures dures ou filamenteuses telles que les poils, les ongles, les plumes, les sabots et l'exosquelette de certains animaux. Certaines protéines globulaires peuvent également jouer un rôle structurel, par exemple l'actine et la tubuline dont les monomères sont glubulaires et solubles mais polymérisent pour former de longs filaments rigides constituant le cytosquelette, ce qui permet à la cellule de maintenir sa forme et sa taille.
Les protéines motrices sont des protéines structurelles particulières qui sont capables de générer des forces mécaniques. Ce sont par exemple la myosine, la kinésine et la dynéine. Ces protéines sont essentielles à la motilité des organismes unicellulaires ainsi qu'aux spermatozoïdes des organismes multicellulaires. Elles permettent également de générer les forces à l'œuvre dans la contraction musculaire et jouent un rôle essentiel dans le transport intracellulaire.
Récapitulatif de fonctions assurées par les protéines
Les protéines remplissent ainsi des fonctions très diverses au sein de la cellule et de l'organisme[22]:
- les protéines structurelles, qui permettent à la cellule de maintenir son organisation dans l'espace, et qui sont les constituants du cytosquelette ;
- les protéines de transport, qui assurent le transfert des différentes molécules dans et en dehors des cellules ;
- les protéines régulatrices, qui modulent l'activité d'autres protéines ou qui contrôlent l'expression des gènes ;
- les protéines de signalisation, qui captent les signaux extérieurs, et assurent leur transmission dans la cellule ou l'organisme ; il en existe plusieurs sortes, par exemple les protéines hormonales, qui contribuent à coordonner les activités d'un organisme en agissant comme des signaux entre les cellules ;
- les protéines réceptrices, qui détectent les molécules messagères et les autres signaux pour que la cellule agisse en conséquence :
- les protéines sensorielles détectent les signaux environnementaux (ex. : lumière) et répondent en émettant des signaux dans la cellule ;
- les récepteurs d'hormone détectent les hormones et envoient des signaux à la cellule pour qu'elle agisse en conséquence (ex. : l'insuline est une hormone qui, lorsqu'elle est captée, signale à la cellule d'absorber et d'utiliser le glucose) ;
- les protéines motrices, permettant aux cellules ou organismes ou à certains éléments (cils) de se mouvoir ou se déformer (ex. : l'actine et la myosine permettent au muscle de se contracter) ;
- les protéines de défense, qui protègent la cellule contre les agents infectieux (ex. : les anticorps) ;
- les protéines de stockage, qui permettent la mise en réserve d'acides aminés pour pouvoir biosynthétiser d'autres protéines (ex. : l'ovalbumine, la principale protéine du blanc d'œuf permet leur stockage pour le développement des embryons de poulet) ;
- les enzymes, qui modifient la vitesse de presque toutes les réactions chimiques dans la cellule sans être transformées dans la réaction.
Méthodes d'étude
La structure et les fonctions des protéines peuvent être étudiées in vivo, in vitro et in silico. Les études in vivo permettent d'explorer le rôle physiologique d'une protéine au sein d'une cellule vivante ou même au sein d'un organisme dans son ensemble. Les études in vitro de protéines purifiées dans des environnements contrôlés sont utiles pour comprendre la façon dont une protéine fonctionne in vivo : par exemple, l'étude de la cinétique d'une enzyme permet d'analyser le mécanisme chimique de son activité catalytique et de son affinité relative vis-à-vis de différents substrats. Les études in silico utilisent des algorithmes informatiques pour modéliser des protéines.
Purification des protéines
Pour pouvoir être analysée in vitro, une protéine doit préalablement avoir été purifiée des autres constituants chimiques de la cellule. Ceci commence généralement par la lyse de la cellule, au cours de laquelle la membrane plasmique est rompue afin d'en libérer le contenu dans une solution pour donner un lysat. Ce mélange peut être purifié par ultracentrifugation, ce qui permet d'en séparer les constituants en fractions contenant respectivement les protéines solubles, les lipides et protéines membranaires, les organites cellulaires, et les acides nucléiques. La précipitation des protéines par relargage permet de les concentrer à partir de ce lysat. Il est alors possible d'utiliser plusieurs types de chromatographie pour isoler les protéines que l'on souhaite étudier en fonction de leurs propriétés physico-chimiques telles que leur masse molaire, leur charge électrique, ou encore leur affinité de liaison. Le degré de purification peut être suivi à l'aide de plusieurs types d'électrophorèse sur gel si la masse moléculaire et le point isoélectrique des protéines étudiées sont connus, par spectroscopie si la protéine présente des caractéristiques spectroscopiques identifiables, ou par dosage enzymatique (en) si la protéine est porteuse d'une activité enzymatique. Par ailleurs, les protéines peuvent être isolées en fonction de leur charge électrique par électrofocalisation (en)[23].
Les protéines naturelles requièrent éventuellement une série d'étapes de purification avant de pouvoir être étudiées en laboratoire. Afin de simplifier ce procédé, le génie génétique est souvent utilisé pour modifier les protéines en les dotant de caractéristiques qui les rendent plus faciles à purifier dans pour autant altérer leur structure ni leur activité. On ajoute ainsi des « étiquettes » reconnaissables sur les protéines sous forme de séquences d'acides aminés identifiées, souvent une série de résidus d'histidine — étiquette poly-histidine , ou His-tag — à l'extrémité C-terminale ou à l'extrémité N-terminale de la chaîne polypeptidique. De ce fait, lorsque le lysat est placé dans une colonne chromatographique contenant du nickel, les résidus d'histidine se complexent au nickel et restent liées à la colonne tandis que les constituants dépourvus d'étiquette la traversent sans être arrêtés. Plusieurs types d'étiquettes ont été développés afin de permettre aux chercheurs de purifier des protéines particulières à partir de mélanges complexes[24].
Localisation cellulaire
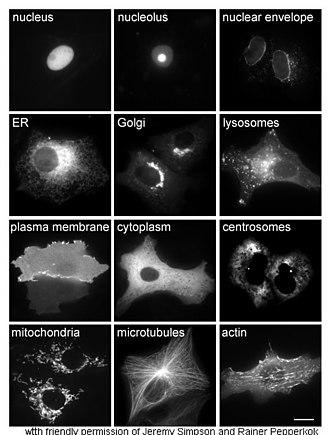
L'étude in vivo des protéines implique souvent de savoir précisement où elles sont synthétisées et où elles se trouvent dans les cellules. Bien que la plupart des protéines intracellulaires soient produites dans le cytoplasme et que la plupart des protéines membranaires ou sécrétées dans le milieux extracellulaire sont produites dans le réticulum endoplasmique, il est rare qu'on comprenne précisément comment les protéines ciblent spécifiquement certaines structures cellulaires ou certains organites. Le génie génétique offre des outils utiles pour se faire une idée de la localisation de certaines protéines, par exemple en liant la protéine étudiée à une protéine permettant de la repérer, c'est-à-dire en réalisant une protéine de fusion entre la protéine étudiée et une protéine utilisée comme marqueur, telle que la protéine fluorescente verte[25]. La localisation intracellulaire de la protéine de fusion résultante peut être facilement et efficacement visualisée par microscopie[26].
D'autres méthodes de localisation intracellulaire des protéines impliquent l'utilisation de marqueurs connus pour certains compartiments cellulaires tels que le réticulum endoplasmique, l'appareil de Golgi, les lysosomes, les mitochondries, les chloroplastes, la membrane plasmique, etc. Il est par exemple possible de localiser des protéines marquées avec une étiquette fluorescente ou ciblées avec des anticorps contre ces marqueurs. Les techniques d'immunofluorescence permettent ainsi de localiser des protéines spécifiques. Des pigments fluorescents sont également utilisés pour marquer des compartiments cellulaires dans un but similaire[27].
L'immunohistochimie utilise généralement un anticorps ciblant une ou plusieurs protéines étudiées qui sont conjugués à des enzymes émettant des signaux luminescents ou chromogènes pouvant être comparés à divers échantillons, ce qui permet d'en déduire des informations sur la localisation des protéines étudiées. Il est également possible d'utiliser des techniques de cofractionnement dans un gradient de saccharose (ou d'une autre substance) à l'aide d'une centrifugation isopycnique.
La microscopie immunoélectronique combine l'utilisation d'une microscopie électronique classique à l'utilisation d'un anticorps dirigé contre la protéine étudiée, cet anticorps étant préalablement conjugué à un matériau à forte densité électronique telle que l'or. Ceci permet de localiser des détails ultrastructurels ainsi que la protéine étudiée[28].
Étymologie

Les protéines furent découvertes à partir de 1835 aux Pays-Bas par le chimiste organicien Gerardus Johannes Mulder[29] (1802-1880), sous le nom de wortelstof. C'est son illustre confrère suédois, Jöns Jacob Berzelius, qui lui suggéra en 1838 le nom de protéine.
Le terme protéine vient du grec ancien prôtos qui signifie premier, essentiel. Ceci fait probablement référence au fait que les protéines sont indispensables à la vie et qu'elles constituent souvent la part majoritaire (≈ 60 %) du poids sec des cellules.
Une autre théorie, voudrait que protéine fasse référence, comme l'adjectif protéiforme, au dieu grec Protée qui pouvait changer de forme à volonté. Les protéines adoptent en effet de multiples formes et assurent de multiples fonctions. Mais ceci ne fut découvert que bien plus tard, au cours du XXe siècle.
Protéome
Le protéome constitue l'ensemble de toutes les protéines d'un organisme, d'un tissu ou d'une cellule. En effet, toutes les cellules de l'organisme possèdent le même génome (même ADN) mais en fonction de son rôle, chacune d'elles exprimera des protéines différentes (hémoglobine dans les hématies, insuline dans les cellules bêta du pancréas, certaines enzymes pour le foie, etc).
Début 2012, plus de 3000 génomes d'organismes vivants ont été séquencés et plus de 7000 sont en cours de séquençage.
Les génomes des organismes modèles principaux tels que la bactérie Escherichia coli, la levure Saccharomyces cerevisiae, la plante Arabidopsis thaliana et de nombreux autres génomes dont celui de l'homme[30] ayant été décryptés, il est admis que la quasi-totalité des gènes a pu être définie.
Par contre l'inventaire des protéines actives (ou protéome) dans un organisme est encore loin d'être établi. En effet, notamment en raison de la variabilité des processus d'activation et de régulation des protéines, cet inventaire n'est pas le résultat de la simple traduction de chaque gène qui donnerait une protéine active : par exemple certains gènes peuvent donner plusieurs formes différentes d'une protéine, ou encore de nombreuses protéines doivent être modifiées pour être actives.
Propriétés
Phénotype
Le plan de fabrication des protéines dépend donc en premier lieu du gène. Or les séquences des gènes ne sont pas strictement identiques d'un individu à l'autre. De plus, dans le cas des êtres vivants diploïdes, il existe deux exemplaires de chaque gène. Et ces deux exemplaires ne sont pas nécessairement identiques. Un gène existe donc en plusieurs versions d'un individu à l'autre et parfois chez un même individu. Ces différentes versions sont appelées allèles. L'ensemble des allèles d'un individu forme le génotype.
Puisque les gènes existent en plusieurs versions, les protéines vont également exister en différentes versions. Ces différentes versions de protéines vont provoquer des différences d'un individu à l'autre : tel individu aura les yeux bleus mais tel autre aura les yeux noirs, etc. Ces caractéristiques, visibles ou non, propres à chaque individu sont appelées le phénotype. Chez un même individu, un groupe de protéines à séquence similaire et fonction identique est dit isoforme. Les isoformes peuvent être le résultat de l'épissage alternatif d'un même gène, l'expression de plusieurs allèles d'un gène, ou encore la présence de plusieurs gènes homologues dans le génome.
Évolution
Au cours de l'évolution, les accumulations de mutations ont fait diverger les gènes au sein des espèces et entre espèces. De là provient la diversité des protéines qui leur sont associées. On peut toutefois définir des familles de protéines, elles-mêmes correspondant à des familles de gènes. Ainsi, dans une espèce peuvent coexister des gènes, et par conséquent des protéines, très similaires formant une famille. Deux espèces proches ont de fortes chances d'avoir des représentants de même famille de protéines.
On parle d'homologie entre protéines lorsque différentes protéines ont une origine commune, un gène ancestral commun.
La comparaison des séquences de protéines permet de mettre en évidence le degré de « parenté » entre différentes protéines, on parle ici de similarité de séquence. La fonction des protéines peut diverger au fur et à mesure que la similarité diminue, donnant ainsi naissance à des familles de protéines ayant une origine commune mais ayant des fonctions différentes.
L'analyse des séquences et des structures de protéine a permis de constater que beaucoup s'organisaient en domaines, c'est-à-dire en parties acquérant une structure et remplissant une fonction spécifique. L'existence de protéines à plusieurs domaines peut être le résultat de la recombinaison en un gène unique de plusieurs gènes originellement individuels, et réciproquement des protéines composés d'un unique domaine peuvent être le fruit de la séparation en plusieurs gènes d'un gène originellement codant une protéine à plusieurs domaines.
Alimentation humaine
Dans l'alimentation, les protéines sont désagrégées durant la digestion à partir de l'estomac. C'est là que les protéines sont hydrolysées par des protéases et coupées en polypeptides pour ensuite fournir des acides aminés pour l'organisme, y compris ceux, dits essentiels, que l'organisme n'est pas capable de synthétiser. Le pepsinogène est converti en pepsine quand il arrive au contact avec l'acide chlorhydrique. La pepsine est la seule enzyme protéolytique qui digère le collagène, la principale protéine du tissu conjonctif. La majeure partie de la digestion des protéines a lieu dans le duodénum.
Presque toutes les protéines sont absorbées quand elles arrivent dans le jéjunum et seulement 1 % des protéines ingérées se retrouvent dans les fèces. Certains acides aminés restent dans les cellules épithéliales et sont utilisés pour la biosynthèse de nouvelles protéines, y compris certaines protéines intestinales, constamment digérées, recyclées et absorbées par l'intestin grêle.
Quantités recommandées
En France, les recommandations nutritionnelles sont données par le PNNS et son site grand public (www.mangerbouger.fr) : « de la viande, du poisson ou des œufs une à deux fois par jour, toujours en quantité inférieure à l'accompagnement, soit de 100 à 150 g de viande maximum sur la journée »[31]. L'AFSSA recommande un apport nutritionnel conseillé (ANC) de 0,83 g.kg-1.j-1 chez l’adulte en bonne santé[32], soit 62 g par jour pour un homme de 75 kg. Il faut noter que les ANC sont supérieurs aux besoins moyens qui sont de 0,66 g.kg-1.j-1 selon ce même rapport, ce qui donnerait 49,5 g par jour pour le cas précédent.
Les besoins moyens en protéines ont été définis par la FAO qui recommande 49 g de protéines pour les hommes adultes et 41 pour les femmes (47 si enceintes, 58,5 si allaitantes)[33].
Protéines animales, protéines végétales
Pendant longtemps on a considéré que les protéines d'origine animale étaient supérieures en qualité aux protéines d'origine végétale. Actuellement ce jugement fait l'objet de révisions importantes. Les protéines animales sont invariablement accompagnées de lipides saturés dont la consommation est souvent excessive, ou bien d'additifs alimentaires comme les nitrites (dans les charcuteries) qui sont soupçonnées d'être cancérigènes. La production de viande consomme des ressources et contribue à l'émission de gaz à effet de serre. Les protéines animales, ou des produits associés comme les amines hétérocycliques seraient également un facteur de risque pour certains cancers (côlon, vessie)[34]. La consommation de viandes rouges serait associée à un risque accru de maladies cardio-vasculaires[35]. Parallèlement, des effets positifs sont associés aux végétaux riches en protéines. Les légumineuses à graines apportent un sentiment de satiété. Elles sont riches en fibres et en minéraux. Leur indice glycémique est faible. La consommation de haricots contribue à faire baisser le taux de cholestérol[36],[37] et également à l'abaissement du risque d'accident cardio-vasculaire[38] et de certains cancers (côlon, prostate, pancréas)[34]. On évolue vers une promotion des protéines végétales.
Ceci dit, une « bonne » protéine contient 9 acides aminés essentiels : l’isoleucine, la leucine, la lysine, la méthionine, la phénylalanine, la thréonine, le tryptophane, la valine et l’histidine.
Les protéines végétales contiennent relativement peu de certains de ces 9 acides aminés (sauf pour le quinoa) alors que les protéines d’origine animale en contiennent plus, en proportion. Le problème concerne essentiellement la méthionine et la lysine.
Selon certains, il ne faut donc pas faire l’impasse sur les protéines d’origine animale et il est recommandé de consommer les 2 types de protéines surtout quand on est âgé [39]. Selon Ellen Muehlhoff, experte en nutrition de la FAO favorable à la consommation de protéines d'origine animale: "Contrairement à la croyance populaire, la plupart des protéines que nous obtenons des aliments d'origine animale sont seulement légèrement supérieures à celles d'origine végétale."[40]
Compléments alimentaires
Les compléments alimentaires peuvent être enrichis en protéines, principalement pour les sportifs souhaitant développer leur volume musculaire, mais aussi pour les personnes qui souffrent de carences en protéines. Les protéines utilisées sont souvent des protéines du lactosérum (sous le nom de "whey"), et des acides aminés ramifiés désignés sous le nom de "BCAA".
Notes et références
- Gregory A. Petsko et Dagmar Ringe, Structure et fonction des protéines, Bruxelles, De Boek Université, (ISBN 978-2-8041-5888-0)
- (en) Alex Gutteridge et Janet M. Thornton, « Understanding nature's catalytic toolkit », Trends in Biochemical Sciences, vol. 30, no 11, , p. 622-629 (PMID 16214343, DOI 10.1016/j.tibs.2005.09.006, lire en ligne)
- (en) Harvey Lodish, Arnold Berk, Paul Matsudaira, Chris A. Kaiser, Monty Krieger, Matthew P. Scott, Lawrence Zipursky et James Darnell, Molecular Cell Biology, New York, W. H. Freeman & Company 5e édition, , 973 p. (ISBN 978-0-7167-4366-8)
- (en) Zhaohui Xu, Arthur L. Horwich et Paul B. Sigler, « The crystal structure of the asymmetric GroEL–GroES–(ADP)7 chaperonin complex », Nature, vol. 388, no 6644, , p. 741-750 (PMID 9285585, DOI 10.1038/41944, lire en ligne)
- (en) Lisa J Harris, Eileen Skaletsky et Alexander McPherson, « Crystallographic structure of an intact IgG1 monoclonal antibody », Journal of Molecular Biology, vol. 275, no 5, , p. 861-872 (PMID 9480774, DOI 10.1006/jmbi.1997.1508, lire en ligne)
- (en) W. Bolton et M. F. Perutz, « Three Dimensional Fourier Synthesis of Horse Deoxyhaemoglobin at 2.8 Å Resolution », Nature, vol. 228, no 5271, , p. 551-552 (PMID 5472471, DOI 10.1038/228551a0, Bibcode 1970Natur.228..551B, lire en ligne)
- (en) Edward N. Baker, Thomas L. Blundell, John F. Cutfield, Susan M. Cutfield, Eleanor J. Dodson, Guy G. Dodson, Dorothy M. Crowfoot Hodgkin, Roderick E. Hubbard, Neil W. Isaacs, Colin D. Reynolds, Kiwako Sakabe, Norioshi Sakabe et Numminate M. Vijayan, « The structure of 2Zn pig insulin crystals at 1.5 Å resolution », Philosophical Transactions B, vol. 319, no 1195, , p. 369-456 (PMID 2905485, DOI 10.1098/rstb.1988.0058, Bibcode 1988RSPTB.319..369B, lire en ligne)
- (en) Michael B. Berry et George N. Phillips Jr., « Crystal structures of Bacillus stearothermophilus adenylate kinase with bound Ap5A, Mg2+ Ap5A, and Mn2+ Ap5A reveal an intermediate lid position and six coordinate octahedral geometry for bound Mg2+ and Mn2+ », Proteins: Structure, Function, and Bioinformatics, vol. 32, no 3, , p. 276-288 (PMID 9715904, lire en ligne)
- (en) Harindarpal S. Gill et David Eisenberg, « The Crystal Structure of Phosphinothricin in the Active Site of Glutamine Synthetase Illuminates the Mechanism of Enzymatic Inhibition », Biochemistry, vol. 40, no 7, , p. 1903-1912 (PMID 11329256, DOI 10.1021/bi002438h, lire en ligne)
- (en) Alice B. Fulton et William B. Isaacs, « Titin, a huge, elastic sarcomeric protein with a probable role in morphogenesis », BioEssays, vol. 13, no 4, , p. 157-161 (PMID 1859393, DOI 10.1002/bies.950130403, lire en ligne)
- (en) « Titin (EC 2.7.11.1) (Connectin) Mus musculus (Mouse) », sur ProtParam sur ExPASy (consulté le )
- (en) Thomas Bruckdorfer, Oleg Marder et Fernando Albericio, « From Production of Peptides in Milligram Amounts for Research to Multi-Tons Quantities for Drugs of the Future », Current Pharmaceutical Biotechnology, vol. 5, no 1, , p. 29-43 (PMID 14965208, DOI 10.2174/1389201043489620, lire en ligne)
- (en) Dirk Schwarzer et Philip A Cole, « Protein semisynthesis and expressed protein ligation: chasing a protein's tail », Current Opinion in Chemical Biology, vol. 9, no 6, , p. 561-569 (PMID 16226484, DOI 10.1016/j.cbpa.2005.09.018, lire en ligne)
- (en) Stephen B. H. Kent, « Total chemical synthesis of proteins », Chemical Society Reviews, vol. 38, no 2, , p. 338-351 (PMID 19169452, DOI 10.1039/B700141J, lire en ligne)
- (en) R. Sankaranarayanan et D. Moras, « The fidelity of the translation of the genetic code », Acta Biochimica Polonica, vol. 48, no 2, , p. 323-335 (PMID 11732604, lire en ligne)
- (en) John A. Copland, Melinda Sheffield-Moore, Nina Koldzic-Zivanovic, Sean Gentry, George Lamprou, Fotini Tzortzatou-Stathopoulou, Vassilis Zoumpourlis, Randall J. Urban et Spiros A. Vlahopoulos, « Sex steroid receptors in skeletal differentiation and epithelial neoplasia: is tissue-specific intervention possible? », BioEssays, vol. 31, no 6, , p. 629-641 (PMID 19382224, DOI 10.1002/bies.200800138, lire en ligne)
- (en) Stanislav Samarin et Asma Nusrat, « Regulation of epithelial apical junctional complex by Rho family GTPases », Frontiers in Bioscience, vol. 14, , p. 1129-1142 (PMID 19273120, DOI 10.2741/3298, lire en ligne)
- (en) Ida Schomburg, Antje Chang, Sandra Placzek, Carola Söhngen, Michael Rother, Maren Lang, Cornelia Munaretto, Susanne Ulas, Michael Stelzer, Andreas Grote, Maurice Scheer et Dietmar Schomburg, « BRENDA in 2013: integrated reactions, kinetic data, enzyme function data, improved disease classification: new options and contents in BRENDA », Nucleic Acids Research, vol. 41, no D1, , D764-D772 (PMID 23203881, DOI 10.1093/nar/gks1049, lire en ligne)
- (en) A. Radzicka et R. Wolfenden, « A proficient enzyme », Science, vol. 267, no 5194, , p. 90-93 (PMID 7809611, DOI 10.1126/science.7809611, lire en ligne)
- (en) Brian P. Callahan et Brian G. Miller, « OMP decarboxylase—An enigma persists », Bioorganic Chemistry, vol. 35, no 6, , p. 465-469 (PMID 17889251, DOI 10.1016/j.bioorg.2007.07.004, lire en ligne)
- (en) Harold Rudiger, Hans-Christian Siebert, Dolores Solis, Jesus initial Jimenez-Barbero, Antonio Romero, Claus-Wilhelm von der Lieth, Teresa Diaz-Maurino et Hans-Joachim Gabius, « Medicinal Chemistry Based on the Sugar Code: Fundamentals of Lectinology and Experimental Strategies with Lectins as Targets », Current Medicinal Chemistry, vol. 7, no 4, , p. 389-416 (PMID 10702616, DOI 10.2174/0929867003375164, lire en ligne)
- Harvey Lodish, Arnold Berk, Paul Matsudaira, Chris A. Kaiser, Monty Krieger, Matthew P. Scott, S. Laurence Zipursky et James Darnell (trad. Pierre L. Masson et Chrystelle Sanlaville), Biologie moléculaire de la cellule [« Molecular Cell Biology »], Bruxelles, De Boeck Université, , 3e éd., 1096 p. [détail de l’édition] (ISBN 2-8041-4802-5)
- (en) Julie Hey, Anton Posch, Andrew Cohen, Ning Liu et Adrianna Harbers, « Fractionation of Complex Protein Mixtures by Liquid-Phase Isoelectric Focusing », Methods in Molecular Biology, vol. 424, , p. 225-239 (PMID 18369866, DOI 10.1007/978-1-60327-064-9_19, lire en ligne)
- (en) K. Terpe, « Overview of tag protein fusions: from molecular and biochemical fundamentals to commercial systems », Applied Microbiology and Biotechnology, vol. 60, no 5, , p. 523-533 (PMID 12536251, DOI 10.1007/s00253-002-1158-6, lire en ligne)
- (en) Olesya V. Stepanenko, Vladislav V. Verkhusha, Irina M. Kuznetsova, Vladimir N. Uversky and K. K. Turoverov, « Fluorescent Proteins as Biomarkers and Biosensors: Throwing Color Lights on Molecular and Cellular Processes », Current Protein & Peptide Science, vol. 9, no 4, , p. 338-369 (PMID 18691124, PMCID 2904242, DOI 10.2174/138920308785132668, lire en ligne)
- (en) Rafael Yuste, « Fluorescence microscopy today », Nature Methods, vol. 2, no 12, , p. 902-904 (PMID 16299474, DOI 10.1038/nmeth1205-902, lire en ligne)
- (en) William Margolin, « Green Fluorescent Protein as a Reporter for Macromolecular Localization in Bacterial Cells », Methods, vol. 20, no 1, , p. 62-72 (PMID 10610805, DOI 10.1006/meth.1999.0906, lire en ligne)
- (en) Terry M. Mayhew et John M. Lucocq, « Developments in cell biology for quantitative immunoelectron microscopy based on thin sections: a review », Histochemistry and Cell Biology, vol. 130, no 2, , p. 299-313 (PMID 18553098, PMCID 2491712, DOI 10.1007/s00418-008-0451-6, lire en ligne)
- Découvertes des protéines
- « Dossier92/Bio-informatique », Université de Genève. Le fondateur de Swiss-Prot estimait en octobre 2008 que les quelques 21 000 gènes décryptés du génome humain pouvaient produire 60 000 protéines différentes.
- La viande le poisson ou les oeufs c'est une à deux fois par jour
- Rapport AFSSA: "Apport en protéines : consommation, qualité, besoins et recommandations"
- « Guide de nutrition familiale » (consulté le )
- Adventist Health Study-1 - Cancer Findings: Some Highlights
- Association between total, processed, red and white meat consumption and all-cause, CVD and IHD mortality: a meta-analysis of cohort studies
- Fiche Haricot Sec sur le site Passeport Santé
- Non-soy legume consumption lowers cholesterol levels: a meta-analysis of randomized controlled trials
- Legume consumption and risk of coronary heart disease in US men and women: NHANES I Epidemiologic Follow-up Study
- « Besoins en protéines »
- Animal vs Vegetable Proteins: The Food Processing Debate
Bibliographie
- (fr) Gregory Petsko, Dagmar Ringe (trad. Chrystelle Sanlaville, Dominique Charmot-Bensimon), Structure et fonction des protéines, De Boeck, Bruxelles, 2009. (ISBN 978-2-8041-5888-0).
- (fr) Lubert Stryer, Jeremy Mark Berg, John L. Tymoczko (trad. Serge Weinman), Biochimie, Flammarion, « Médecine-Sciences », Paris, 2008, 6e éd. (ISBN 978-2-257-00003-3).
- (fr) Carl-Ivar Brändén, John Tooze (trad. Bernard Lubochinsky, préf. Joël Janin), Introduction à la structure des protéines, De Boeck Université, Bruxelles, 1996. (ISBN 978-2-8041-2109-9).
Voir aussi
Articles connexes
- Acide aminé
- Peptide
- Hétéroprotéine
- Métalloprotéine
- Polypeptide
- Protéines recombinantes
- Sous-famille de protéines
Disciplines et méthodologies
Classes et familles de protéine
- Aquaporine
- Protéine de Rieske
- Transporteur lysosomal d'acide aminé
- Protéine fibreuse
- Protéine globulaire
- Viciline
- facteurs de transcription
- Protéines Cdx
- Protéine d'échafaudage
- Protéine tau
- Thioprotéine
- Sélénoprotéine
Liens externes
- (en) Projet Predictor Un logiciel de calcul partage qui utilise la plateforme BOINC, pour étudier le repliement des protéines.
- (en) Serveur MRS Un serveur de banques de données biologiques, où l'identification d'une entrée dans la banque PDB permet de visualiser la structure à l'écran, en mode dynamique (voyez par exemple ce que produit une recherche dans la banque PDB des entrées correspondant à la trypsine).
- (en) Proteins@home Un projet à grande échelle pour étudier le repliement des protéines, auquel vous pouvez participer avec votre ordinateur.