« SARS-CoV-2 » : différence entre les versions
m Formulation |
Section "ressources scientifiques" |
||
| Ligne 72 : | Ligne 72 : | ||
}}.</ref>. Son [[génome]], stable<ref>https://www.lexpress.fr/actualite/societe/sante/coronavirus-chinois-les-raisons-pour-lesquelles-il-ne-faut-pas-paniquer_2116926.html</ref>, constitué d'un [[Acide ribonucléique|ARN]] [[Monocaténaire|simple-brin]] de {{unité|29903|[[nucléotide]]s}}, a été [[Séquence (acide nucléique)|séquencé]] pour la première fois le {{Date-|5|1|2020}} par une équipe de l'[[université Fudan]] de [[Shanghai]] (Chine)<ref name=":9" />{{,}}<ref name=":0">{{Lien web|titre=New-type coronavirus causes pneumonia in Wuhan: expert – Xinhua {{!}} English.news.cn|url=http://www.xinhuanet.com/english/2020-01/09/c_138690570.htm|consulté le=9 janvier 2020}}.</ref>{{,}}<ref name=":1">{{Lien web|titre=CoV2020|url=https://platform.gisaid.org/epi3/start/CoV2020|série=platform.gisaid.org|consulté le=12 janvier 2020}}.</ref>{{,}}<ref name=":32">{{Lien web|langue=en|auteur1=Wu, F. et al.|titre=Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome|url=https://www.ncbi.nlm.nih.gov/nuccore/MN908947|site=GenBank|périodique=|date=2020-01-23|consulté le=2020-01-23}}.</ref>. |
}}.</ref>. Son [[génome]], stable<ref>https://www.lexpress.fr/actualite/societe/sante/coronavirus-chinois-les-raisons-pour-lesquelles-il-ne-faut-pas-paniquer_2116926.html</ref>, constitué d'un [[Acide ribonucléique|ARN]] [[Monocaténaire|simple-brin]] de {{unité|29903|[[nucléotide]]s}}, a été [[Séquence (acide nucléique)|séquencé]] pour la première fois le {{Date-|5|1|2020}} par une équipe de l'[[université Fudan]] de [[Shanghai]] (Chine)<ref name=":9" />{{,}}<ref name=":0">{{Lien web|titre=New-type coronavirus causes pneumonia in Wuhan: expert – Xinhua {{!}} English.news.cn|url=http://www.xinhuanet.com/english/2020-01/09/c_138690570.htm|consulté le=9 janvier 2020}}.</ref>{{,}}<ref name=":1">{{Lien web|titre=CoV2020|url=https://platform.gisaid.org/epi3/start/CoV2020|série=platform.gisaid.org|consulté le=12 janvier 2020}}.</ref>{{,}}<ref name=":32">{{Lien web|langue=en|auteur1=Wu, F. et al.|titre=Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome|url=https://www.ncbi.nlm.nih.gov/nuccore/MN908947|site=GenBank|périodique=|date=2020-01-23|consulté le=2020-01-23}}.</ref>. |
||
Plusieurs des premiers cas ayant été mis en relation avec le [[Marché de gros de fruits de mer de Huanan|marché de gros en fruits de mer et autres animaux]], SARS-CoV-2 a été présumé d'origine [[Zoonose|zoonotique]] mais cela n'a pas été formellement confirmé à ce jour<ref>{{Lien web|langue=en|auteur1=|titre=Update and Interim Guidance on Outbreak of 2019 Novel Coronavirus (2019-nCoV) in Wuhan, China|url=https://www.nj.gov/health/cd/documents/topics/NCOV/NCoV_LINCS_wuhan_update_011820_combined.pdf|site=NJ.gov|périodique=|date=17/01/2020|consulté le=01/02/2020}}</ref>. Le génome de SARS-CoV-2 est semblable à 79,5 % avec celui de [[SARS-CoV]] et à 96 % avec celui de coronavirus de chauves-souris<ref name=":2" />, ce qui laisse supposer que l'origine du virus se trouve chez les [[Chiroptera|chauves-souris]]<ref name=":11" />{{,}}<ref name=":12" />. |
Plusieurs des premiers cas ayant été mis en relation avec le [[Marché de gros de fruits de mer de Huanan|marché de gros en fruits de mer et autres animaux]], SARS-CoV-2 a été présumé d'origine [[Zoonose|zoonotique]] mais cela n'a pas été formellement confirmé à ce jour<ref>{{Lien web|langue=en|auteur1=|titre=Update and Interim Guidance on Outbreak of 2019 Novel Coronavirus (2019-nCoV) in Wuhan, China|url=https://www.nj.gov/health/cd/documents/topics/NCOV/NCoV_LINCS_wuhan_update_011820_combined.pdf|site=NJ.gov|périodique=|date=17/01/2020|consulté le=01/02/2020}}</ref>. Le génome de SARS-CoV-2 est semblable à 79,5 % avec celui de [[SARS-CoV]] et à 96 % avec celui de coronavirus de chauves-souris<ref name=":2" />{{,}}<ref>{{Article |langue=en |prénom1=Peng |nom1=Zhou |prénom2=Xing-Lou |nom2=Yang |prénom3=Xian-Guang |nom3=Wang |prénom4=Ben |nom4=Hu |titre=A pneumonia outbreak associated with a new coronavirus of probable bat origin |périodique=Nature |date=2020-02-03 |issn=0028-0836 |issn2=1476-4687 |doi=10.1038/s41586-020-2012-7 |lire en ligne=http://www.nature.com/articles/s41586-020-2012-7 |consulté le=2020-03-06 }}</ref>, ce qui laisse supposer que, comme d'ailleurs dans le cas de SARS-CoV, l'origine du virus se trouve chez les [[Chiroptera|chauves-souris]]<ref name=":11" />{{,}}<ref name=":12" />. |
||
| ⚫ | L'archive de [[Prépublication (édition scientifique)|prépublications]] [[bioRxiv]], ainsi que divers forums de chercheurs, permettent une diffusion rapide {{Incise |mais non formellement [[Évaluation par les pairs|validée par les pairs]]}} de l'information scientifique concernant SARS-CoV-2<ref>{{Lien web|langue=en|auteur1=|titre=2019-nCoV in bioRxiv|url=https://www.biorxiv.org/search/%25222019-ncov%2522%20jcode%3Abiorxiv%7C%7Cmedrxiv%20numresults%3A50%20sort%3Apublication-date%20direction%3Adescending%20format_result%3Acondensed|site=bioRxiv|périodique=|date=|consulté le=27/01/2020}}</ref>. |
||
Il ne faut pas confondre SARS-CoV-2 et [[COVID-19]]. Le premier est le nom du virus, le deuxième est le nom (féminin) de la maladie que provoque le virus. |
Il ne faut pas confondre SARS-CoV-2 et [[COVID-19]]. Le premier est le nom du virus, le deuxième est le nom (féminin) de la maladie que provoque le virus. |
||
== Ressources scientifiques == |
|||
| ⚫ | L'archive de [[Prépublication (édition scientifique)|prépublications]] [[bioRxiv]], ainsi que divers forums de chercheurs, permettent une diffusion rapide {{Incise |mais non formellement [[Évaluation par les pairs|validée par les pairs]]}} de l'information scientifique concernant SARS-CoV-2<ref>{{Lien web|langue=en|auteur1=|titre=2019-nCoV in bioRxiv|url=https://www.biorxiv.org/search/%25222019-ncov%2522%20jcode%3Abiorxiv%7C%7Cmedrxiv%20numresults%3A50%20sort%3Apublication-date%20direction%3Adescending%20format_result%3Acondensed|site=bioRxiv|périodique=|date=|consulté le=27/01/2020}}</ref>. |
||
Les premières [[Publication scientifique|publications scientifiques]] validées par les pairs sont publiées le 24 janvier 2020<ref name=":4" />{{,}}<ref name=":5" />. Plusieurs grandes revues et grands éditeurs scientifiques, devant l'urgence de la situation, ont décidé de rendre disponibles hors abonnement un certain nombre de publications scientifiques sur le nouveau coronavirus et la pneumonie associée: [[Elsevier (éditeur)|Elsevier]]<ref>{{Lien web|langue=en|nom1=Elsevier|titre=Novel Coronavirus Information Center|url=https://www.elsevier.com/connect/coronavirus-information-center|site=Elsevier Connect|consulté le=2020-03-06}}</ref>, [[The Lancet]]<ref>{{Lien web|langue=en|titre=COVID-19 Resource Centre|url=https://www.thelancet.com/coronavirus|site=www.thelancet.com|consulté le=2020-03-06}}</ref>, [[The New England Journal of Medicine]]<ref>{{Lien web|langue=en|titre=Coronavirus (Covid-19) — NEJM|url=https://www.nejm.org/coronavirus|site=New England Journal of Medicine|consulté le=2020-03-06}}</ref>, [[Science (revue)|Science]]<ref>{{Lien web|langue=en|titre=Coronavirus|url=https://www.sciencemag.org/tags/coronavirus|site=Science {{!}} AAAS|consulté le=2020-03-06}}</ref>, [[Springer Publishing|Springer]]-[[Nature (revue)|Nature]]<ref>{{Lien web|titre=Coronavirus {{!}} Springer Nature {{!}} For Researchers {{!}} Springer Nature|url=https://www.springernature.com/gp/researchers/campaigns/coronavirus|site=www.springernature.com|consulté le=2020-03-06}}</ref>, [[John Wiley & Sons|Wiley]]<ref>{{Lien web|langue=en|titre=Novel Coronavirus - Wiley Online Library|url=https://novel-coronavirus.onlinelibrary.wiley.com/|site=Novel Coronavirus|consulté le=2020-03-06}}</ref>. |
|||
== Nom du virus et de la maladie == |
== Nom du virus et de la maladie == |
||
Version du 6 mars 2020 à 23:03
2019-nCoV
| Domaine | Riboviria |
|---|---|
| Ordre | Nidovirales |
| Sous-ordre | Cornidovirineae |
| Famille | Coronaviridae |
| Sous-famille | Orthocoronavirinae |
| Genre | Betacoronavirus |
| Sous-genre | Sarbecovirus |
Modèle:Arbre début
- Modèle:Arbre/Branche finale Espèce : Coronavirus associé au SRAS
- Coronavirus du SRAS (SARS-CoV)
- Modèle:Arbre/Branche finale Coronavirus du SRAS 2 (SARS-CoV-2, proposition ICTV[2] en prépublication)
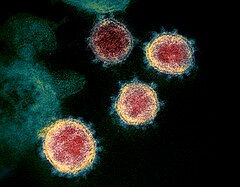
Le coronavirus du syndrome respiratoire aigu sévère 2, en abrégé SARS-CoV-2, est le coronavirus à l'origine de l’épidémie de maladie à coronavirus de 2019-2020 de la forme de pneumonie dénommée maladie à coronavirus 2019 (COVID-19), déclarée urgence de santé publique de portée internationale par l'Organisation mondiale de la santé (OMS) le [3]. Il a d'abord été désigné provisoirement par l'OMS[4] et les CDC[5] sous le terme anglais 2019 novel coronavirus[a], ou 2019-nCoV, avant d'être nommé SARS-CoV-2 sur recommandation de l'ICTV[2]. Il a été signalé pour la première fois dans la ville de Wuhan (province de Hubei, république populaire de Chine).
Apparenté au coronavirus du SRAS, il appartient au sous-genre Sarbecovirus[6]. Son génome, stable[7], constitué d'un ARN simple-brin de 29 903 nucléotides, a été séquencé pour la première fois le par une équipe de l'université Fudan de Shanghai (Chine)[8],[9],[10],[11].
Plusieurs des premiers cas ayant été mis en relation avec le marché de gros en fruits de mer et autres animaux, SARS-CoV-2 a été présumé d'origine zoonotique mais cela n'a pas été formellement confirmé à ce jour[12]. Le génome de SARS-CoV-2 est semblable à 79,5 % avec celui de SARS-CoV et à 96 % avec celui de coronavirus de chauves-souris[13],[14], ce qui laisse supposer que, comme d'ailleurs dans le cas de SARS-CoV, l'origine du virus se trouve chez les chauves-souris[15],[16].
Il ne faut pas confondre SARS-CoV-2 et COVID-19. Le premier est le nom du virus, le deuxième est le nom (féminin) de la maladie que provoque le virus.
Ressources scientifiques
L'archive de prépublications bioRxiv, ainsi que divers forums de chercheurs, permettent une diffusion rapide — mais non formellement validée par les pairs — de l'information scientifique concernant SARS-CoV-2[17].
Les premières publications scientifiques validées par les pairs sont publiées le 24 janvier 2020[18],[19]. Plusieurs grandes revues et grands éditeurs scientifiques, devant l'urgence de la situation, ont décidé de rendre disponibles hors abonnement un certain nombre de publications scientifiques sur le nouveau coronavirus et la pneumonie associée: Elsevier[20], The Lancet[21], The New England Journal of Medicine[22], Science[23], Springer-Nature[24], Wiley[25].
Nom du virus et de la maladie
Au début de l'épidémie, le virus a souvent été nommé « le coronavirus », « le nouveau coronavirus », « le coronavirus de Wuhan », ou encore « le coronavirus du marché aux fruits de mer de Wuhan ». L'Organisation mondiale de la santé (OMS) avait recommandé dans un premier temps l'appellation « 2019-nCoV » (sous-titre du présent article) tant qu'un nom définitif n'avait pas été fixé par l'International Committee on Taxonomy of Viruses (ICTV) conformément à ses recommandations générales en cas d'émergence épidémiologique[26].
Le , le groupe d'étude Nidovirales de l'ICTV a proposé le nom SARS-CoV-2 au terme d'une analyse taxonomique poussée[2]. Ce travail montre notamment que le nouveau coronavirus appartient à la même espèce biologique que SARS-CoV qui avait causé l'épidémie de SRAS en 2003, même si le syndrome observé en 2019 diffère de celui-ci. L'OMS donne à la pneumonie de Wuhan le nom officiel de COVID-19[27], pour "coronavirus disease 2019" (acronyme à utiliser au féminin pour "la" maladie: "la COVID-19").
Même si nommer différemment la maladie et l'agent qui la cause est habituel (ex. HIV cause le sida), et si la définition des espèces biologiques dépend d'autres éléments que ceux purement conjoncturels (ex. Botrytis cinerea cause la pourriture grise mais aussi la pourriture noble recherchée pour la vinification des sauternes), l'apparition des deux noms le même jour a d'abord suscité quelques incompréhensions tant chez un public peu averti mais sensibilisé par une situation de crise[28], que dans la communauté scientifique[29],[30].
Symptômes chez l'humain

Les maladies que provoquent les coronavirus peuvent aller du rhume (ils en sont la seconde cause aux États-Unis[31]) à des maladies plus graves telles que le syndrome respiratoire aigu sévère (SRAS, épidémie en 2003) et le syndrome respiratoire du Moyen-Orient (MERS-CoV, épidémie en 2012).
Une première description scientifiquement éprouvée de la pathologie et de l'épidémiologie de SARS-CoV-2 a été publiée le 24 janvier 2020 par un consortium de médecins de Wuhan et de Pékin, concernant la petite cohorte des 41 premiers cas étudiés à Wuhan pour décembre 2019[19]. L'âge médian des patients était de 49 ans et aucun d'eux n'avait moins de 18 ans. Le délai médian entre l'infection et l'admission à l'hôpital était de 7 jours (4-8 jours) ; la dyspnée s'est déclarée à 8 jours (5-13 jours) pour 51 % d'entre eux, et s'est aggravée à 9 jours (8-14 jours) pour 27 % d'entre eux ; l'admission en unité de soins intensifs est devenue nécessaire à 10,5 jours (8-17 jours) pour 39 % d'entre eux. Quelques semaines plus tard, 28 (68 %) des 41 patients étaient sortis de l'hôpital tandis que 6 (15 %) étaient décédés.
Les symptômes rapportés qui ressemblent grossièrement à ceux de la grippe saisonnière incluent la fièvre, la fatigue, une toux sèche, un manque de souffle, des difficultés à respirer[32],[33], la pneumonie, une insuffisance rénale, et le décès dans les cas sévères[34],[35]. Un calcul de la létalité basé sur les chiffres fournis par les différents pays à l'OMS au sujet des infections avérées et des décès, produit un taux d'environ 2 % en février 2020 (426 décès sur 20 588 infections)[36], une létalité qui semble faible en comparaison de celle d'autres coronavirus comme le SARS-CoV ou le MERS-COV (9,8 % et 34 % respectivement[37]), mais beaucoup plus élevée par exemple que la plupart des virus de la grippe touchant les humains. Néanmoins, le taux de létalité demeure incertain, du fait de la difficulté à estimer sur le terrain le nombre de cas et le nombre de décès attribuables à SARS-CoV-2[38],[39],[40].
La majorité des patients hospitalisés présentent des signes vitaux stables lors de leur admission, une lymphopénie (manque de lymphocytes[32]. Toutefois, un quart des patients diagnostiqués avaient des symptômes sévères. Une étude sur les 41 premiers patients hospitalisés avec un diagnostic confirmé de maladie à coronavirus 2019 a relevé qu'une majorité de ceux-ci étaient en bonne santé avant leur infection, un tiers ayant été concerné par des problèmes de santé préalables. Les trois quarts des patients toussaient, un peu plus de la moitié ressentait une gêne respiratoire, et un tiers avait des difficultés respiratoires sévères nécessitant des soins intensifs[41],[19]. Le 23 janvier 2020, l'OMS déclare que la plupart des patients décédés avaient un système immunitaire affaibli dû à des problèmes de santé tels que l'hypertension, le diabète ou une maladie cardiovasculaire[42].
La période d'incubation de SARS-CoV-2 est estimée entre 2 et 14 jours[43], mais la période d'incubation serait plus longue dans certains cas, jusqu'à 24 jours[44].
Taxonomie et phylogénie (origine du virus)

Le virus SARS-CoV-2 appartient à l'espèce SARS-CoV, dans le genre Betacoronavirus et la famille Coronaviridae[2]. La morphologie des virions est typique de celle des coronavirus[33], avec notamment le halo de protubérances protéiques ("Spike") qui leur a donné leur nom ("virus à couronne").
Le nombre de génomes isolés et séquencés croît rapidement ainsi que leurs origines géographiques : ils sont 179 au [45] (les premiers génomes séquencés, originaires de Wuhan, l'ont été par le CDC chinois, l'Institut de biologie des agents pathogènes et l'hôpital Wuhan Jinyintan[46],[47]). Cette dynamique a rapidement permis de définir que SARS-CoV-2 présente des similitudes avec les Betacoronavirus trouvés chez les chauves-souris[13],[15]. Il forme une souche virale génétiquement distincte des autres coronavirus humains MERS[13] ou à d'autres espèces plus bénignes, mais appartenant à la même espèce biologique que le SARS-CoV[2], dans le sous-genre Sarbecovirus[48],[49],[50].
Une analyse phylogénétique et génomique publiée le excluait quasiment une descendance directe de SARS-CoV-2 à partir des souches classiques de SARS-CoV[51] : selon ses auteurs un événement de recombinaison homologue pourrait avoir "mélangé" un Betacoronavirus de « clade A » (virus de type Bat SARS CoVZC45 et CoVZXC21) avec un bétacoronavirus encore inconnu. Cette étude suggère par ailleurs, en examinant la fréquence d'usage des codons, qu'un serpent de la région de Wuhan serait le « réservoir d'animaux sauvages le plus probable » pour SARS-CoV-2 (bien qu'aucun coronavirus ne soit connu dans les animaux à sang froid), ce qui a été repris par la presse[52] ; cette dernière interprétation a toutefois été remise en cause le jour même, et l'origine « serpent » formellement écartée[53],[16]. L'hypothèse d'une recombinaison a quant à elle aussi été écartée quelques jours plus tard par une équipe grecque[54].
Le marché de gros de fruits de mer de Huanan, à Wuhan, a été identifié comme la source initiale de l'épidémie dans la première annonce officielle par les autorités locales, le 31 décembre 2019[55]. Le marché a été fermé au lendemain de l'alerte, le 1er janvier 2020. Les premiers symptômes attribuables a posteriori à SARS-CoV-2 sont toutefois apparus dès le 1er décembre 2019 chez un patient qui n'avait pas fréquenté ce marché[19], de même qu'un tiers des 41 cas signalés à Wuhan en décembre 2019 : cela suggère à certains experts que l'origine de l'épidémie pourrait être antérieure à décembre 2019, et ne pas se trouver sur le marché de Wuhan[56],[57].
En outre, s'il est clair que ce marché vendait une grande diversité de faune sauvage[58], les chauves-souris n'avaient pas l'air d'en faire partie et leur consommation n'est d'ailleurs pas traditionnelle dans la région, malgré ce que prétendent des vidéos devenues virales sur Internet[59],[60] ; si SARS-CoV-2 était bel et bien issu d'un virus de chauve-souris, d'autres événements que la consommation de viande de cet animal peuvent avoir été la source de contamination, comme cela semble avoir été le cas pour une autre épidémie virale[61]. Il est également possible que, de la même façon que le SARS-CoV semble être passé d'une chauve-souris à l'homme par la civette qui a joué le rôle d'hôte intermédiaire, ou encore le MERS-CoV d'une chauve-souris à l'homme via le dromadaire comme hôte intermédiaire, SARS-CoV-2 soit passé par un hôte intermédiaire qui reste à identifier ; certains experts avancent l'hypothèse du Pangolin, un mammifère en danger qui fait l'objet d'une consommation en Chine et qui était en vente sur le marché de Wuhan[62],[63] ; cette hypothèse reste discutée[64], voire formellement contestée[65].
Sur la base des séquences complètes disponibles, l'initiative GISAID commence dès fin janvier 2020 à distinguer une histoire évolutive de SARS-CoV-2[66] : des mutations groupées dans le temps et dans l'espace de l'épidémie, ainsi que le long du génome.
Épidémiologie (dispersion du virus)
Dans les premières semaines de l'épidémie de 2019-2020, le virus s'est propagé par l'intermédiaire de voyageurs vers différents pays d'Asie[67],[68],[69], le 21 janvier en Amérique du Nord[70], le 24 janvier, en Australie[71], en Europe[72],[73], le 16 février en Afrique[74] et le 26 février en Amérique du Sud[75].
À la date du 6 mars 2020 on compte 3 408 décès et 55 694 guérisons pour 100 330 cas confirmés[76] (le cap des 100 000 cas est franchi le 6 mars), le tout principalement dans la province du Hubei. Ces chiffres constituent probablement une large sous-estimation de l'ampleur de l'épidémie[77],[78], la maladie restant souvent discrète notamment chez les sujets jeunes.
Le , la transmission interhumaine a été confirmée dans le Guangdong, en Chine, selon Zhong Nanshan, chef de l'équipe de la commission de la santé enquêtant sur l'épidémie[79]. La première description scientifiquement détaillée d'une transmission interhumaine au sein d'une famille a été publiée par une équipe de médecins de Hong Kong et de Shenzhen le 24 janvier 2020[18]. Le taux de reproduction (Ro) préliminaire de SARS-CoV-2, c’est-à-dire le nombre moyen de personnes infectées par un patient, a été estimé entre 1,4 et 2,5 par l'OMS le 23 janvier 2020[80]. Il serait comparable à celui de SARS-CoV et de la grippe de 1918, compris entre 1,4 et 3,8[81].
La durée de vie du virus hors organisme est quant à elle estimée entre 3 heures (en milieu sec) et 3 jours (en milieu humide), excluant la possibilité de contamination par colis, qui dans les cas les plus rapides, parcourent la distance Chine–États-Unis en 4 jours[82].
Situation épidémique
En décembre 2019 et janvier 2020, l'épidémie reste principalement localisée en Chine et notamment dans la province du Hunei. Le premier cas en dehors de la Chine continentale est découvert le 13 janvier: un cas importé en Thailande[83]. La situation évoluant rapidement au niveau mondial, elle est déclarée urgence de santé publique de portée internationale par l'OMS le 30 janvier 2020[3]. C'est la sixième fois seulement que l'OMS déclenchait ce niveau d'alerte dans son histoire.
Mi-février 2020, le plus important foyer épidémique hors de Chine était le paquebot de croisière Diamond Princess, en quarantaine dans le port de Yokohama[84]. Dans la troisième semaine de février toutefois, des foyers secondaires d'importance ont émergé en Corée du Sud, en Italie et en Iran[85], faisant craindre une installation durable de l'épidémie au niveau mondial[86].
Un décès est confirmé pour la première fois le 1er février hors de la Chine, aux Philippines : il s'agit d'un touriste venant de Wuhan où il a probablement été contaminé[87]. Le premier décès hors d'Asie se produit en France le 15 février[88]: il s'agit là encore d'un cas importé de Chine. Le 6 mars, l'Italie devenait le second pays en nombre de décès après la Chine[76].
Biologie moléculaire (fonctionnement du virus)
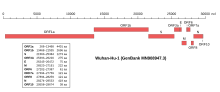

Le premier génome entièrement séquencé est celui de la souche Wuhan-Hu-1, diffusé le 23 janvier 2020[8]. L'organisation de l'ARN génomique, qui comprend 29903 nucléotides, est typique de celle d'un betacoronavirus. Un segment de la protéine « Spike » semble caractéristique de SARS-CoV-2, ainsi que peut-être un court gène supplémentaire, dont la réalité biologique et le rôle éventuel restent toutefois à démontrer[89].
La connaissance du nouveau génome a conduit à plusieurs expériences de modélisation de la protéine Spike (S), présumée jouer un rôle dans la liaison du virus au récepteur cellulaire. Au 23 janvier 2020, deux groupes chinois pensent que la protéine S conserve une affinité suffisante avec le récepteur du SARS-CoV (enzyme de conversion de l'angiotensine 2, ACE2) pour l'utiliser comme mécanisme d'entrée cellulaire[90]. L'identification de ACE2 comme récepteur cellulaire est également décrite indépendamment par trois autres groupes de chercheurs[13],[91],[92]. Le récepteur cellulaire de MERS-CoV, DPP4, ne semble pas reconnaître la protéine Spike de SARS-CoV-2[92], en accord avec la plus grande proximité moléculaire de SARS-CoV-2 avec SARS-CoV qu'avec MERS-CoV.
Lors de l'entrée du virus dans la cellule, la protéine Spike est maturée par la protéase cellulaire à sérine TMPRSS2, là encore comme c'est le cas pour SARS-CoV: cette découverte donne des pistes pour l'élaboration de traitements antiviraux[92].
Le virus provoquerait en particulier un excès de réponse inflammatoire du système immunitaire inné[93],[94].
Traitement et vaccins
L'essentiel de la lutte contre la COVID-19 repose sur des mesures épidémiologiques: diagnostic fiable et rapide de la maladie en période d'incubation, mesures de confinement (qui peuvent aussi être un facteur de diffusion intragroupe[95]).
Traitements antiviraux
Le SARS-CoV-2 étant de nature virale, les antibiotiques et les bactériophagiques sont inadaptés, étant donné qu'ils ne traitent que les pathologies bactériennes. Toutefois, les bactéries étant plus susceptibles d'adhérer aux cellules infectées par un virus tel le coronavirus[96], on observe une surinfection bactérienne, en particulier par le Streptococcus pneumoniae dans 10 % des cas d'infection virale[33]. Dans cette situation, la prescription d'un antibiotique adapté à la sensibilité de la bactérie se justifie donc.
Aucun traitement antiviral spécifique n'est actuellement disponible[97], mais des antiviraux existants pourraient être repositionnés[98], y compris des inhibiteurs de protéase comme l'indinavir, le saquinavir et le lopinavir / ritonavir[99], ou encore le Nelfinavir[100]. L'efficacité de l'inhibiteur de polymérase sofosbuvir, utilisé contre l'hépatite C, est également en cours d'évaluation en janvier 2020[101]. Le 2 février, des médecins thaïlandais ont annoncé avoir traité un patient avec succès en utilisant une combinaison des anti-HIV lopinavir et ritonavir ainsi que l'anti-grippal oseltamivir[102],[103]. Les traitements reçus par les patients concernent donc essentiellement l'atténuation des symptômes de fièvre, toux et dyspnée, afin de favoriser leur guérison spontanée[32],[76],[104]. Le Centre chinois de contrôle et de prévention des maladies a démarré dès janvier l'évaluation de l'efficacité de traitements existants contre les pneumonies[105], l'efficacité ayant été montrée in vitro[106].
Un repositionnement constituant une première piste est celui d'une molécule initialement antipaludique, la chloroquine, dont l'effet anticoronaviral avait déjà été démontré sur le SRAS[107]. Le 19 février, une lettre parue dans la revue BioScience Trends indique que des études cliniques chinoises ont montré une efficacité apparente du phosphate de chloroquine dans le traitement du virus[108]. Ces données sont cependant encore limitées notamment en raison de la petite taille de l'échantillon statistique (100 patients)[109]. Un consensus d'experts chinois recommande le 20 février d'inclure le phosphate de chloroquine dans les recommandations de prise en charge des patients sans contre-indication à la chloroquine, à raison de 500 mg deux fois par jour pendant 10 jours pour les patients diagnostiqués comme des cas légers, modérés et sévères de maladie[109],[110]. Il est cependant à noter que la prise de chloroquine à haute dose peut provoquer des effets secondaires potentiellement sérieux, en particulier celui d'une intoxication aiguë susceptible de déclencher des problèmes cardiaques ou respiratoires, et que la dose recommandée par les chercheurs chinois se rapproche de cette zone de risque[109]. Le 25 février, le professeur Didier Raoult recommande l'utilisation de la chloroquine pour les patients porteurs du virus[111], prédisant même une « fin de partie » pour le virus[112]. Sa déclaration est vivement critiquée par des médecins et scientifiques français sur les réseaux sociaux pour son manque de pondération[109] vis-à-vis de la limitation des données cliniques et des risques liés à la prise de chloroquine. L'Inserm tempère également et rappelle qu'aucune prise de médicament quel qu'il soit n'est anodine, et qu'il ne faut pas se faire prescrire la molécule pour l’utiliser en automédication[113],[114].
Vaccins à l'étude
À plus long terme, plusieurs équipes internationales travaillent à la mise au point ou à l'adaptation d'un vaccin[115],[116]. Même si l'épidémie de SRAS a finalement été maitrisée sans vaccin[117], la proximité moléculaire de SARS-CoV-2 avec SARS-CoV est un atout puisqu'au-delà d'indéniables différences entre ces deux virus[118], elle permet de capitaliser sur les connaissances acquises alors[119] (faisant au passage regretter que les recherches sur la mise au point d'un vaccin anti-SARS-CoV aient été interrompues lorsque ce premier virus a été considéré éradiqué).
Début février 2020, une équipe de recherche de l'Imperial College de Londres a fait une percée significative en réduisant une partie du temps de développement normal du vaccin « de deux à trois ans à seulement quatorze jours »[120],[121]. L'équipe de l'Imperial College est désormais au stade de test du vaccin sur les animaux[120]. En , un vaccin est en cours d'élaboration en Russie[122],[123], et un autre est préparé par la firme pharmaceutique Johnson & Johnson, mais ne serait pas disponible avant un an[124].
À la même période, en France, dans le cadre d'une recherche vaccinale concernant le MERS-CoV en cours depuis 2012, l'institut Pasteur pourrait utiliser et adapter ces vaccins pour SARS-CoV-2. Plusieurs équipes de l'Institut Pasteur travaillent sur son adaptation et seraient en mesure de produire ce vaccin en cas d'épidémie[125].
L'institut Robert-Koch préconise la vaccination contre le pneumocoque des personnes âgées, en particulier autour des foyers d'infection[126].
Le développement d'anticorps monoclonaux spécifiques est une autre piste[127],[128] de traitement qui a par ailleurs déjà montré son efficacité dans le cas du MERS-CoV[129],[130]. Ceci, d'autant que les anticorps produits naturellement n’empêcheraient pas les infections récidivantes chez certains patients[131].
Notes et références
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Severe acute respiratory syndrome coronavirus 2 » (voir la liste des auteurs).
- La désignation novel coronavirus est une dénomination provisoire standard pour tous les coronavirus nouvellement identifiés en attente de classification taxonomique.
- (en) « Virus Taxonomy: 2018b Release », ICTV, (consulté le ).
- (en) Alexander E. Gorbalenya, Susan C. Baker, Ralph S. Baric, Raoul J. de Groot, Christian Drosten, Anastasia A. Gulyaeva, Bart L. Haagmans, Chris Lauber, Andrey M Leontovich, Benjamin W. Neuman, Dmitry Penzar, Stanley Perlman, Leo L.M. Poon, Dmitry Samborskiy, Igor A. Sidorov, Isabel Sola et John Ziebuhr, « Severe acute respiratory syndrome-related coronavirus – The species and its viruses, a statement of the Coronavirus Study Group », bioRxiv, (DOI 10.1101/2020.02.07.937862, lire en ligne)
- « Déclaration sur la deuxième réunion du Comité d’urgence du Règlement sanitaire international (2005) concernant la flambée de nouveau coronavirus 2019 (2019-nCoV) tenue le 30 janvier 2020 », sur www.who.int (consulté le )
- (en) « Novel Coronavirus (2019-nCoV) », sur https://www.who.int/, OMS (consulté le ).
- (en) « 2019 Novel Coronavirus (2019-nCoV), Wuhan, China », sur https://www.cdc.gov/, CDC (consulté le ).
- (en) « Phylogeny of SARS-like betacoronaviruses including novel coronavirus from Wuhan using data generated by the Shanghai Public Health Clinical Center & School of Public Health, the National Institute for Viral Disease Control and Prevention, the Institute of Pathogen Biology, and the Wuhan Institute of Virology shared via GISAID », sur https://nextstrain.org/, (consulté le ).
- https://www.lexpress.fr/actualite/societe/sante/coronavirus-chinois-les-raisons-pour-lesquelles-il-ne-faut-pas-paniquer_2116926.html
- (en) « Wuhan seafood market pneumonia virus », sur NCBI (consulté le ).
- « New-type coronavirus causes pneumonia in Wuhan: expert – Xinhua | English.news.cn » (consulté le ).
- « CoV2020 », platform.gisaid.org (consulté le ).
- (en) Wu, F. et al., « Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome », sur GenBank, (consulté le ).
- (en) « Update and Interim Guidance on Outbreak of 2019 Novel Coronavirus (2019-nCoV) in Wuhan, China », sur NJ.gov, (consulté le )
- (en) Shi, ZL et al., « Discovery of a novel coronavirus associated with the recent pneumonia outbreak in humans and its potential bat origin », bioRxiv, (lire en ligne)
- (en) Peng Zhou, Xing-Lou Yang, Xian-Guang Wang et Ben Hu, « A pneumonia outbreak associated with a new coronavirus of probable bat origin », Nature, (ISSN 0028-0836 et 1476-4687, DOI 10.1038/s41586-020-2012-7, lire en ligne, consulté le )
- (en) Benvenuto D. et al., « The 2019-new Coronavirus epidemic: evidence for virus evolution », bioRxiv.org, (lire en ligne)
- (en) Callaway E. et Cyranoski D., « Why snakes probably aren’t spreading the new China virus », Nature, (ISSN 0028-0836 et 1476-4687, DOI 10.1038/d41586-020-00180-8, lire en ligne, consulté le )
- (en) « 2019-nCoV in bioRxiv », sur bioRxiv (consulté le )
- (en) Jasper Fuk-Woo Chan, Shuofeng Yuan, Kin-Hang Kok et Kelvin Kai-Wang To, « A familial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission: a study of a family cluster », The Lancet, , S0140673620301549 (DOI 10.1016/S0140-6736(20)30154-9, lire en ligne, consulté le )
- (en) Chaolin Huang, Yeming Wang, Xingwang Li et Lili Ren, « Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China », The Lancet, , S0140673620301835 (DOI 10.1016/S0140-6736(20)30183-5, lire en ligne, consulté le )
- (en) Elsevier, « Novel Coronavirus Information Center », sur Elsevier Connect (consulté le )
- (en) « COVID-19 Resource Centre », sur www.thelancet.com (consulté le )
- (en) « Coronavirus (Covid-19) — NEJM », sur New England Journal of Medicine (consulté le )
- (en) « Coronavirus », sur Science | AAAS (consulté le )
- « Coronavirus | Springer Nature | For Researchers | Springer Nature », sur www.springernature.com (consulté le )
- (en) « Novel Coronavirus - Wiley Online Library », sur Novel Coronavirus (consulté le )
- « WHO | WHO best practices for naming of new human infectious diseases », sur WHO (consulté le )
- (en) « Novel Coronavirus (2019-nCoV) Situation Report – 22 » [PDF], sur who.int, Organisation mondiale de la santé, (consulté le ) : « Following WHO best practices for naming of new human infectious diseases, which were developed in consultation and collaboration with the World Organisation for Animal Health (OIE) and the Food and Agriculture Organization of the United Nations (FAO), WHO has named the disease COVID-19, short for “coronavirus disease 2019.” », p. 1.
- « Epidémie de Covid-19 : la difficulté de bien nommer le virus et la maladie », Le Monde.fr, (lire en ligne, consulté le )
- (en) Shibo Jiang, Zhengli Shi, Yuelong Shu et Jingdong Song, « A distinct name is needed for the new coronavirus », The Lancet, , S0140673620304190 (DOI 10.1016/S0140-6736(20)30419-0, lire en ligne, consulté le )
- (en) Yuntao Wu, Wenzhe Ho, Yaowei Huang et Dong-Yan Jin, « SARS-CoV-2 is an appropriate name for the new coronavirus », The Lancet, , S0140673620305572 (DOI 10.1016/S0140-6736(20)30557-2, lire en ligne, consulté le )
- (en) Marie E. Killerby, Holly M. Biggs, Amber Haynes et Rebecca M. Dahl, « Human coronavirus circulation in the United States 2014–2017 », Journal of Clinical Virology, vol. 101, , p. 52–56 (DOI 10.1016/j.jcv.2018.01.019, lire en ligne, consulté le )
- (en) Hui DS, I Azhar E, Madani TA, Ntoumi F, Kock R,, Dar O, Ippolito G, Mchugh TD, Mchugh TD, Memish ZA et al., « The continuing 2019-nCoV epidemic threat of novel coronaviruses to global health – The latest 2019 novel coronavirus outbreak in Wuhan, China », International Journal of Infectious Diseases, vol. 91, , p. 264-266 (PMID 31953166, DOI 10.1016/j.ijid.2020.01.009, lire en ligne, consulté le )

- (en) Na Zhu, Dingyu Zhang, Wenling Wang et Xingwang Li, « A Novel Coronavirus from Patients with Pneumonia in China, 2019 », New England Journal of Medicine, , NEJMoa2001017 (ISSN 0028-4793 et 1533-4406, DOI 10.1056/NEJMoa2001017, lire en ligne, consulté le )
- (en-CA) « Wuhan Novel Coronavirus (2019-nCoV) », sur Ontario.ca, (consulté le )
- « Experts explain the latest bulletin of unknown cause of viral pneumonia » [archive du ], sur Wuhan Municipal Health Commission, (consulté le )
- (en) « Novel Coronavirus (2019-nCoV) situation reports », sur www.who.int (consulté le )
- « Coronavirus (SRAS-CoV et MERS-CoV) », sur Inserm - La science pour la santé (consulté le )
- (en) Anna Fifield, « As families tell of pneumonia-like deaths in Wuhan, some wonder if China virus count is too low », The Washington Post, (lire en ligne)
- (en-GB) Lily Kuo et Lillian Yang, « Coronavirus: Chinese hospitals not testing patients, say relatives », The Guardian, (ISSN 0261-3077, lire en ligne, consulté le )
- (en-US) Wenxin Fan, « Relatives Wonder Why Pneumonia Deaths Not in Coronavirus Tally », Wall Street Journal, (ISSN 0099-9660, lire en ligne, consulté le )
- « Coronavirus : ce que l'on sait des premiers patients touchés par l'épidémie », sur Franceinfo, (consulté le )
- « Déclaration du Directeur général de l’OMS sur les recommandations du Comité d’urgence du RSI sur le nouveau coronavirus », sur www.who.int, (consulté le )
- (en-US) Roni Caryn Rabin, « What Is the Coronavirus? Symptoms, Treatment and Risks », The New York Times, (ISSN 0362-4331, lire en ligne, consulté le )
- Clinical characteristics of 2019 novel coronavirus infection in China
- Trevor Bedford and Richard Neher, « Genomic epidemiology of novel coronavirus (nCoV) using data generated by Fudan University, China CDC, Chinese Academy of Medical Sciences, Chinese Academy of Sciences, Zhejiang Provincial Center for Disease Control and Prevention and the Thai National Institute of Health shared via GISAID », nextstrain.org (consulté le )
- « Initial genome release of novel coronavirus », Virological, (consulté le )
- (en) Wu, F. et al., « Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome », sur GenBank, (consulté le )
- « Phylogeny of SARS-like betacoronaviruses », nextstrain (consulté le )
- Hui DS, I Azhar E, Madani TA, Ntoumi F, Kock R, Dar O, Ippolito G, Mchugh TD, Memish ZA, Drosten C, Zumla A, Petersen E. The continuing 2019-nCoV epidemic threat of novel coronaviruses to global health – The latest 2019 novel coronavirus outbreak in Wuhan, China. Int J Infect Dis. 2020 Jan 14;91:264–266. DOI 10.1016/j.ijid.2020.01.009

- Antonio C. P. Wong, Xin Li, Susanna K. P. Lau, Patrick C. Y. Woo: Global Epidemiology of Bat Coronaviruses, in: Viruses. 2019 Feb; 11(2): 174, doi:10.3390/v11020174
- Ji, Wang, Zhao et Zai, « Homologous recombination within the spike glycoprotein of the newly identified coronavirus may boost cross‐species transmission from snake to human », Journal of Medical Virology, (DOI 10.1002/jmv.25682)
- CNN: Snakes could be the source of the Wuhan coronavirus outbreak
- (en-US) Robertson D.L., « nCoV's relationship to bat coronaviruses & recombination signals (no snakes) », sur Virological, (consulté le )
- (en) Dimitrios Paraskevis, Evangelia Georgia Kostaki, Gkikas Magiorkinis et Georgios Panayiotakopoulos, « Full-genome evolutionary analysis of the novel corona virus (2019-nCoV) rejects the hypothesis of emergence as a result of a recent recombination event », bioRxiv, Microbiology, (DOI 10.1101/2020.01.26.920249, lire en ligne, consulté le )
- (zh) « Notice d'information de la Commission municipale de la santé de Wuhan sur la situation actuelle de l'épidémie de pneumonie dans notre ville [en chinois] », sur http://wjw.wuhan.gov.cn/, (consulté le )
- (en-US) Antigone Barton, « UPDATE Wuhan coronavirus – 2019-nCoV Q&A #6: An evidence-based hypothesis », sur Science Speaks: Global ID News, (consulté le )
- (en) Jon CohenJan. 26 et 2020, « Wuhan seafood market may not be source of novel virus spreading globally », sur Science | AAAS, (consulté le )
- « Wuhan virus: a visual explainer », sur South China Morning Post (consulté le ).
- « #VraiOuFake Coronavirus : la consommation d'une soupe de chauve-souris à Wuhan est-elle à l'origine de l'épidémie ? », sur Franceinfo, (consulté le )
- (en-US) James Palmer, « Don’t Blame Bat Soup for the Wuhan Virus », sur Foreign Policy (consulté le )
- (en) Gretchen VogelDec. 30 et 2014, « Bat-filled tree may have been ground zero for the Ebola epidemic », sur Science | AAAS, (consulté le )
- « Coronavirus : le pangolin a-t-il pu servir d’hôte intermédiaire ? », Le Monde.fr, (lire en ligne, consulté le )
- (en) Ping Liu, Wu Chen et Jin-Ping Chen, « Viral Metagenomics Revealed Sendai Virus and Coronavirus Infection of Malayan Pangolins (Manis javanica) », Viruses, vol. 11, no 11, , p. 979 (ISSN 1999-4915, PMID 31652964, PMCID PMC6893680, DOI 10.3390/v11110979, lire en ligne, consulté le )
- (en-US) « expert reaction to statement from South China Agricultural University that research has identified the pangolin as a possible coronavirus host | Science Media Centre » (consulté le )
- (en) Sandeep Chakraborty, « Comment on "Viral Metagenomics Revealed Sendai Virus and Coronavirus Infection of Malayan Pangolins (Manis javanica)" », PubPeer, (lire en ligne, consulté le )
- « GISAID - Next betaCoV App », sur www.gisaid.org (consulté le )
- (en) « China coronavirus: Hong Kong scraps major Lunar New Year events », sur South China Morning Post, (consulté le )
- (en) hermesauto, « Wuhan virus: Vietnam confirms 2 cases of Sars-like coronavirus », The Straits Times, (consulté le )
- (en) hermesauto, « Singapore confirms first case of Wuhan virus », The Straits Times, (consulté le )
- « New Virus Spreads to U.S., Sparking Rush to Contain Outbreak », sur www.bloomberg.com, (consulté le )
- (en) Scott Morrison, « Update on Coronavirus pic.twitter.com/Ol8DRQtVQJ », sur @ScottMorrisonMP, (consulté le )
- « Coronavirus : un troisième cas d’infection confirmé en France », Le Monde.fr, (lire en ligne, consulté le )
- « Coronavirus : 5 nouveaux cas "sans gravité" en France dont un enfant de 9 ans scolarisé dans 2 écoles », sur lci.fr, (consulté le )
- APANEWS, « Coronavirus : l’Egypte enregistre le premier cas en Afrique », sur apanews.net (consulté le )
- (en) « Brazil confirms first coronavirus case in Latin America: source », Reuters, (lire en ligne, consulté le )
- (en-US) « Wuhan Coronavirus (2019-nCoV) global cases (by JHU CSSE) », sur Johns Hopkins University Center for Systems Science and Engineering (consulté le )
- (en) « Two thirds of COVID-19 cases exported from mainland China may be undetected | Imperial News | Imperial College London », sur Imperial News (consulté le )
- « L’épidémie de coronavirus très sous-estimée dans le monde », La Croix, (ISSN 0242-6056, lire en ligne, consulté le )
- (en) Associated Press, « China confirms human-to-human transmission of new coronavirus », sur CBC.ca, (consulté le )
- (en) « Statement on the meeting of the International Health Regulations (2005) Emergency Committee regarding the outbreak of novel coronavirus 2019 (n-CoV) on 23 January 2020 », sur www.who.int (consulté le )
- (en) Julien Riou et Christian L. Althaus, « Pattern of early human-to-human transmission of Wuhan 2019-nCoV », bioRxiv, , p. 2020.01.23.917351 (DOI 10.1101/2020.01.23.917351, lire en ligne, consulté le )
- « VERIFY: Can you get Coronavirus from packages shipped from China? », sur wbir.com (consulté le )
- « Déclaration de l’OMS sur le nouveau coronavirus en Thaïlande », sur www.who.int (consulté le )
- (en) Princess Cruises, « Princess Cruises: Diamond Princess Coronavirus & Quarantine Updates - Notices & Advisories », sur www.princess.com, mon feb 17 15:21:24 pst 2020 (consulté le )
- « Coronavirus: l'Italie, la Corée du Sud et l'Iran tentent d'endiguer l'épidémie », sur RFI, (consulté le )
- « Coronavirus : la contamination s’accélère à travers la planète », Le Monde.fr, (lire en ligne, consulté le )
- « Coronavirus : les Philippines enregistrent le premier décès hors de Chine », sur France 24, (consulté le )
- « Coronavirus : un premier décès sur le territoire français », sur Franceinfo, (consulté le )
- (en) Sandeep Chakraborty, « Ubiquitous genomic fragment in human 2019-nCoV viruses in the spike-protein, also encoding a novel 87 aa protein, completely missing in all other coronaviruses », OSF preprints, Open Science Framework, (DOI 10.31219/osf.io/6vd54, lire en ligne, consulté le )
- (en) Xu, X. et al., « Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission », SCIENCE CHINA Life Sciences, (lire en ligne)
- (en) Michael C. Letko et Vincent Munster, « Functional assessment of cell entry and receptor usage for lineage B β-coronaviruses, including 2019-nCoV », bioRxiv, (DOI 10.1101/2020.01.22.915660, lire en ligne, consulté le )
- (en) Markus Hoffmann, Hannah Kleine-Weber, Nadine Krueger et Marcel A Mueller, « The novel coronavirus 2019 (2019-nCoV) uses the SARS-coronavirus receptor ACE2 and the cellular protease TMPRSS2 for entry into target cells », bioRxiv, Molecular Biology, (DOI 10.1101/2020.01.31.929042, lire en ligne, consulté le )
- Christopher M. Coleman, Krystal L. Matthews, Lindsay Goicochea et Matthew B. Frieman, « Wild-type and innate immune-deficient mice are not susceptible to the Middle East respiratory syndrome coronavirus », The Journal of General Virology, vol. 95, no Pt 2, , p. 408–412 (ISSN 0022-1317, PMID 24197535, PMCID 3917065, DOI 10.1099/vir.0.060640-0, lire en ligne, consulté le )
- Christopher M. Coleman, Krystal L. Matthews, Lindsay Goicochea et Matthew B. Frieman, « Wild-type and innate immune-deficient mice are not susceptible to the Middle East respiratory syndrome coronavirus », The Journal of General Virology, vol. 95, no Pt 2, , p. 408–412 (ISSN 0022-1317, PMID 24197535, PMCID 3917065, DOI 10.1099/vir.0.060640-0, lire en ligne, consulté le )
- « Coronavirus : des passagers du paquebot en quarantaine ont échappé aux contrôles », sur Europe 1 (consulté le )
- "Nbci, 2011: Infection with human coronavirus NL63 enhances streptococcal adherence to epithelial cells"
- Lu H. Drug treatment options for the 2019-new coronavirus (2019-nCoV). Biosci Trends. 28 Jan 2020. DOI 10.5582/bst.2020.01020
- (en) « WHO says new China coronavirus could spread, warns hospitals worldwide », Reuters, (lire en ligne, consulté le )
- « China Testing HIV Drug as Treatment for New Coronavirus, AbbVie Says », The New York Times, (lire en ligne)
- (en) Zhijian Xu, Cheng Peng, Yulong Shi et Zhengdan Zhu, « Nelfinavir was predicted to be a potential inhibitor of 2019 nCov main protease by an integrative approach combining homology modelling, molecular docking and binding free energy calculation », bioRxiv, Pharmacology and Toxicology, (DOI 10.1101/2020.01.27.921627, lire en ligne, consulté le )
- Abdo Elfiky, « Sofosbuvir Can Inhibit the Newly Emerged Coronavirus (2019-nCoV) in Wuhan, China », (Preprints with the Lancet, Powered by SSRN),
- « Thai doctors have been using a cocktail of flu and HIV drugs to treat coronavirus cases », Business Insider, (lire en ligne, consulté le )
- « Coronavirus: Thailand has apparent treatment success with antiviral drug cocktail », South China Morning Post, (lire en ligne, consulté le )
- « Q&A on coronaviruses » [archive du ], sur World Health Organization (WHO) (consulté le )
- « China CDC developing novel coronavirus vaccine » [archive du ], sur Xinhua, (consulté le )
- "Nature, 4/02/20: Remdesivir and chloroquine effectively inhibit the recently emerged novel coronavirus (2019-nCoV) in vitro"
- Martin J Vincent, Eric Bergeron, Suzanne Benjannet et Bobbie R Erickson, « Chloroquine is a potent inhibitor of SARS coronavirus infection and spread », Virology Journal, vol. 2, no 1, , p. 69 (DOI 10.1186/1743-422X-2-69, lire en ligne, consulté le )
- Jianjun Gao, Zhenxue Tian et Xu Yang, « Breakthrough: Chloroquine phosphate has shown apparent efficacy in treatment of COVID-19 associated pneumonia in clinical studies », BioScience Trends, vol. advpub, (DOI 10.5582/bst.2020.01047, lire en ligne, consulté le )
- « La chloroquine est-elle un bon traitement contre le coronavirus ? », sur Sciences et Avenir (consulté le )
- (en) Philippe Colson, Jean-Marc Rolain et Didier Raoult, « Chloroquine for the 2019 novel coronavirus SARS-CoV-2 », International Journal of Antimicrobial Agents, , p. 105923 (DOI 10.1016/j.ijantimicag.2020.105923, lire en ligne, consulté le )
- « Coronavirus : le professeur Raoult, spécialiste des maladies infectieuses, valide un traitement contre le Covid-19 », sur France 3 Provence-Alpes-Côte d'Azur (consulté le )
- « ”Fake news” et désinformation autour du coronavirus SARS-CoV2 », sur Salle de presse | Inserm, (consulté le )
- Pauline Moullot, « La chloroquine, un médicament contre le paludisme, est-elle efficace contre le Covid-19 ? », sur Libération.fr, (consulté le )
- « DIRECT. Le gouvernement américain tente de développer un vaccin contre le coronavirus 2019-nCoV », sur Franceinfo, (consulté le ).
- « Coronavirus : « La France travaille à un vaccin » », La Croix, (ISSN 0242-6056, lire en ligne, consulté le )
- Pierre Sautreuil, « Coronavirus : mais au fait, c'était quoi le Sras ? », sur Le Figaro.fr, (consulté le )
- « COVID-19 : S’inspirer du SRAS pour développer le vaccin », sur santé log, (consulté le )
- (en) Xiaolong Tian, Cheng Li, Ailing Huang et Shuai Xia, « Potent binding of 2019 novel coronavirus spike protein by a SARS coronavirus-specific human monoclonal antibody », bioRxiv, Microbiology, (DOI 10.1101/2020.01.28.923011, lire en ligne, consulté le )
- (en) « Coronavirus: 'Significant breakthrough' in race for vaccine made by UK scientists », sur Sky News (consulté le )
- (en) « Imperial researchers in race to develop a coronavirus vaccine | Imperial News | Imperial College London », sur Imperial News (consulté le )
- Le Figaro avec Reuters, « Coronavirus: un vaccin en cours d'élaboration en Russie », sur Le Figaro.fr, (consulté le ).
- (en) « Coronavirus 'may become global emergency' as 18 deaths confirmed », sur Sky News (consulté le ).
- (en-GB) Haroon Siddique (now), Ben Quinn, Stephanie Convery (earlier) et Libby Brooks, « China coronavirus: 14 test negative in UK as military doctors sent to Wuhan – as it happened », The Guardian, (ISSN 0261-3077, lire en ligne, consulté le ).
- « Coronavirus : « La France travaille à un vaccin » », La Croix, (ISSN 0242-6056, lire en ligne, consulté le ).
- (de) « Gesundheitssenatorin begrüßt Ausweitung des Risikogebiets durch Robert-Koch-Institut », sur www.berlin.de, (consulté le )
- (en) Lin Li, Ting Sun, Yufei He et Wendong Li, « Epitope-based peptide vaccine design and target site characterization against novel coronavirus disease caused by SARS-CoV-2 », bioRxiv, , p. 2020.02.25.965434 (DOI 10.1101/2020.02.25.965434, lire en ligne, consulté le )
- (en-GB) « Vir Biotechnology discovers two antibodies against new coronavirus » (consulté le )
- (en) Anurodh Shankar Agrawal, Tianlei Ying, Xinrong Tao et Tania Garron, « Passive Transfer of A Germline-like Neutralizing Human Monoclonal Antibody Protects Transgenic Mice Against Lethal Middle East Respiratory Syndrome Coronavirus Infection », Scientific Reports, vol. 6, no 1, , p. 1–8 (ISSN 2045-2322, DOI 10.1038/srep31629, lire en ligne, consulté le )
- (en) Jia Liu, Xin Zheng, Qiaoxia Tong et Wei Li, « Overlapping and discrete aspects of the pathology and pathogenesis of the emerging human pathogenic coronaviruses SARS-CoV, MERS-CoV, and 2019-nCoV », Journal of Medical Virology, (ISSN 1096-9071, DOI 10.1002/jmv.25709, lire en ligne, consulté le )
- (en) « People can get the coronavirus more than once, experts warn — recovering does not necessarily make you immune », sur Business Insider France (consulté le )
Voir aussi
Articles connexes
- Épidémie du coronavirus de 2019-2020
- Maladie à coronavirus 2019 ("coronavirus disease 2019")
- Syndrome respiratoire aigu sévère (SRAS)
- Syndrome respiratoire du Moyen-Orient (MERS)
- Orthocoronavirinae (les coronavirus)
Liens externes
- CDC chinois (Chinese Center for Disease Control and Prevention)
- Institut de Biologie des Agents pathogènes (Institute of Pathogen Biology)
- Carte de l'épidémie actualisée en temps réel par le Center for Systems Science and Engineering de la Johns Hopkins University de Baltimore
- (en-US) Gardner L., « Mapping the Wuhan Coronavirus (2019-nCoV) », sur systems.jhu.edu, (consulté le )
- Questions fréquentes sur les nouveaux coronavirus et Rapports quotidiens de situation sur le nouveau Coronavirus (2019-nCoV) [en anglais] par l'Organisation Mondiale de la Santé
- Fiche-maladie régulièrement actualisée par l'Institut Pasteur
- « Coronavirus 2019-nCoV : le point sur la situation en France et dans le monde », sur le site du Dictionnaire Vidal (consulté le ).
- "Analyses bioinformatiques du coronavirus 2019-nCoV : pourquoi et comment ?", article rédigé par deux bioinformaticiens sur bioinfo-fr.net, 05 février 2020