« Origine africaine de l'Homme moderne » : différence entre les versions
m →ADN chromosomique Y : coquille |
m →Sortie d'Afrique : relecture, correction sources |
||
| Ligne 55 : | Ligne 55 : | ||
== Sortie d'Afrique == |
== Sortie d'Afrique == |
||
{{article connexe|Dispersion australe}} |
|||
[[Fichier:Red Sea2.png|vignette |
[[Fichier:Red Sea2.png|vignette|La traversée de la [[mer Rouge]].]] |
||
| ⚫ | |||
| ⚫ | |||
| ⚫ | Certains scientifiques pensent que seulement quelques personnes ont quitté l'Afrique dans le cadre d'une unique migration et qu'elles ont peuplé le reste du monde<ref> |
||
| ⚫ | Certains scientifiques pensent que seulement quelques personnes ont quitté l'Afrique dans le cadre d'une unique migration et qu'elles ont peuplé le reste du monde{{référence à confirmer}}<ref>{{lien web |langue=en |url=http://news.softpedia.com/news/Both-Aborigines-and-Europeans-Rooted-in-Africa-54225.shtml |titre=Both Australian Aborigines and Europeans Rooted in Africa |site=news.softpedia.org |consulté le=10 juillet 2018}} {{référence à confirmer}}</ref>. On estime qu'à partir d'une population de {{formatnum:2000}} à {{formatnum:5000}} personnes présentes en Afrique, seul un petit groupe de près de 150 personnes a franchi la Mer Rouge. C'est pourquoi, de tous les lignages présents en Afrique, seules les filles d'un seul lignage, L3, sont présentes hors d'Afrique. S'il y avait eu plusieurs migrations, on trouverait plus d'un lignage africain hors d'Afrique. Les filles du L3, les lignages M et N, sont peu fréquentes en Afrique (bien que l'haplogroupe M1 soit très ancien et diversifié en [[Afrique du Nord]] et en [[Corne de l'Afrique|Afrique du Nord-Est]]) et semblent y être arrivées récemment. Une explication possible est que ces mutations se sont passées en Afrique de l'Est peu avant l'exode et, par [[effet fondateur]], sont devenues les haplogroupes dominants après l'exode d'Afrique. Au contraire, les mutations ont pu se passer peu après l'exode d'Afrique. |
||
D'autres scientifiques ont proposé un modèle de dispersion multiple selon lequel il y aurait eu deux migrations hors d'Afrique : |
|||
* une par la Mer Rouge en traversant les régions côtières vers l'[[Asie du Sud|Inde]] (la Route de la Côte), qui serait représentée par l'Haplogroupe M ; |
|||
D'autres scientifiques ont proposé un modèle de dispersion multiple selon lequel il y aurait eu deux migrations hors d'Afrique, une par la Mer Rouge en traversant les régions côtières vers l'[[Asie du Sud|Inde]] (la [[Dispersion australe|Route de la Côte]]), qui serait représentée par l'Haplogroupe M ; un autre groupe de migrants, avec un Haplogroupe N, aurait suivi le Nil à partir de l'Afrique de l'Est, se dirigeant vers le nord et passant en [[Asie]] à travers le [[Sinaï]]. Puis, ce groupe se serait séparé dans plusieurs directions, certains allant en Europe et d'autres se dirigeant vers l'est en [[Asie]]. Cette hypothèse tente d'expliquer pourquoi l'Haplogroupe N est prédominant en Europe et pourquoi l'haplogroupe M y est absent. On pense que les preuves d'une migration par la côte ont été détruites par la montée du niveau de la mer pendant l'[[Holocène]]<ref>{{article |langue=en |consulté le=10 juillet 2018 |titre=Major genomic mitochondrial lineages delineate early human expansions |nom1=Nicole Maca-Meyer |nom2=Ana M. González |nom3=José M. Larruga |nom4=Carlos Flores |nom5=Vicente M. Cabrera |périodique=BMC Genetics|année=2001|volume=2|numéro=13 |doi=10.1186/1471-2156-2-13}}</ref>{{,}}<ref>{{lien web |langue=en |consulté le=10 juillet 2018 |url=http://archive.wikiwix.com/cache/?url=http://www.human-evol.cam.ac.uk/Projects/sdispersal/sdispersal.htm |titre=Searching for traces of the Southern Dispersal |auteur1=Marta Mirazón Lahr |auteur2=Mike Petraglia |auteur3=Stephen Stokes |éditeur=University of Cambridge}}</ref>. Au contraire, une population de pionniers européens qui initialement possédait à la fois les haplogroupes M et N pourrait avoir perdu l'haplogroupe M en raison d'une déviation accidentelle génétique résultant d'un goulot d'étranglement de population (c'est-à-dire d'un [[effet fondateur]]). |
|||
Aujourd'hui le détroit de [[Bab-el-Mandeb]] dans la [[mer Rouge]] fait environ 20 kilomètres de largeur. Il y a {{formatnum:50000}} ans, lorsque le niveau de la mer était {{unité|70|mètres}} plus bas qu'aujourd'hui, il était beaucoup plus étroit. Bien que les détroits n'aient jamais été complètement fermés, il est possible qu'il y ait eu des îles qui pouvaient être atteintes avec de simples radeaux. Des [[tumulus|tertres]] de coquilles datés de {{formatnum:125000}} ans ont été trouvés en [[Érythrée]], indiquant que le régime alimentaire des premiers hommes incluait des fruits de mer obtenus par ramassage sur le rivage. |
Aujourd'hui le détroit de [[Bab-el-Mandeb]] dans la [[mer Rouge]] fait environ 20 kilomètres de largeur. Il y a {{formatnum:50000}} ans, lorsque le niveau de la mer était {{unité|70|mètres}} plus bas qu'aujourd'hui, il était beaucoup plus étroit. Bien que les détroits n'aient jamais été complètement fermés, il est possible qu'il y ait eu des îles qui pouvaient être atteintes avec de simples radeaux. Des [[tumulus|tertres]] de coquilles datés de {{formatnum:125000}} ans ont été trouvés en [[Érythrée]], indiquant que le régime alimentaire des premiers hommes incluait des fruits de mer obtenus par ramassage sur le rivage. |
||
Version du 10 juillet 2018 à 10:49
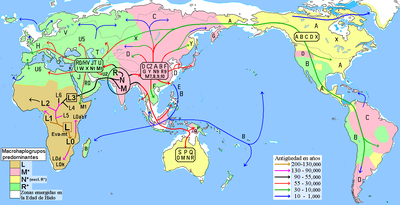
En paléoanthropologie, l'origine africaine de l'homme moderne est le modèle théorique dominant pour décrire l'origine et les premières migrations humaines de l'homme moderne, Homo sapiens. Cette théorie est connue du grand public sous le nom anglais d'Out of Africa (sortie d'Afrique), et sur le plan scientifique sous le nom d'« hypothèse d'une origine unique récente » (OUR), « hypothèse du remplacement » (HR) ou modèle de l'« origine africaine récente » (OAR). L'hypothèse selon laquelle l'homme moderne est originaire d'Afrique fut formulée dès 1871 dans La Filiation de l'homme et la sélection liée au sexe de Charles Darwin. Toutefois, l'idée est restée une spéculation jusque dans les années 1980, lorsqu'elle a été finalement corroborée par l'étude de l'ADN mitochondrial, puis de l'ADN nucléaire, alliée à des éléments fondés sur l'anthropologie physique de spécimens fossiles.
Selon des éléments à la fois génétiques et paléontologiques, l'Homo sapiens archaïque aurait évolué vers l'homme moderne en Afrique, entre environ 300 000 ans et 100 000 ans avant le présent.
La publication en 2017 d'une étude sur les Homo sapiens de Djebel Irhoud, datés de 300 000 ans, donna l'occasion à ses auteurs de défendre l'idée d'une émergence d'Homo sapiens à l'échelle de l'ensemble du continent africain, selon une sorte de modèle multirégional limité à l'Afrique. L'homme moderne aurait ensuite quitté l'Afrique il y a entre 100 000 et 70 000 ans, et se serait répandu sur tous les continents en remplaçant les espèces humaines antérieures, comme l'Homme de Néandertal et l'Homme de Denisova, avec des épisodes d'hybridation limitée entre espèces.
L'hypothèse concurrente est celle de l'origine multirégionale de l'homme moderne, ou de continuité avec hybridation, défendue depuis les années 1980 notamment par l'Américain Milford Wolpoff. Cette théorie repousse la date de la migration originelle hors d'Afrique à deux millions d'années avant le présent et l'associe à Homo ergaster plutôt qu'à Homo sapiens[1],[2]. Parfois nommée Out of Nowhere, elle propose un « grand métissage » entre les différentes populations humaines de la planète depuis des centaines de milliers d'années.
Histoire de la théorie
Avec l'essor de l'anthropologie au début du XIXe siècle, un débat virulent oppose les tenants du monogénisme comme Johann Friedrich Blumenbach et James Cowles Pritchard, pour qui les différentes races humaines sont des variétés partageant une ascendance commune, et ceux du polygénisme tels que Louis Agassiz et Josiah C. Nott, qui soutiennent que les races humaines sont des espèces distinctes ou se sont développées comme espèces distinctes par transmutation à partir de singes, sans avoir d'ancêtres communs.
Charles Darwin fut l'un des premiers à proposer une origine commune de tous les organismes vivants, et parmi les premiers à suggérer que tous les hommes avaient des ancêtres communs qui vivaient en Afrique. Dans La Filiation de l'homme et la sélection liée au sexe, publié en 1871, il émet l'idée selon laquelle les hommes descendent de singes qui possédaient un petit cerveau mais marchaient debout, ce qui libérait leurs mains et favorisa l'intelligence, et que ces singes étaient africains [3]:
« Dans chaque grande région du monde, les mammifères vivants sont étroitement apparentés aux espèces disparues de cette même région. C'est pourquoi il est probable que l'Afrique était autrefois habitée par des singes disparus étroitement apparentés au gorille et au chimpanzé ; et ces deux espèces sont maintenant les plus proches parents de l'homme, il est en un sens plus probable que nos lointains parents aient vécu sur le continent africain qu'ailleurs. Toutefois, il est inutile de spéculer sur cette question, car un singe presque aussi grand que l'homme, à savoir le Dryopithèque de Lartet, qui était étroitement apparenté à l'anthropomorphe gibbon, existait en Europe lors du Miocène supérieur ; et depuis une période aussi éloignée, la Terre a certainement subi bien de grandes révolutions, et il y a eu amplement le temps pour une migration à grande échelle[4]. »
Cette prédiction était perspicace, parce qu'en 1871, les scientifiques ne disposaient de quasiment aucun fossile d'hominines anciens. La conjecture de Darwin fut corroborée à partir de 1924, avec l'Enfant de Taung, quand les anthropologues commencèrent à trouver en Afrique des fossiles d'anciens hominines bipèdes à petit cerveau.
La majorité des anthropologues s'étaient ralliés à la théorie du monogénisme vers le milieu du XXe siècle, bien qu'il y ait encore eu quelques partisans du polygénisme vers la fin du XXe siècle, tels que Carleton Coon. Ce dernier a émis aussi tard qu'en 1962 l'hypothèse d'une évolution d’Homo sapiens à partir d’Homo erectus en cinq endroits et époques différents[5]. L'origine africaine de l'homme moderne implique une origine unique, c'est-à-dire un monogénisme, et a été utilisée dans plusieurs contextes comme un antonyme du polygénisme.
Le développement de l'archéogénétique dans les années 1990 a permis de dater la migration depuis le continent africain avec une certaine précision.
La question de l'existence d'un héritage génétique chez Homo sapiens provenant d'autres espèces du genre Homo reste discutée.
Premiers Homo sapiens
Homo sapiens est apparu en Afrique il y a plus de 300 000 ans[6],[7],[8],[9], et il aurait colonisé tout le continent avant de migrer hors d'Afrique. Selon Jean-Jacques Hublin, l'arbre phylogénétique de l'humanité est un « arbre dont il manque de nombreuses branches » et la surreprésentation de certaines régions et périodes serait due avant tout à l'abondance de fossiles en Afrique orientale, région dont les conditions ont été propices a la conservation des ossements[10].
Le passage de l'industrie lithique acheuléenne aux industries dites de mode 3, qui s'est produit en Afrique entre 400 000 et 300 000 ans avant le présent, témoignerait peut-être de la transition d'Homo rhodesiensis vers Homo sapiens.
L'Homme de Herto, découvert sur un site de la moyenne vallée de l'Awash en Éthiopie, vivait il y a environ 157 000 ans[11] et représente une forme relativement moderne d'Homo sapiens. Des fossiles humains furent mis au jour dans la grotte de Qafzeh en Israël et ont été datés d'environ 100 000 ans. Cependant, ces hommes semblent soit s'être éteints soit être retournés en Afrique il y a 70 000 à 80 000 ans, peut-être remplacés par des Néandertaliens méridionaux fuyant les régions les plus froides de l'Europe glaciaire. Hua Liu & al., analysant des marques microsatellites autosomiques, datent d'il y a environ 56 000 ans (±5 700 années) les traces ADN. Ils interprètent le fossile paléontologique de l'Homo sapiens de la caverne de Qafzeh comme une émanation récente isolée d'humains retournés en Afrique[12].
Le plus ancien fossile d'Homo sapiens trouvé hors d'Afrique a été découvert en 2002 dans la grotte de Misliya, en Israël. Il a été daté en 2018 de 185 000 ans[13],[14].
Il y a environ 100 000 ans des indices de technologies et d'artisanat plus sophistiqués commencent à apparaitre en Afrique du Sud, à Pinnacle Point puis à Blombos, et il y a environ 45 000 ans, un comportement pleinement moderne apparait en Europe. À cette époque, les morts bénéficient de sépultures[15]. Les outils en pierre tendent à se standardiser et les outils en os et en bois de cervidé font leur apparition[16],[17].
Reconstruction génétique
Deux éléments de la génétique humaine sont particulièrement utiles pour déchiffrer l'histoire de l'homme. L'un est l'ADN mitochondrial, et l'autre le chromosome Y. Ce sont les deux seules parties du génome qui ne se recombinent pas au moment de la fécondation. L'ADN mitochondrial et le chromosome Y sont transmis respectivement le long des lignées féminine et masculine. Sur la base de ces éléments, la totalité de l'humanité actuelle a hérité son ADN mitochondrial d'une femme qui vivait en Afrique il y a environ 160 000 ans[18],[19] ; cette femme a été baptisée Ève mitochondriale. Tous les hommes d'aujourd'hui ont hérité leurs chromosomes Y d'un homme qui vivait à une date controversée en Afrique. Cet homme est le plus récent ancêtre patrilinéaire commun.
ADN mitochondrial

Le premier lignage à se séparer du plus récent ancêtre matrilinéaire commun, surnommé « Ève mitochondriale », est l'haplogroupe L0. Cet haplogroupe se trouve en proportions élevées parmi les San d'Afrique australe et les Sandawe de Tanzanie. On trouve l'haplogroupe L1 (en) chez les Pygmées Binga[20],[21]. Les macro haplogroupes M et N, qui sont les lignages du reste du monde en dehors de l'Afrique, descendent du L3.
ADN chromosomique Y
Les mutations définissant les macro-haplogroupes CT (ADN-Y) (tous les haplogroupes Y sauf A et B) datent d'avant la migration hors d'Afrique, son descendant le macro-groupe DE étant confiné à l'Afrique. Les mutations qui distinguent l'haplogroupe C (ADN-Y) de tous les autres descendants de CT sont apparus il y a environ 140 000 ans, peu de temps avant la première migration hors d'Afrique.
L'haplogroupe F (ADN-Y) est apparu il y a environ 45 000 ans, en Afrique du Nord (dans ce cas il montrerait une seconde vague de migration hors d'Afrique) ou en Asie du Sud. Plus de 90 % des hommes nés ailleurs qu'en Afrique descendent en ligne masculine du premier porteur de l'haplogroupe F.
Analyse du génome
Bien que l'ADN mitochondrial et l'ADN chromosomique Y soient particulièrement utiles pour déchiffrer l'histoire humaine, les données sur les génomes de douzaines de groupes de population ont aussi été étudiées. En juin 2009, une analyse des données sur les génomes SNP de l'International HapMap Project (Phase II) et du CEPH Human Genome Diversity Panel samples a été publiée[22]. Ces modèles furent prélevés sur 1 138 individus qui n'étaient pas liés[22]. Avant cette analyse, les généticiens de population s'attendaient à trouver des différences importantes entre les groupes ethniques, avec des allèles partagés entre ces groupes mais différents ou n'existant pas dans les autres groupes[23]. Au lieu de cela, l'étude de 53 populations à partir des données des HapMap et CEPH a révélé que les groupes de population étudiés comprenaient seulement trois groupes génétiques : les Africains, les Eurasiens (qui incluent les natifs d'Europe et du Moyen-Orient, et les Asiatiques du Sud-Ouest à l'Est de l'actuel Pakistan), et les Asiatiques de l'Est, qui incluent les natifs de l'Asie, du Japon, d'Asie du Sud, des Amériques et d'Océanie[23]. L'étude a montré que la plupart des différences des groupes ethniques pouvaient être attribuées à la dérive génétique, avec des populations africaines modernes ayant une plus grande diversité génétique que les deux autres groupes génétiques et les Eurasiens modernes quelque chose de plus que les Asiatiques de l'Est modernes[23]. L'étude a suggéré que la sélection naturelle pouvait former le génome humain beaucoup plus lentement qu'on ne le pensait précédemment, avec des facteurs tels que la migration à l'intérieur et entre les continents ayant influencé plus profondément la distribution des variations génétiques[24].
Sortie d'Afrique

Il y a quelque 70 000 ans, une partie des porteurs de l'haplogroupe mitochondrial L3 ont migré d'Afrique orientale vers le Proche-Orient.
Certains scientifiques pensent que seulement quelques personnes ont quitté l'Afrique dans le cadre d'une unique migration et qu'elles ont peuplé le reste du monde[réf. à confirmer][25]. On estime qu'à partir d'une population de 2 000 à 5 000 personnes présentes en Afrique, seul un petit groupe de près de 150 personnes a franchi la Mer Rouge. C'est pourquoi, de tous les lignages présents en Afrique, seules les filles d'un seul lignage, L3, sont présentes hors d'Afrique. S'il y avait eu plusieurs migrations, on trouverait plus d'un lignage africain hors d'Afrique. Les filles du L3, les lignages M et N, sont peu fréquentes en Afrique (bien que l'haplogroupe M1 soit très ancien et diversifié en Afrique du Nord et en Afrique du Nord-Est) et semblent y être arrivées récemment. Une explication possible est que ces mutations se sont passées en Afrique de l'Est peu avant l'exode et, par effet fondateur, sont devenues les haplogroupes dominants après l'exode d'Afrique. Au contraire, les mutations ont pu se passer peu après l'exode d'Afrique.
D'autres scientifiques ont proposé un modèle de dispersion multiple selon lequel il y aurait eu deux migrations hors d'Afrique, une par la Mer Rouge en traversant les régions côtières vers l'Inde (la Route de la Côte), qui serait représentée par l'Haplogroupe M ; un autre groupe de migrants, avec un Haplogroupe N, aurait suivi le Nil à partir de l'Afrique de l'Est, se dirigeant vers le nord et passant en Asie à travers le Sinaï. Puis, ce groupe se serait séparé dans plusieurs directions, certains allant en Europe et d'autres se dirigeant vers l'est en Asie. Cette hypothèse tente d'expliquer pourquoi l'Haplogroupe N est prédominant en Europe et pourquoi l'haplogroupe M y est absent. On pense que les preuves d'une migration par la côte ont été détruites par la montée du niveau de la mer pendant l'Holocène[26],[27]. Au contraire, une population de pionniers européens qui initialement possédait à la fois les haplogroupes M et N pourrait avoir perdu l'haplogroupe M en raison d'une déviation accidentelle génétique résultant d'un goulot d'étranglement de population (c'est-à-dire d'un effet fondateur).
Aujourd'hui le détroit de Bab-el-Mandeb dans la mer Rouge fait environ 20 kilomètres de largeur. Il y a 50 000 ans, lorsque le niveau de la mer était 70 mètres plus bas qu'aujourd'hui, il était beaucoup plus étroit. Bien que les détroits n'aient jamais été complètement fermés, il est possible qu'il y ait eu des îles qui pouvaient être atteintes avec de simples radeaux. Des tertres de coquilles datés de 125 000 ans ont été trouvés en Érythrée, indiquant que le régime alimentaire des premiers hommes incluait des fruits de mer obtenus par ramassage sur le rivage.
Expansion
Il y a 100 000 à 70 000 ans, Homo sapiens se répand du Proche-Orient vers l'est et l'Asie du Sud. Il y a 60 000 ans, d'autres poursuivent vers l'Australie où, pour la première fois, Homo sapiens colonise un territoire vierge de toute population humaine. Cro-Magnon arrive en Europe il y a environ 45 000 ans. Le Japon est atteint il y a 30 à 40 000 ans. On débat encore pour déterminer la date du premier peuplement de l'Amérique, entre 50 000 ans et 14 000 ans avant le présent.
La différenciation entre groupes humains est due à une longue séparation géographique causée probablement par les glaciations, causant une différenciation allopatrique, début d'un phénomène de spéciation. Les populations d'Homo sapiens ayant atteint le centre de l'Eurasie vers -60 000 ans se sont retrouvées isolées par les masses glaciaires de l'Himalaya, de l'Hindou Kouch, ainsi que par les déserts arides (l'atmosphère devient plus sèche lors des glaciations, et les déserts s'étendent) de l'Asie du Sud lors du stade II de la glaciation de Würm (-70 000 à -50 000 ans). Avec une intense pression environnementale instable, ils ont dû s'adapter, physiquement et culturellement, à des climats froids, tout comme l'avaient fait avant eux les hommes de Neandertal en Europe. Cet isolement explique la dépigmentation des populations européennes et est-asiatiques et les nombreuses différences morphologiques et physiologiques, tandis que les populations restant dans les climats tropicaux d'Asie du Sud ont conservé leur pigmentation les protégeant du soleil, comme les Négritos des îles Andaman, les Papous ou les Aborigènes d'Australie. Les populations du Nord de l'Eurasie ont ensuite pu s'étendre dans le reste du continent eurasien lors de la période de réchauffement de l'interstade de Laufen (-50 000 à -30 000 ans). L'épisode glaciaire suivant (Würm IV, de -30 000 à -10 000 ans) aurait séparé les populations « européennes » des populations « asiatiques » et permis, par la baisse du niveau des eaux, l'expansion humaine en Amérique.
Le groupe qui a traversé la Mer Rouge a voyagé le long de la route de la côte autour de la côte de l'Arabie et de la Perse jusqu'à atteindre l'Inde, ce qui semble être le premier point majeur de peuplement. L'haplogroupe M est trouvé fréquemment le long des régions des côtes sud du Pakistan et de l'Inde et il possède sa plus grande diversité en Inde, démontrant que c'est ici que les mutations se sont passées[28]. 60 % de la population indienne appartient à l'Haplogroupe M. Le peuple indigène des Îles Andaman appartient également au lignage M. On pense que les Andamanais possèdent les gènes des premiers habitants de l'Asie en raison de leur long isolement de l'Asie continentale. On trouve des traces de l'utilisation de la route de la côte par les premiers colons d'Asie de l'Inde jusqu'aux côtes de Thaïlande et d'Indonésie jusqu'en Papouasie-Nouvelle-Guinée. Puisque l'haplogroupe M est également trouvé fréquemment dans les hautes terres de la Nouvelle-Guinée, et que les Andamanais et les habitants de la Nouvelle-Guinée ont une peau sombre et des cheveux de type africain, certains scientifiques pensent que ceux-ci font tous partie de la même vague d'immigration ayant franchi la Mer Rouge il y a environ 60 000 ans au cours d'une migration côtière. De façon notable, les recherches de Harding et al. (2000, p. 1355) montrent que, au moins en raison de la couleur de la peau sombre, le fond de l'haplotype MC1R des Papous-Néo Guinéens (un des nombreux gènes impliqués dans la production de mélanine) est le même que celui des africains (à l'exception d'une seule mutation silencieuse). Ainsi, bien que ces groupes soient distincts des africains d'autres régions (à cause de la déviation, des goulets d'étranglement, etc.), il est évident que la sélection de la caractéristique de la couleur de peau sombre a sûrement continué (au moins pour le MC1R) après l'exode. Cela démontrerait l'hypothèse selon laquelle les migrants originels venus d'Afrique ressemblaient aux africains d'après l'exode (au moins s'agissant de la couleur de la peau) et expliqueraient que les restes actuels de cet ancien phénotype puissent être trouvés chez les africains actuels, les Andamanais et les Néo-Guinéens. D'autres suggèrent que leur ressemblance physique pourrait résulter de leur évolution convergente[29],[30],[31].
De l'Arabie à l'Inde, la fréquence de l'haplogroupe M augmente vers l'est : dans l'Inde de l'est, l'haplogroupe M surpasse en nombre l'haplogroupe N par un ratio de 3 à 1. Cependant, plus on va vers l'Asie de l'Est, plus l'haplogroupe N réapparait en tant que lignage dominant. L'haplogroupe M est prédominant en Asie du Sud-Est mais parmi les Aborigènes d'Australie, l'haplogroupe N réapparait comme le lignage le plus commun. Cette répartition discontinue de l'haplogroupe N de l'Europe à l'Australie peut s'expliquer par un effet fondateur et un goulet d'étranglement de population[32].
Hybridations
Des résultats génétiques récents ont démontré l'hybridation des branches non africaines de l'homme moderne avec l'homme de Néandertal d'une part et l'Homme de Denisova d'autre part, qui auraient eu lieu après la sortie d’Afrique des premiers Hommes modernes.
Un séquençage partiel de l'ADN nucléaire d'ossements de néandertaliens a été effectué en 2010 par une équipe de l'Institut Max Planck coordonnée par Svante Pääbo[33],[34]. La comparaison avec les mêmes séquences d'humains modernes montre que 2,2 % en moyenne des polymorphismes nucléotidiques de l'ADN d'un non-africain actuel sont d'origine néandertalienne ; ces croisements auraient pu survenir il y a 100 000 à 60 000 ans au Proche-Orient[35].
Fin 2010, selon une étude basée sur le séquençage de l'ADN nucléaire extrait d'une phalange, l'Homme de Denisova[36] aurait également contribué à hauteur de 4 à 6 % au génome des Mélanésiens et Aborigènes d'Australie actuels et aurait été relativement répandu en Asie à la fin du Pléistocène.
En 2013, une étude publiée dans le Journal of Biological Chemistry[37] relate la découverte dans le génome de l'homme moderne européen d'un gène lié à l'immunité qui pourrait être issu du génome de l'homme de Néandertal[38].
En 2014, l'étude du génome d'un Homo sapiens découvert à Kostenki, en Russie, et daté de 37 000 ans avant le présent, confirme encore l'hybridation et permet d'avancer une date à laquelle elle aurait eu lieu[39],[40].
Hypothèses concurrentes
Selon la thèse de l'origine multirégionale de l'homme moderne proposée par Milford Wolpoff, l'évolution de l'homme à partir d'Homo ergaster, il y a 1,8 million d'années, jusqu'à nos jours a eu lieu au sein d'une population unique et continue à travers le monde. Ceux qui soutiennent la thèse de l'origine multirégionale rejettent l'hypothèse de l'isolement reproductif entre les différentes populations africaines et eurasiennes appartenant aux anciennes espèces du genre Homo. Ceux-ci s'appuient sur les restes fossiles et des preuves génétiques se trouvant dans l'ADN chromosomique. Les résultats récents sur les hybridations entre l'homme moderne, Neandertal et Denisova, viennent donner des éléments de crédit supplémentaires à cette théorie sans toutefois l'accréditer globalement. Selon ces résultats, l'origine de l'homme moderne resterait malgré tout principalement africaine avec une petite part mixte.
Références
- http://www.actionbioscience.org/evolution/johanson.html Origins of Modern Humans : Multiregional or Out of Africa ? by Donald Johanson
- http://discovermagazine.com/2002/aug/featafrica Discover : Not Out of Africa, Alan Thorne's challenging ideas about human evolution
- Bowler 2003, p. 213
- The descent of man Chapter 6 - On the Affinities and Genealogy of Man
- Jackson Jr., John P. (2001). "InWays Unacademical": The Reception of Carleton S. Coon's The Origin of Races
- (en) Jean-Jacques Hublin, Abdelouahed Ben-Ncer et al., « New fossils from Jebel Irhoud, Morocco and the pan-African origin of Homo sapiens », Nature, vol. 546, , p. 289-292 (DOI 10.1038/nature22336)
- (en) Daniel Richter et al., « The age of the hominin fossils from Jebel Irhoud, Morocco, and the origins of the Middle Stone Age », Nature, vol. 546, , p. 293-296 (DOI 10.1038/nature22335)
- (en) Michael Balter, « Was North Africa the Launch Pad for Modern Human Migrations ? », Science, vol. 331, no 6013, , p. 20-23 (DOI 10.1126/science.331.6013.20, lire en ligne)
- (en) Fulvio Cruciani, Beniamino Trombetta, Andrea Massaia, Giovanni Destro-Bisol, Daniele Sellitto et Rosaria Scozzari, « A Revised Root for the Human Y Chromosomal Phylogenetic Tree : The Origin of Patrilineal Diversity in Africa », The American Journal of Human Genetics, (lire en ligne)
- Jean Jacques Hublin, Nicolas Martin, « Faut-il réécrire les débuts de l’histoire de l’Homme ? », France Culture, (lire en ligne, consulté le )
- (en) White, Tim D., Asfaw, B., DeGusta, D., Gilbert, H., Richards, G.D., Suwa, G. and Howell, F.C., « Pleistocene Homo sapiens from Middle Awash, Ethiopia », Nature, vol. 423, no 6491, , p. 742–747 (DOI 10.1038/nature01669)
- Hua Liu, et al., « A Geographically Explicit Genetic Model of Worldwide Human-Settlement History », The American Journal of Human Genetics, volume 79 (2006), pages 230–237.
- (en) Israel Hershkovitz et al., « The earliest modern humans outside Africa », Science, vol. 359, no 6374, , p. 456-459 (DOI 10.1126/science.aap8369)
- Frédéric Lewino, « Israël : découverte du plus vieux fossile appartenant à un homme moderne », Le Point, (lire en ligne, consulté le )
- Maureille, B. (2004) - Les premières sépultures, Paris, Le Pommier, Cité des sciences et de l'industrie, 123 p.
- (en) « Ancestral tools », Handprint.com, (consulté le )
- Middle to upper paleolithic transition
- Cann, R. L., Stoneking, M., Wilson, A. C. (1987) « Mitochondrial DNA and human evolution », Nature, vol. 325., pp. 31-36.
- Vigilant, L., Stoneking, M., Harpending, H., Hawkes, K., Wilson, A. C. (1991) « African Populations and the Evolution of Human Mitochondrial DNA », Science, vol. 253, pp. 1503-1507.
- Whole-mtDNA Genome Sequence Analysis of Ancient African Lineages
- mtDNA Variation in the South African Kung and Khwe
- (en) Coop G, Pickrell JK, Novembre J, Kudaravalli S, Li J, et al, « The Role of Geography in Human Adaptation », PLoS Genet, vol. 5, no 6, , p. 1–16 (DOI 10.1371/journal.pgen.1000500, lire en ligne)
- « Among Many Peoples, Little Genomic Variety », The Washington Post, (consulté le )
- « Geography And History Shape Genetic Differences In Humans », Science Daily, (consulté le )
- (en) « Both Australian Aborigines and Europeans Rooted in Africa », sur news.softpedia.org (consulté le ) [réf. à confirmer]
- (en) Nicole Maca-Meyer, Ana M. González, José M. Larruga, Carlos Flores et Vicente M. Cabrera, « Major genomic mitochondrial lineages delineate early human expansions », BMC Genetics, vol. 2, no 13, (DOI 10.1186/1471-2156-2-13)
- (en) Marta Mirazón Lahr, Mike Petraglia et Stephen Stokes, « Searching for traces of the Southern Dispersal », University of Cambridge (consulté le )
- Most of the extant mtDNA boundaries in South and Southwest Asia were likely shaped during the initial settlement of Eurasia by anatomically modern humans
- « Evolution of Human Languages »(Archive.org • Wikiwix • Archive.is • Google • Que faire ?) (consulté le )
- genetic origins of the Andaman Islanders
- « Genetic affinities of the Andaman Islanders »(Archive.org • Wikiwix • Archive.is • Google • Que faire ?) (consulté le )
- Mitochondrial Genome Variation and Evolutionary History of Australian and New Guinean Aborigines
- (en) Richard E. Green et al., « A Draft Sequence of the Neandertal Genome », Science, vol. 328, no 5979, , p. 710-722 (DOI 10.1126/science.1188021, lire en ligne)
- Hervé Morin, « Il y a du Néandertal en nous », Le Monde, (lire en ligne).
- Rachel Mulot, « On a tous quelque chose de Néandertal », Sciences et Avenir, , p. 30.
- (en) Reich et al., « Genetic history of an archaic hominin group from Denisova Cave in Siberia », Nature, vol. 468, p. 1053-1060, 23 décembre 2010.
- (en) Sebastian Temme, Martin Zaccharias, Jürgen Neumann, Sebastian Wohlfromm, Angelika König, Nadine Temme, Sebastian Springer, John Trowsdale et Norbert Koch, « A novel family of human lymphocyte antigen class II receptors may have its origin in archaic human species », Journal of Biological Chemistry, (lire en ligne).
- « Un gène de l'immunité qui nous vient de l’Homme de Néandertal », sur futura-sciences.com
- (en) Seguin-Orlando et al., « Genomic structure in Europeans dating back at least 36,200 years », Science, (lire en ligne).
- « L'homme de Kostenki précise le métissage Sapiens-Néandertal », sur fr.news.yahoo.com
Bibliographie
- Peter J. Bowler, Evolution: The History of an Idea, University of California Press, , 3e éd. (ISBN 0-520-23693-9)
- Charles Darwin, The Descent of Man, and Selection in Relation to Sex, London, John Murray, , 1re éd. (lire en ligne)
- Modern Men Trace Ancestry to African Migrants, Science, 11 mai 2001
- Underhill et al. (2001). "The phylogeography of Y chromosome binary haplotypes and the origins of modern human populations" Ann. Hum. Genet. 65: 43-62. « PDF »(Archive.org • Wikiwix • Archive.is • Google • Que faire ?) (consulté le ). Retrieved 25 July 2007.
- Neanderthals 'mated with modern humans', BBC News, 21 avril 1999
- New analysis shows three human migrations out of Africa - Replacement theory 'demolished', Université Washington à Saint-Louis, 2 février 2006
- (en) Rosalind M. Harding, « Evidence for variable selective pressures at MC1R », American Journal of Human Genetics, vol. 66, , p. 1351–1361 (DOI 10.1086/302863)
- Long and Kittles (2003). "Human genetic variation and the nonexistence of human races" Human Biology, 75: 449-471. PDF. Retrieved 10 January 2007.
- (en) Risch, N., Burchard, E., Ziv, E. and Tang, H., « Categorization of humans in biomedical research: genes, race and disease », Genome Biology, vol. 3, no 7, , comment2007.2001 - comment2007.2012 (DOI 10.1186/gb-2002-3-7-comment2007)
- Tishkoff, S. and Kidd, K. Implications of biogeography of human populations for 'race' and medicine Nature Genetics 36: S21 - S27 (2004) DOI 10.1038/ng1438
- Cavalli-Sforza, Luigi Luca and Francesco Cavalli-Sforza, The Great Human Diasporas - The History of Diversity and Evolution (Italian original Chi Siamo: La Storia della Diversit`a Umana), (ISBN 0-201-44231-0) (paperback), 1993.
- Crow, Tim J, Editor The Speciation of Modern Homo Sapiens, (ISBN 0-19-726311-9) (paperback) 2002.
- Foley, Robert, Humans Before Humanity, (ISBN 0-631-20528-4) (paperback), 1995.
- Olsen, Steve, Mapping Human History: Discovering the past through our genes (ISBN 0-618-35210-4)2002
- Oppenheimer, Stephen, The Real Eve: Modern Man's Journey Out of Africa, (ISBN 0-7867-1192-2) (Hardcover), 2003.
- Stringer, Chris and Robin McKie, African Exodus, (ISBN 0-7126-7307-5) (paperback), 1996.
- Sykes, Bryan, Les sept filles d'Ève : The Science That Reveals Our Genetic Ancestry (2002) (ISBN 0-552-15218-8)
- Wade, Nicholas, Before the Dawn: Recovering the Lost History of Our Ancestors (2006) (ISBN 1-59420-079-3)
- Wells, Spencer, The Journey of Man: A Genetic Odyssey (2003) (ISBN 0-691-11532-X)
- Wells, Spencer, Deep Ancestry: Inside the Genographic Project (2006) (ISBN 0-7922-6215-8)
- "New Research Proves Single Origin Of Humans In Africa," Science Daily, July 19, 2007, retrieved July 19, 2007
- Climate change led mankind out of Africa
- Ancient African Megadroughts May Have Driven Human Evolution Out Of Africa
- DNA studies trace human migration from Africa retrieved February 21, 2008
Voir aussi
Articles connexes
- Macrohaplogroupe L (ADNmt)
- Origine multirégionale de l'homme moderne
- Théorie de la catastrophe de Toba
- Behavioral modernity (en)
- Early human migrations (en)
- Identical ancestors point (en)
- Dernier ancêtre commun
- Ève mitochondriale
- Plus récent ancêtre patrilinéaire commun
- Sahara pump theory
- Hybridation entre les humains archaïques et modernes
