« Micro-ARN » : différence entre les versions
m références antérieures |
|||
| Ligne 26 : | Ligne 26 : | ||
=== Origine et évolution === |
=== Origine et évolution === |
||
Les miARN sont bien conservés chez les organismes [[eucaryote]]s et seraient une composante ancestrale et indispensable de la régulation de l’expression des gènes{{référence souhaitée}}. |
Les miARN sont bien conservés chez les organismes [[eucaryote]]s et seraient une composante ancestrale et indispensable de la régulation de l’expression des gènes{{référence souhaitée}}. |
||
La voie des miARNs, chez les animaux et chez les plantes, pourrait avoir une origine différente et être apparue de manière indépendante<ref>{{Lien PMID|21554756}}</ref>. |
|||
La voie des miARNs dériverait d'un système plus ancestral, l'[[interférence par ARN]]<ref>{{Lien PMID|18715673}}</ref>. |
La voie des miARNs dériverait d'un système plus ancestral, l'[[interférence par ARN]]<ref>{{Lien PMID|18715673}}</ref>. |
||
Les enzymes Dicer sont notamment responsables de la production de [[ARN interférent|petits ARN interférents (pARNi)]] à partir de longs ARNs double-brin<ref>Cette publication discute des processus où sont impliqués les siRNAs et les microARNs, dans le contexte de deux articles publiés dans le journal ''[[Science (revue)|Science]]''. {{Lien PMID|12242426}}</ref>. |
Les enzymes Dicer sont notamment responsables de la production de [[ARN interférent|petits ARN interférents (pARNi)]] à partir de longs ARNs double-brin<ref>Cette publication discute des processus où sont impliqués les siRNAs et les microARNs, dans le contexte de deux articles publiés dans le journal ''[[Science (revue)|Science]]''. {{Lien PMID|12242426}}</ref>. |
||
Version du 9 avril 2020 à 14:33

Les microARNs (abréviation : miARN) sont une catégorie de petits (en) acides ribonucléiques (ARNs), simple-brin, non-codants et propres aux cellules eucaryotes[1]. Ils ont une longueur moyenne de 22 nucléotides (en général de 21 à 24), soit moins que d'autres ARNs.
Les miARNs sont des régulateurs post-transcriptionnels capables d’extinction de l’expression d’un gène[2],[3] : leur appariement à un ARN messager (ARNm) cible peut conduire à l'inhibition de sa traduction ou à sa dégradation selon le degré de complémentarité entre la séquence du miARN et celle de son ARNm cible[4]. Parce qu’ils affectent l’expression de nombreux gènes, les miARNs interviennent dans la plupart des processus biologiques. Les miARNs ont été impliqués dans de nombreux processus, allant du développement[5] à la formation de tumeurs[6].
Le génome humain contiendrait environ 2 000 gènes à l’origine de la transcription de miARNs[7],[8] qui cibleraient environ 60 % des gènes [9],[10]. Il a été mis en évidence que les miARNs peuvent être exprimés différentiellement d’un tissu ou d’un type cellulaire à l’autre ; ils sont abondants dans plusieurs types cellulaires chez l'humain[réf. souhaitée].
L’expression aberrante de certains miARNs est associée à de nombreuses pathologies[5] ; des thérapies fondées sur une correction de leur abondance sont actuellement à l’étude[11].
Historique
Le premier miARN, lin-4 (en), a été caractérisé au début des années 1990 chez le nématode C. elegans[12]. Les auteurs de cette étude ont découvert que ce petit ARN s'exprimait à la fin du premier stade du développement larvaire. Il se fixait alors à l'ARNm de la protéine LIN-14, inhibait sa synthèse et, ce faisant, induisait la transition entre les deux premières phases larvaires. lin-4 avait donc un rôle fondamental dans le contrôle de la séquence des phases du développement.
Il fallut toutefois attendre le début des années 2000 et la découverte de let-7 (en) chez C. elegans, ainsi que celle d'autres miARNs chez différentes espèces, pour qu'un mécanisme de régulation post-transcriptionnel par les miARNs puisse être généralisable[13].
Biosynthèse

Origine et évolution
Les miARN sont bien conservés chez les organismes eucaryotes et seraient une composante ancestrale et indispensable de la régulation de l’expression des gènes[réf. souhaitée]. La voie des miARNs, chez les animaux et chez les plantes, pourrait avoir une origine différente et être apparue de manière indépendante[14]. La voie des miARNs dériverait d'un système plus ancestral, l'interférence par ARN[15]. Les enzymes Dicer sont notamment responsables de la production de petits ARN interférents (pARNi) à partir de longs ARNs double-brin[16]. Ce processus, décrit chez la plante, agit comme défense antivirale[17].
Traitement nucléaire
Les miARNs sont transcrits, de manière canonique, par une ARN polymérase de type II sous la forme de longs précurseurs tige-boucle appelés miARNs primaires (pri-miARN)[1] (figure 1, gauche). Ces pri-miARNs sont ensuite clivés par un complexe protéique hétérotrimérique nommé "microprocesseur" (en), formé, notamment, d'une molécule de la ribonucléase III Drosha (en) et deux molécules de son partenaire DGCR8 (en) (Di George syndrome Critical Region 8 - appelé Pasha chez la drosophile et le nématode)[18]. Un produit intermédiaire appelé miARN précurseur (pre-miARN) est ainsi produit.
Un certain nombre de miARNs échappent au traitement par Drosha. Dans certains cas, les pre-miARNs sont produits à partir d'introns par le spliceosome lors de l'épissage, et alors appelés "miRtrons"[19] (figure 1, droite). Dans d'autres cas, les pre-miARNs sont produits à partir d'ARNt[20] ou de petits ARNs nucléolaires (snoRNA pour small nucleolar RNAs)[21].
Le pre-miARN a une longueur d'environ 60 nucléotides, replié en tige-boucle imparfaite par complémentarité de bases entre la première moitié et la deuxième moitié de sa séquence.
Les pre-miARNs sont transportés de manière active, dépendante du GTP, depuis le noyau vers le cytosol part l'exportine 5 (en).
Traitement cytoplasmique
Le pre-miARN est clivé par un complexe enzymatique comprenant un membre de la famille de la ribonucléase III Dicer et son partenaire protéique TRBP (appelé Loquatious chez la drosophile). Dicer permet l'hydrolyse de la structure boucle du pre-miARN et libère un miARN mature, aux extrémités 3' cohésives, appelé « duplex miARN/miARN* (lire miARN étoile) » - ou miARNdb (db pour bouble brin) - avec un brin guide et un brin passager (figure 1).
Il existe au moins une exception à la maturation des miARNs par Dicer, c'est le cas de miR-451 (en), un miARN spécifique des vertébrés et impliqué dans l’érythropoïèse. Dans ce cas, c'est la protéine AGO2 (de la famille Argonaute) qui clive la structure tige-boucle au milieu du bras 3'[22],[23]. Une ribonucléase (PARN) rogne ensuite dans le sens 3' vers 5' pour générer le brin guide de miR-451[24].
Mode d'action
Un brin du duplex miARN/miARN* interagit avec une protéine de la famille Argonaute (AGO1 ou AGO2) pour former un complexe "RISC" (RNA-induced silencing complex) parfois appelé "miRISC" (figure 3). Ce complexe d'environ 160 kDa a été décrit comme étant suffisant à l'activité de gene silencing des miARNs, cependant d'autres protéines tel que la géminine peuvent s'y ajouter pour former des complexes allant jusqu'à 550 kDa[réf. souhaitée]. Le complexe RISC recrute une protéine de la famille GW182 qui recrute à son tour plusieurs effecteurs.

Au cours de la formation du miRISC il y a passage d'un miARNdb à un miARNsb (sb=simple brin). La choix du brin restant associé au miRISC s'effectue selon des principes complexes, avec en général une voie majeure et une voie mineure pouvant coexister[25]. La sélection du brin conservé par le miRISC se fait sur la base de l'extrémité 5' qui est la plus à même de se loger au sein de la protéine Argonaute. Celle-ci favorise la présente d'un nucléotide A ou U ainsi que l'appariement le moins stable à l'extrémité 5'[1].
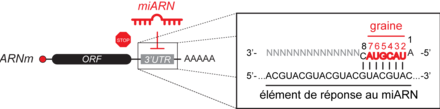
L'ARNm cible est ensuite chargé au sein du complexe miRISC selon la règle de complémentarité des bases avec le miARN. L’hybridation des miARNs à leur cible est cependant déterminée préférentiellement par une région restreinte du miARN s’étendant des nucléotides 2 à 7 et appelée "graine" (figure 2). Un miARN peut donc cibler plusieurs sites sur un même ARNm ou cibler plusieurs ARNm ayant une séquence de reconnaissance similaire, appelée élément de réponse aux miARNs (MRE, pour miRNA Response Elements) (figure 2). Chez les métazoaires, le site d’hybridation se trouve majoritairement dans la région 3’ non traduite (3’UTR).
Deux voies de « silencing » sont alors possibles, soit un clivage endonucléolytique de l'ARNm cible si la complémentarité avec le miARN est importante, soit une répression de la traduction, associée à une dégradation exonucléolytique des ARNm, dans le cas où la complémentarité entre miARN et ARNm est imparfaite (figure 3).
Chez les plantes

Il existe certaines différences entre la biosynthèse et le mode d'action des miARNs chez les plantes et chez les métazoaires. Chez les plantes, le nombre de miARNs serait plus restreint et il n'y aurait pas d'intermédiaires pri-miARNs (figure 4). La formation du duplex miARN/miARN* par DCL1 (en) (un homologue de Dicer) a lieu dans le noyau ; le duplex mature est ensuite transporté dans le cytoplasme. De plus, la répression post-transcriptionnelle implique généralement une hybridation parfaite ou quasi parfaite entre le miARN et son ARNm cible, elle va induire une coupure de l'ARNm au milieu du site d'hybridation du miARN[27]. Enfin, chez les plantes, si le site d’hybridation avec le miARN peut se trouver dans la région 3’UTR de l'ARNm, il se situe le plus souvent dans la séquence codante[réf. souhaitée].
Dans certains cas, comme chez la mousse Physcomitrella patens, l'accumulation de duplex miARN:ARN cible pourrait participer à la formation d'un complexe appelé "RITS" (en) (RNA-induced initiation of transcriptional gene silencing) qui, s'associant directement à l'ADN induirait sa méthylation conduisant à une « extinction » de cibles des miARNs[28]. Il n'est pas exclu qu'un tel processus existe chez l'animal[réf. souhaitée].
Régulations
Les miARNs sont régulés transcriptionnellement comme les gènes codant des protéines. Les miRtrons sont pour leur part régulés transcriptionnellement de la même manière que leur gènes hôtes[29].
Fonctions
Les miARNs sont impliqués dans un grand nombre de fonctions physiologiques essentielles[30] telles que :
- la croissance[31] et la différenciation cellulaire[32],
- le métabolisme[34],
- etc.
- Controverse
On a pu spéculer que les miARNs obtenus par l'alimentation, à partir d'autres organismes (riz), pouvaient être retrouvés dans l'organisme hôte et influencer son métabolisme, si bien qu'ils pourraient être considérés comme une nouvelle classe de micronutriments, au même titre que les vitamines, les phytohormones et les autres phytonutriments[36],[37]. Ceci pouvait paraître d'autant plus surprenant que les acides nucléiques sont normalement hydrolysés en nucléotides (mono, di et tri) par les nucléases pancréatiques[38]. Les enzymes de la bordure en brosse (nucléosidases et phosphatases) hydrolysent à leur tour les nucléotides en bases azotées et en pentoses avant que ces derniers ne pénètrent dans les entérocytes supprimant ainsi la séquence des miARNs et par la même leur caractère informatif. L'hypothèse a été invalidée par la suite, une première étude contestant tout d'abord la reproductibilité des résultats[39] puis une seconde démontrant que l'utilisation des outils de séquençage de nouvelle génération, extrêmement sensibles, n'avaient fait que détecter une contamination des échantillons de l'hôte par des miARNs de plantes[40].
Cette controverse pourrait paraître anecdotique si elle ne traduisait pas des enjeux majeurs de la recherche scientifique à l'heure actuelle. Il y a tout d'abord un besoin de reproductibilité des expériences, ensuite, la nécessité d'établir des groupes contrôles adaptés. Par ailleurs, elle alimente un débat qui agite depuis longtemps la biologie de l'évolution et qui pourrait se résumer en un "affrontement" des théories de Charles Darwin et de Jean-Baptiste de Lamarck[41]. En bref, une des problématiques est de déterminer l'influence que peut avoir l'environnement ("circonstances externes") sur le patrimoine génétique, s'y inscrire, et dans qu'elle mesure ces influences pourraient se transmettre d'une génération à l'autre. Le débat est relancé de manière récurrente par des idéologies comme le lyssenkisme, ou par de nouvelles découvertes comme le récent développement de l'épigénétique, auquel participent les miARNs[42].
Autres rôles
Mitochondrial
Le rôle central des mitochondries dans le métabolisme cellulaire induit qu'elles soient l'objet d'une régulation fine et réactive. Les gènes importants pour la fonction mitochondriale, et localisés dans le noyau, sont la cible d'une régulation par les miARNs, notamment par miR-210 (en)[43]. D'autres liens entre miARNs et mitochondrie ont été suggérés par la découverte de miARNs localisés dans la mitochondrie et nommés "mitomiR"[44], ainsi que par la présence de la protéine AGO2[45]. De manière surprenante, il a été mis en évidence que miR-1, lors de la différentiation des cellules musculaires, pénétrait dans la mitochondrie où il stimulait la traduction de gènes mitochondriaux, au lieu de la réprimer comme on s'y serait attendu pour un miARN[46]. Plusieurs autres études sont venues confirmer la présence de miARNs dans la mitochondrie, ainsi que la présence de composants du RISC, et renforcer de cette manière l'idée selon laquelle les mitomiRs influenceraient le métabolisme mitochondrial et auraient un potentiel impact physiopathologique[47],[48]. La possibilité d'utiliser une régulation de l'expression des miARNs pour moduler directement l'homéostasie et la fonction mitochondriales a donc été proposée et un brevet déposé dans ce sens[49].
Viral
Quelques équipes ont étudié l'impact des miARNs sur le fonctionnement viral. En effet, certains miARNs des cellules hôtes seraient capables de cibler des ARNs viraux, conférant un rôle antiviral aux miARNs, ou au contraire permettraient au virus de s'accumuler dans la cellule[50]. Cette situation a été notamment décrite pour miR-122, spécifiquement exprimé dans le foie, il favoriserait la réplication de l'ARN des virus de l'hépatite B et C[51].
Pathologies
Tumorales
Une mutation ou une dérégulation de certains miARNs semble être à l'origine, directement ou indirectement, d'un grand nombre de tumeurs. Ceux qui induisent directement un mécanisme d'initiation du cancer sont qualifiés d'"oncomiRs" (en)[52]. Un même miARN, comme miR-22, semble pouvoir être tantôt oncogène, tantôt au contraire contribuer à la suppression tumorale[53]. De nombreux miARNs sont dérégulés dans les tumeurs solides de façon récurrente. Par exemple, dans les tumeurs du foie, on retrouve souvent une surexpression de miR-21 et une sous-expression de miR-122[54]. De plus, il existe des dérégulations d'expression de miARNs associées à des facteurs de risques des carcinomes hépatocellulaires[55].
Certains types de miARN sont également présents dans le plasma sanguin, comme miR-92a, et pourraient être de bons biomarqueurs de leucémies[56].
Il semble donc exister des voies de carcinogenèse spécifiques aux miARNs qui agiraient alors comme des facteurs de risques ; ceci indique tout l'intérêt des miARNs comme marqueurs de diagnostic, de pronostic voir de thérapeutique.
Oncogenèse virale
Il semblerait que certains rétrovirus au pouvoir oncogène possèdent des miARNs au sein de leur génome. Ces miARNs, suite à leur transcription, pourraient participer à une répression d'ARN viraux et/ou de la cellule hôte et ainsi participer à la progression tumorale[57]. Un défi sera donc de pouvoir mieux identifier les gènes ciblés par ces miARNs afin de mieux comprendre leur rôle et les conséquences de leur dérégulation dans le processus d'oncogenèse.
Cardiaques
Certains microARNs sont enrichis (miR-1, miR-133a, miR-499) ou exprimés de manière spécifique dans le muscle, en particulier cardiaque (miR-208a). La survenue d'un infarctus du myocarde entraînerait une libération de ceux-ci dans la circulation sanguine lors de la mort cellulaire ; leur quantification pourrait donc être utilisée comme un marqueur plasmatique précoce de la survenue de l'infarctus[58].
Du système nerveux
Certains miARNs circulants pourraient être utilisés comme biomarqueurs précoces de la maladie d'Alzheimer[59] ou de Parkinson[60].
Cibles thérapeutiques
Un certain nombre d'inhibiteurs des miARNs sont en cours de développement, ce sont essentiellement de courtes chaînes d'ARNs, des oligonucléotides antisens appelés "antagomirs", se fixant par complémentarité sur une partie du microARN cible[61]. Un vecteur qui permettrait leur administration serait un virus non pathogène qui introduirait la séquence de nucléotides dans la cellule[62].
L'administration de miARNs à but thérapeutique n'est pas simple car ces derniers sont dégradés avant qu'ils atteignent leur cible. L'utilisation d'acides nucléiques bloqués - analogues conformationnels des nucléotides mais relativement plus résistants aux nucléases - pourrait servir au développement de solutions thérapeutiques basées sur les miARNs[11].
Des tests sont ainsi en cours pour le traitement de l'hépatite C[63] et le syndrome d'Alport[64].
Notes et références
- (en) David P Bartel, « Metazoan MicroRNAs », Cell, vol. 173, no 1, , p. 20-51 (PMID 29570994, DOI 10.1016/j.cell.2018.03.006, lire en ligne [PDF], consulté le )
- (en) D. Bartel, « MicroRNAs: Target Recognition and Regulatory Functions », Cell, vol. 136, no 2, , p. 215–233 (PMID 19167326, PMCID 3794896, DOI 10.1016/j.cell.2009.01.002, lire en ligne, consulté le )
- (en) B. Kusenda, M. Mraz, J. Mayer et S. Pospisilova, « MicroRNA biogenesis, functionality and cancer relevance », Biomedical papers, vol. 150, no 2, , p. 205–215 (PMID 17426780, DOI 10.5507/bp.2006.029, lire en ligne [PDF], consulté le )
- (en) Huntzinger E et Izaurralde E, « Gene silencing by microRNAs: contributions of translational repression and mRNA decay », Nat Rev Genet., vol. 12, no 2, , p. 99-110 (PMID 21245828, DOI 10.1038/nrg2936, lire en ligne, consulté le )
- (en) Kloosterman WP et Plasterk RH., « The diverse functions of microRNAs in animal development and disease. », Dev Cell., vol. 11, no 4, , p. 441-50 (PMID 17011485, DOI 10.1016/j.devcel.2006.09.009, lire en ligne [EPUB], consulté le )
- (en) Adams BD, Kasinski AL et Slack FJ, « Aberrant regulation and function of microRNAs in cancer. », Curr Biol., vol. 24, no 16, , p. 762-76 (PMID 25137592, DOI 10.1016/j.cub.2014.06.043, lire en ligne [PDF], consulté le )
- Homo sapiens miRNAs in the miRBase at Manchester University
- (en) Bentwich I, Avniel A, Karov Y, Aharonov R, Gilad S, Barad O, Barzilai A, Einat P, Einav U, Meiri E, Sharon E, Spector Y et Bentwich Z, « Identification of hundreds of conserved and nonconserved human microRNAs. », Nat Genet., vol. 37, no 7, , p. 766-70 (PMID 15965474, DOI 10.1038/ng1590, lire en ligne, consulté le )
- (en) Lewis BP, Burge CB et Bartel DP., « Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets. », Cell, vol. 120, no 1, , p. 15-20 (PMID 15652477, DOI 10.1016/j.cell.2004.12.035, lire en ligne [PDF], consulté le )
- (en) Friedman RC, Farh KK, Burge CB et Bartel DP., « Most mammalian mRNAs are conserved targets of microRNAs. », Genome Res., vol. 19, no 1, , p. 92-105 (PMID 18955434, DOI 10.1101/gr.082701.108, lire en ligne [PDF], consulté le )
- (en) Heh-In Im et Paul J Kenny, « MicroRNAs in Neuronal Function and Dysfunction », Trends Neurosci, vol. 35, no 5, , p. 325-34 (PMID 22436491, DOI 10.1016/j.tins.2012.01.004, lire en ligne [PDF], consulté le )
- Cette publication décrit la découverte de lin-4, le premier miRNA découvert chez C.elegans.(en) Lee RC, Feinbaum RL et Ambros V, « The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. », Cell, vol. 75, no 5, , p. 843-54 (PMID 8252621, DOI 10.1016/0092-8674(93)90529-y, lire en ligne [PDF], consulté le )
- (en) Lagos-Quintana M, Rauhut R, Lendeckel W et Tuschl T, « Identification of novel genes coding for small expressed RNAs. », Science, vol. 294, no 5543, , p. 853-8 (PMID 11679670, DOI 10.1126/science.1064921, lire en ligne, consulté le )
- (en) Axtell MJ, Westholm JO et Lai EC., « Vive la différence: biogenesis and evolution of microRNAs in plants and animals. », Genome Biol., vol. 12, no 4, , p. 221 (PMID 21554756, PMCID PMC3218855, DOI 10.1186/gb-2011-12-4-221, lire en ligne [PDF], consulté le )
- (en) Shabalina SA et Koonin EV., « Origins and evolution of eukaryotic RNA interference. », Trends Ecol Evol., vol. 23, no 10, , p. 578-87 (PMID 18715673, PMCID PMC2695246, DOI 10.1016/j.tree.2008.06.005, lire en ligne [PDF], consulté le )
- Cette publication discute des processus où sont impliqués les siRNAs et les microARNs, dans le contexte de deux articles publiés dans le journal Science. (en) Baulcombe D, « DNA events. An RNA microcosm. », Science., vol. 297, no 5589, , p. 2002-3 (PMID 12242426, DOI 10.1126/science.1077906, lire en ligne, consulté le )
- (en) Wang MB et Metzlaff M., « RNA silencing and antiviral defense in plants. », Curr Opin Plant Biol., vol. 8, no 2, , p. 216-22 (PMID 15753004, DOI 10.1016/j.pbi.2005.01.006, lire en ligne, consulté le )
- (en) Denli AM, Tops BB, Plasterk RH, Ketting RF et Hannon GJ, « Processing of primary microRNAs by the Microprocessor complex. », Nature, vol. 432, no 7014, , p. 231-5 (PMID 15531879, DOI 10.1038/nature03049, lire en ligne, consulté le )
- (en) Okamura K, Hagen JW, Duan H, Tyler DM et Lai EC, « The mirtron pathway generates microRNA-class regulatory RNAs in Drosophila. », Cell, vol. 130, no 1, , p. 89-100 (PMID 17599402, DOI 10.1016/j.cell.2007.06.028, lire en ligne, consulté le )
- (en) Haussecker D, Huang Y, Lau A, Parameswaran P, Fire AZ et Kay MA., « Human tRNA-derived small RNAs in the global regulation of RNA silencing. », RNA, vol. 16, no 4, , p. 673-95 (PMID 20181738, PMCID PMC2844617, DOI 10.1261/rna.2000810, lire en ligne [PDF], consulté le )
- (en) Ender C, Krek A, Friedländer MR, Beitzinger M, Weinmann L, Chen W, Pfeffer S, Rajewsky N et Meister G., « A human snoRNA with microRNA-like functions. », Mol Cell., vol. 32, no 4, , p. 519-28 (PMID 19026782, DOI 10.1016/j.molcel.2008.10.017, lire en ligne [PDF], consulté le )
- (en) Cheloufi S, Dos Santos CO, Chong MM et Hannon GJ., « A dicer-independent miRNA biogenesis pathway that requires Ago catalysis. », Nature, vol. 465, no 7298, , p. 584-9 (PMID 20424607, PMCID 2995450, DOI 10.1038/nature09092, lire en ligne [PDF], consulté le )
- (en) Cifuentes D, Xue H, Taylor DW, Patnode H, Mishima Y, Cheloufi S, Ma E, Mane S, Hannon GJ, Lawson ND, Wolfe SA et Giraldez AJ., « A novel miRNA processing pathway independent of Dicer requires Argonaute2 catalytic activity. », Science, vol. 328, no 5986, , p. 1694-8 (PMID 20448148, PMCID 3093307, DOI 10.1126/science.1190809, lire en ligne [PDF], consulté le )
- (en) Yoda M, Cifuentes D, Izumi N, Sakaguchi Y, Suzuki T, Giraldez AJ et Tomari Y., « Poly(A)-specific ribonuclease mediates 3'-end trimming of Argonaute2-cleaved precursor microRNAs. », Cell Rep., vol. 5, no 3, , p. 715-26 (PMID 24209750, PMCID 3856240, DOI 10.1016/j.celrep.2013.09.029, lire en ligne [PDF], consulté le )
- (en) Okamura K, Phillips MD, Tyler DM, Duan H, Chou YT et Lai EC, « The regulatory activity of microRNA* species has substantial influence on microRNA and 3' UTR evolution. », Nat Struct Mol Biol., vol. 15, no 4, , p. 354-63 (PMID 18376413, DOI 10.1038/nsmb.1409, lire en ligne, consulté le )
- (en) Gruber AR, Lorenz R, Bernhart SH, Neuböck R et Hofacker IL., « The Vienna RNA websuite. », Nucleic Acids Res., vol. 36, no Web Server issue, , W70-4 (PMID 18424795, PMCID PMC2447809, DOI 10.1093/nar/gkn188, lire en ligne [PDF], consulté le )
- (en) Brodersen P, Sakvarelidze-Achard L, Bruun-Rasmussen M, Dunoyer P, Yamamoto YY, Sieburth L et Voinnet O, « Widespread translational inhibition by plant miRNAs and siRNAs. », Science, vol. 320, no 5880, , p. 1185-90 (PMID 18483398, DOI 10.1126/science.1159151, lire en ligne, consulté le )
- (en) Khraiwesh B, Arif MA, Seumel GI, Ossowski S, Weigel D, Reski R et Frank W, « Transcriptional control of gene expression by microRNAs. », Cell, vol. 140, no 1, , p. 111-22 (PMID 20085706, DOI 10.1016/j.cell.2009.12.023, lire en ligne, consulté le )
- (en) Baskerville S et Bartel DP., « Microarray profiling of microRNAs reveals frequent coexpression with neighboring miRNAs and host genes. », RNA., vol. 11, no 3, , p. 241-7 (PMID 15701730, PMCID PMC1370713, DOI 10.1261/rna.7240905, lire en ligne [PDF], consulté le )
- (en) Sayed D et Abdellatif M., « MicroRNAs in development and disease. », Physiol Rev., vol. 91, no 3, , p. 827-87 (PMID 21742789, DOI 10.1152/physrev.00006.2010, lire en ligne, consulté le )
- (en) Bueno MJ et Malumbres M., « MicroRNAs and the cell cycle. », Biochim Biophys Acta., vol. 1812, no 5, , p. 592-601 (PMID 21315819, DOI 10.1016/j.bbadis.2011.02.002, lire en ligne, consulté le )
- (en) Ivey KN et Srivastava D, « MicroRNAs as regulators of differentiation and cell fate decisions. », Cell Stem Cell, vol. 7, no 1, , p. 36-41 (PMID 20621048, DOI 10.1016/j.stem.2010.06.012, lire en ligne, consulté le )
- (en) Jovanovic M et Hengartner MO., « miRNAs and apoptosis: RNAs to die for. », Oncogene., vol. 25, no 46, , p. 6176-87 (PMID 17028597, DOI 10.1038/sj.onc.1209912, lire en ligne, consulté le )
- Pour revue, voir : (en) Rottiers V et Näär AM., « MicroRNAs in metabolism and metabolic disorders. », Nat Rev Mol Cell Biol., vol. 13, no 4, , p. 239-50 (PMID 22436747, PMCID PMC4021399, DOI 10.1038/nrm3313, lire en ligne, consulté le )
- (en) Saab BJ et Mansuy IM, « Neuroepigenetics of memory formation and impairment: the role of microRNAs. », Neuropharmacology, vol. 80, , p. 61-9 (PMID 24486712, DOI 10.1016/j.neuropharm.2014.01.026, lire en ligne, consulté le )
- (en) Anne-Marie C. Hodge, « Food We Eat Might Control Our Genes - Scientists find rice microRNA inside human cells », Scientific American, (consulté le )
- (en) Zhang L et al., « Exogenous plant MIR168a specifically targets mammalian LDLRAP1: evidence of cross-kingdom regulation by microRNA. », Cell Res., vol. 22, no 1, , p. 107-26 (PMID 21931358, DOI 10.1038/cr.2011.158, lire en ligne, consulté le )
- Elaine N. Marieb et Katja Hoehn, Anatomie et Physiologie Humaines, Montréal, Pearson ERPI, , 9e éd., 1291 p. (ISBN 978-2761369329), p. 1052
- (en) Brent Dickinson, Yuanji Zhang, Jay S Petrick, Gregory Heck, Sergey Ivashuta et William S Marshall, « Lack of detectable oral bioavailability of plant microRNAs after feeding in mice », Nature Biotechnology, vol. 31, no 11, , p. 965-7 (PMID 24213763, DOI 10.1038/nbt.2737, lire en ligne, consulté le )
- (en) Tosar JP, Rovira C, Naya H et Cayota A., « Mining of public sequencing databases supports a non-dietary origin for putative foreign miRNAs: underestimated effects of contamination in NGS. », RNA, vol. 20, no 6, , p. 754-7 (PMID 24729469, PMCID PMC4024629, DOI 10.1261/rna.044263.114, lire en ligne [PDF], consulté le )
- Article de synthèse sur les différences existants entre les théories de Darwin et de Lamarck ainsi que sur l'influence de l'épigénétique : Jean-Claude BREGLIANO, « Lamarck et Darwin : deux visions divergentes du monde vivant », sur Encyclopédie de environnement, (consulté le ).
- (en) Yao Q, Chen Y et Zhou X., « The roles of microRNAs in epigenetic regulation. », Curr Opin Chem Biol., vol. 51, , p. 11-17 (PMID 30825741, DOI 10.1016/j.cbpa.2019.01.024, lire en ligne, consulté le )
- (en) Chan SY, Zhang YY, Hemann C, Mahoney CE, Zweier JL et Loscalzo J., « MicroRNA-210 controls mitochondrial metabolism during hypoxia by repressing the iron-sulfur cluster assembly proteins ISCU1/2. », Cell Metab., vol. 10, no 4, , p. 273-84 (PMID 19808020, DOI 10.1016/j.cmet.2009.08.015, lire en ligne, consulté le )
- (en) Srinivasan H et Das S., « Mitochondrial miRNA (MitomiR): a new player in cardiovascular health. », Can J Physiol Pharmacol., vol. 93, no 10, , p. 855-61 (PMID 26030833, DOI 10.1139/cjpp-2014-0500, lire en ligne, consulté le )
- (en) Bandiera S, Rüberg S, Girard M, Cagnard N, Hanein S, Chrétien D, Munnich A, Lyonnet S et Henrion-Caude A., « Nuclear outsourcing of RNA interference components to human mitochondria. », PLoS One., vol. 6, no 6, , e20746 (PMID 21695135, DOI 10.1371/journal.pone.0020746, lire en ligne, consulté le )
- (en) Zhang X, Zuo X, Yang B, Li Z, Xue Y, Zhou Y, Huang J, Zhao X, Zhou J, Yan Y, Zhang H, Guo P, Sun H, Guo L, Zhang Y et Fu XD., « MicroRNA directly enhances mitochondrial translation during muscle differentiation. », Cell., vol. 158, no 3, , p. 607-19 (PMID 25083871, DOI 10.1016/j.cell.2014.05.047, lire en ligne, consulté le )
- Pour revue voir : (en) Geiger J et Dalgaard LT., « Interplay of mitochondrial metabolism and microRNAs. », Cell Mol Life Sci., vol. 74, no 4, , p. 631-646 (PMID 27563705, DOI 10.1007/s00018-016-2342-7, lire en ligne, consulté le )
- (en) Bandiera S, Matégot R, Girard M, Demongeot J et Henrion-Caude A., « MitomiRs delineating the intracellular localization of microRNAs at mitochondria. », Free Radic Biol Med., vol. 64, , p. 12-9 (PMID 23792138, DOI 10.1016/j.freeradbiomed.2013.06.013, lire en ligne, consulté le )
- Alexandra Henrion-Caude, Simonetta Bandiera et Stanislas Lyonnet, Methods for Adjusting Expression of Mitochondrial Genome by Microrna, (lire en ligne)
- (en) Pfeffer S et Voinnet O., « Viruses, microRNAs and cancer. », Oncogene., vol. 25, no 46, , p. 6211-9 (PMID 17028601, DOI 10.1038/sj.onc.1209915, lire en ligne, consulté le )
- (en) Girard M, Jacquemin E, Munnich A, Lyonnet S et Henrion-Caude A., « miR-122, a paradigm for the role of microRNAs in the liver. », J Hepatol., vol. 48, no 4, , p. 648-56 (PMID 18291553, DOI 10.1016/j.jhep.2008.01.019, lire en ligne, consulté le )
- (en) Esquela-Kerscher A et Slack FJ., « Oncomirs - microRNAs with a role in cancer. », Nat Rev Cancer., vol. 6, no 4, , p. 259-69 (PMID 16557279, DOI 10.1038/nrc1840, lire en ligne, consulté le )
- (en) Xiong J., « Emerging roles of microRNA-22 in human disease and normal physiology. », Curr Mol Med., vol. 12, no 3, , p. 247-58 (PMID 22300138, DOI 10.2174/156652412799218886, lire en ligne, consulté le )
- Ladeiro Y et Zucman-Rossi J., « [miRNAs in cancer: the case of liver tumors]. », Med Sci (Paris)., vol. 25, no 5, , p. 467-72 (PMID 19480827, DOI 10.1051/medsci/2009255467, lire en ligne [PDF], consulté le )
- (en) Ladeiro Y, Couchy G, Balabaud C, Bioulac-Sage P, Pelletier L, Rebouissou S et Zucman-Rossi J., « MicroRNA profiling in hepatocellular tumors is associated with clinical features and oncogene/tumor suppressor gene mutations. », Hepatology., vol. 47, no 6, , p. 1955-63 (PMID 18433021, DOI 10.1002/hep.22256, lire en ligne, consulté le )
- (en) Tanaka M, Oikawa K, Takanashi M, Kudo M, Ohyashiki J, Ohyashiki K et Kuroda M., « Down-regulation of miR-92 in human plasma is a novel marker for acute leukemia patients. », PLoS One., vol. 4, no 5, , e5532 (PMID 19440243, DOI 10.1371/journal.pone.0005532, lire en ligne, consulté le )
- (en) Rosewick N, Momont M, Durkin K, Takeda H, Caiment F, Cleuter Y, Vernin C, Mortreux F, Wattel E, Burny A, Georges M et Van den Broeke A., « Deep sequencing reveals abundant noncanonical retroviral microRNAs in B-cell leukemia/lymphoma. », Proc Natl Acad Sci U S A., vol. 110, no 6, , p. 2306-11 (PMID 23345446, DOI 10.1073/pnas.1213842110, lire en ligne, consulté le )
- (en) Wang GK, Zhu JQ, Zhang JT, Li Q, Li Y, He J, Qin YW et Jing Q, « Circulating microRNA: a novel potential biomarker for early diagnosis of acute myocardial infarction in humans. », Eur Heart J., vol. 31, no 6, , p. 659-66 (PMID 20159880, DOI 10.1093/eurheartj/ehq013, lire en ligne, consulté le )
- (en) Subodh Kumar et P Hemachandra Reddy, « Are Circulating microRNAs Peripheral Biomarkers for Alzheimer's Disease? », Biochim Biophys Acta, vol. 1862, no 9, , p. 1617-27 (PMID 27264337, DOI 10.1016/j.bbadis.2016.06.001, lire en ligne, consulté le )
- (en) Ahmad R Arshad, Siti A Sulaiman, Amalia A Saperi, Rahman Jamal, Norlinah Mohamed Ibrahim et Nor Azian Abdul Murad, « MicroRNAs and Target Genes As Biomarkers for the Diagnosis of Early Onset of Parkinson Disease », Front Mol Neurosci, vol. 10, , p. 352 (PMID 29163029, DOI 10.3389/fnmol.2017.00352, lire en ligne, consulté le )
- (en) Krützfeldt J, Rajewsky N, Braich R, Rajeev KG, Tuschl T, Manoharan M et Stoffel M., « Silencing of microRNAs in vivo with 'antagomirs'. », Nature., vol. 438, no 7068, , p. 685-9 (PMID 16258535, DOI 10.1038/nature04303, lire en ligne, consulté le )
- (en) Borel F, Kay MA et Mueller C., « Recombinant AAV as a platform for translating the therapeutic potential of RNA interference. », Mol Ther., vol. 22, no 4, , p. 692-701 (PMID 24352214, DOI 10.1038/mt.2013.285, lire en ligne, consulté le )
- (en) Janssen HL, Reesink HW, Lawitz EJ, Zeuzem S, Rodriguez-Torres M, Patel K, van der Meer AJ, Patick AK, Chen A, Zhou Y, Persson R, King BD, Kauppinen S, Levin AA et Hodges MR., « Treatment of HCV infection by targeting microRNA. », N Engl J Med., vol. 368, no 18, , p. 1685-94 (PMID 23534542, DOI 10.1056/NEJMoa1209026, lire en ligne, consulté le )
- (en) Gomez IG et al., « Anti-microRNA-21 oligonucleotides prevent Alport nephropathy progression by stimulating metabolic pathways. », J Clin Invest., vol. 125, no 1, , p. 141-56 (PMID 25415439, PMCID PMC4382246, DOI 10.1172/JCI75852, lire en ligne, consulté le )
Voir aussi
Bibliographie
- Caroline Hartmann, Fabienne Corre-Menguy, Adnane Boualem, Mariana Jovanovic, et Christine Lelandais-Brière, "Les microARN - Une nouvelle classe de régulateurs de l’expression génique", Med Sci (Paris), 2004, 20, 894–898 (lire en ligne, consulté le 16 mars 2020).
- (en) Alberts B., Johnson A., Lewis J., Raff M., Roberts K. and Walter P., Molecular Biology of the Cell, 5th edition, Garland Science, New York, 2007, pp. 493-495 (ISBN 978-0815341055).
Articles connexes
Liens externes
- Site internet de l'équipe de recherche "Impact systémique des petits ARN régulateurs" du CNRS à l'IGH - UMR9002
- (en) Animation expliquant le fonctionnement de l'interférence par ARN
- (en) Site du laboratoire de Victor Ambros à UMASS Medical School
- (en) Site du laboratoire de David Bartel au MIT
- (en) Site du laboratoire de Thomas Tuschl au Rockefeller University
- (en) La base de données miRBase

