« Biologie de synthèse » : différence entre les versions
m [Bot] : transformation de liens avec le modèle {{Lien}} en lien interne, suite à la création de l'article correspondant; changements de type cosmétique |
→Réécriture génétique : Rajout de 3 paragrpahes sur des avancées dans la réécriture génétique en citant 2 articles publiés en 2019 |
||
| Ligne 37 : | Ligne 37 : | ||
=== Le premier organisme utilisant un génome synthétique === |
=== Le premier organisme utilisant un génome synthétique === |
||
Des généticiens ont recherché quel était le plus petit organisme connu pouvant être cultivé en souche pure dans un environnement dépourvu de stress et fournissant les nutriments nécessaires. Cela était nécessaire pour pouvoir étudier, reproduire (et éventuellement utiliser ou breveter) un génome correspondant approximativement à l'ensemble minimal de gènes indispensables à la vie et à la reproduction d'une bactérie<ref name=Venter>John I. Glass, Nacyra Assad-Garcia, Nina Alperovich, Shibu Yooseph, Matthew R. Lewis, Mahir Maruf, Clyde A. Hutchison, Hamilton O. Smith et Craig Venter ''[http://www.pnas.org/content/103/2/425.full?ref=%25C4%25B0lk Essential genes of a minimal bacterium]'' ; 18 novembre 2005; [[PNAS]]</ref>. |
Des généticiens ont recherché quel était le plus petit organisme connu pouvant être cultivé en souche pure dans un environnement dépourvu de stress et fournissant les nutriments nécessaires. Cela était nécessaire pour pouvoir étudier, reproduire (et éventuellement utiliser ou breveter) un [[génome]] correspondant approximativement à l'ensemble minimal de gènes indispensables à la vie et à la reproduction d'une bactérie<ref name=Venter>John I. Glass, Nacyra Assad-Garcia, Nina Alperovich, Shibu Yooseph, Matthew R. Lewis, Mahir Maruf, Clyde A. Hutchison, Hamilton O. Smith et Craig Venter ''[http://www.pnas.org/content/103/2/425.full?ref=%25C4%25B0lk Essential genes of a minimal bacterium]'' ; 18 novembre 2005; [[PNAS]]</ref>. |
||
La bactérie correspondant le mieux à ces critères était à l'époque ''[[Mycoplasma genitalium]]'' dont le génome (482 gènes codant des protéines, avec 580 kb<ref>Séquençage déposé dans la base de donnée ''GenBank database'' (pour ''Mycoplasma genitalium G37 ATCC 33530 genomic sequence'' ; accession no. L43967).</ref>) était aussi le plus petit connu parmi toutes les espèces connues et pouvant être cultivées<ref name=Venter/>. |
La bactérie correspondant le mieux à ces critères était à l'époque ''[[Mycoplasma genitalium]]'' dont le génome (482 gènes codant des protéines, avec 580 kb<ref>Séquençage déposé dans la base de donnée ''GenBank database'' (pour ''Mycoplasma genitalium G37 ATCC 33530 genomic sequence'' ; accession no. L43967).</ref>) était aussi le plus petit connu parmi toutes les espèces connues et pouvant être cultivées<ref name=Venter/>. |
||
| Ligne 71 : | Ligne 71 : | ||
=== Mieux comprendre la biologie === |
=== Mieux comprendre la biologie === |
||
Les biologistes cherchant à connaître le fonctionnement des systèmes vivants naturels, certains ont proposé de vérifier leur compréhension actuelle du vivant en construire un exemplaire synthétique (ou une version) d’un système [[Vie|vivant]]. |
Les biologistes cherchant à connaître le fonctionnement des systèmes vivants naturels, certains ont proposé de vérifier leur compréhension actuelle du vivant en construire un exemplaire synthétique (ou une version) d’un système [[Vie|vivant]]. |
||
Le travail avant-gardiste de Michael Elowitz<ref>[http://www.elowitz.caltech.edu/ The Elowitz Lab] sur caltech.edu</ref> sur les [[répressilateur]]s<ref>[https://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=pubmed&dopt=Abstract&list_uids=10659856&query_hl=2 ''A synthetic oscillatory network of transcriptional regulators.''] Nature 2000, sur ''Pubmed''.</ref> est un exemple d’une telle approche : Elowitz avait conçu un modèle mathématique du fonctionnement d'un oscillateur génétique dans les cellules vivantes. Pour le vérifier, il construisit une molécule |
Le travail avant-gardiste de Michael Elowitz<ref>[http://www.elowitz.caltech.edu/ The Elowitz Lab] sur caltech.edu</ref> sur les [[répressilateur]]s<ref>[https://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=pubmed&dopt=Abstract&list_uids=10659856&query_hl=2 ''A synthetic oscillatory network of transcriptional regulators.''] Nature 2000, sur ''Pubmed''.</ref> est un exemple d’une telle approche : Elowitz avait conçu un modèle mathématique du fonctionnement d'un oscillateur génétique dans les cellules vivantes. Pour le vérifier, il construisit une molécule d’[[Acide désoxyribonucléique|ADN]] contenant le circuit selon son modèle, la plaça dans des bactéries et analysa le comportement du circuit. De légères différences entre ce qu’il attendait et ce qu’il observa mirent en lumière qu’une nouvelle discipline scientifique valait d’être créée. De tels travaux utilisent beaucoup de mathématiques pour prédire et [[modèle mathématique|modéliser]] la dynamique des systèmes biologiques avant de les reconstruire ou d'en construire de nouveaux expérimentalement. Une large palette de méthodes mathématiques ont été utilisées avec une précision variable, les plus utilisées pour décrire les interactions entre molécules et les réactions enzymatiques étant les [[Équation différentielle ordinaire|équations différentielles ordinaires]] et les [[Équation différentielle stochastique|équations différentielles stochastiques]]. La [[théorie des graphes]] et les réseaux [[booléens]] ont été aussi utilisés. Parmi les exemples probants, citons les travaux d’Adam Arkin<ref>[http://genomics.lbl.gov Adam Arkin home page]</ref> et Alexander van Oudenaarden<ref>[http://web.mit.edu/biophysics/ van Oudenaarden Lab]</ref> ; voir aussi le numéro spécial de ''PBS Nova'' sur la vie artificielle<ref>[https://www.pbs.org/wgbh/nova/sciencenow/3214/01.html numéro spécial de ''PBS Nova'' sur la vie artificielle]</ref>. |
||
=== Ingénierie du vivant === |
=== Ingénierie du vivant === |
||
| Ligne 91 : | Ligne 91 : | ||
==== Réécriture génétique ==== |
==== Réécriture génétique ==== |
||
Les « ''réécrivains'' » sont des biologistes de synthèse souhaitant vérifier l’idée que, puisque les systèmes biologiques naturels sont si compliqués et pas nécessairement optimaux, ils feraient mieux de reconstruire le système naturel qui nous intéresse à partir de zéro, afin de fournir des {{lang|en|texte=engineered surrogates}} (substituts artificiels) plus faciles à comprendre et avec lesquels l’interaction soit plus facile. Ils tirent leur inspiration du [[réusinage]], une procédure parfois utilisée pour améliorer les logiciels. [[Drew Endy]] et son groupe<ref>[http://mit.edu/endy/ groupe de Drew Eddy]</ref> ont effectué quelques travaux préliminaires sur la réécriture<ref>par exemple, [http://www.nature.com/msb/journal/v1/n1/full/msb4100025.html Refactoring Bacteriophage T7]</ref>. Les oligonucléotides récoltés depuis une [[puce à ADN]] fabriquée par photolithographie ou jet d’encre [[DNA chip]] combinés avec les corrections d’erreurs d’ADN permettent des changements de [[codon]]s à vaste échelle dans les systèmes génétiques pour améliorer l’[[expression génétique]] ou incorporer des amino-acides novateurs<ref>voir les [http://arep.med.harvard.edu/SBP projets de cellule de synthèse] du laboratoire de [[George McDonald Church|George Church]]</ref>. Tout comme dans l’exemple ci-dessus du T7, ceci favorise une approche ''ex nihilo'' de la synthèse. |
Les « ''réécrivains'' » sont des biologistes de synthèse souhaitant vérifier l’idée que, puisque les systèmes biologiques naturels sont si compliqués et pas nécessairement optimaux, ils feraient mieux de reconstruire le système naturel qui nous intéresse à partir de zéro, afin de fournir des {{lang|en|texte=engineered surrogates}} (substituts artificiels) plus faciles à comprendre et avec lesquels l’interaction soit plus facile. Ils tirent leur inspiration du [[réusinage]], une procédure parfois utilisée pour améliorer les logiciels. [[Drew Endy]] et son groupe<ref>[http://mit.edu/endy/ groupe de Drew Eddy]</ref> ont effectué quelques travaux préliminaires sur la réécriture<ref>par exemple, [http://www.nature.com/msb/journal/v1/n1/full/msb4100025.html Refactoring Bacteriophage T7]</ref>. Les oligonucléotides récoltés depuis une [[puce à ADN]] fabriquée par photolithographie ou jet d’encre [[DNA chip]] combinés avec les corrections d’erreurs d’ADN permettent des changements de [[codon]]s à vaste échelle dans les systèmes génétiques pour améliorer l’[[expression génétique]] ou incorporer des amino-acides novateurs<ref>voir les [http://arep.med.harvard.edu/SBP projets de cellule de synthèse] du laboratoire de [[George McDonald Church|George Church]]</ref>. Tout comme dans l’exemple ci-dessus du T7, ceci favorise une approche ''ex nihilo'' de la synthèse. |
||
===== Avancées dans la réécriture ===== |
|||
En 2019, plusieurs articles concernant la réécriture ont été publiées, notamment une sur la synthèse de quatre bases artificielles qui peuvent s'insérer dans l'ADN sans perturber sa structure thermodynamique<ref name=":0">{{Article|langue=en|prénom1=Steven A.|nom1=Benner|prénom2=Millie M.|nom2=Georgiadis|prénom3=John|nom3=SantaLucia|prénom4=Andrew D.|nom4=Ellington|titre=Hachimoji DNA and RNA: A genetic system with eight building blocks|périodique=Science|volume=363|numéro=6429|date=2019-02-22|issn=0036-8075|issn2=1095-9203|pmid=30792304|doi=10.1126/science.aat0971|lire en ligne=https://science.sciencemag.org/content/363/6429/884|consulté le=2019-05-30|pages=884–887}}</ref> ainsi qu'une autre sur le recodage entier du génome d'''[[Escherichia coli|E. coli]]''<ref name=":1">{{Article|langue=En|prénom1=Julius|nom1=Fredens|prénom2=Kaihang|nom2=Wang|prénom3=Daniel|nom3=de la Torre|prénom4=Louise F. H.|nom4=Funke|titre=Total synthesis of Escherichia coli with a recoded genome|périodique=Nature|volume=569|numéro=7757|date=2019-05|issn=0028-0836|issn2=1476-4687|doi=10.1038/s41586-019-1192-5|lire en ligne=https://www.nature.com/articles/s41586-019-1192-5.epdf?shared_access_token=krbAQSmffa6DgCBqlyYgIdRgN0jAjWel9jnR3ZoTv0NOX3G84MyIpdOYaDpXEkW47AwlE3tl-5aOPYS1CU61-TFzA3P1Hc4MPl2OA4NntaO9sZ2sDno1MAnig8iTZN312Q73AZvZSthHTpLUqqRj-g==|consulté le=2019-05-30|pages=514–518}}</ref>''.'' |
|||
La synthèse du [[ADN hachimoji|Hachimoji]] a pu démontrer que la nature dispose de plusieurs façons de fabriquer l'ADN et de stocker et transmettre l'information génétique<ref name=":0" />. Les chercheurs au Japon ont fait plusieurs expériences démontrant que l'addition de ces bases ne perturbent pas la structure de l'ADN et que les séquences contenant ces bases peuvent subir un certain taux de mutation et donc peuvent évoluer comme toute autre séquence d'ADN naturelle. De plus, cette addition artificielle permet aussi de potentiellement augmenter la densité d'informations contenues dans une molécule d'ADN, (avec plus de bases à disposition il existe plus de choix de combinaisons et donc plus d'information peut se trouver dans une même espace) ce qui pourrait donner des formes de vie encore plus complexes. Surtout intéressant pour les domaines tels que l'[[Exobiologie|astrobiologie]], cette synthèse permet d'élargir les critères de recherche de la [[vie extraterrestre]]. |
|||
Le recodage du génome complet de la bactérie ''E. coli''<ref name=":1" /> avait pour but de démontrer qu'un organisme n'a pas besoin de 64 [[Codon|codons]] (dont la plupart qui sont redondants) pour être fonctionnel. Les chercheurs ont alors réécrit tout le génome mais en n'utilisant que 61 codons, ce qui libère 3 codons déjà existants dans l'organisme qui peuvent être utilisés pour coder des [[Acide aminé|acides aminés]] synthétiques et donc la création de nouvelles protéines. Cette étude a alors montré qu'il est possible de prendre avantage de la redondance déjà existante dans le [[code génétique]] pour l'intégration d'acides aminés artificiels et que la simplification du génome n'affecte pas la survie de l'organisme. |
|||
==== Exemples d'applications ==== |
==== Exemples d'applications ==== |
||
Version du 30 mai 2019 à 11:46
La biologie de synthèse, ou biologie synthétique, est un domaine scientifique et biotechnologique émergeant qui combine biologie et principes d'ingénierie, dans le but de concevoir et construire (« synthétiser ») de nouveaux systèmes et fonctions biologiques, avec des applications notamment développées par les secteurs agropharmaceutique, chimique, agricole et énergétique.

Objectifs
Les objectifs de la biologie de synthèse sont de deux types :
- Tester et améliorer notre compréhension des principes gouvernant la biologie (apprendre en construisant).
- Construire de façon fiable des organismes accomplissant des fonctions biologiques complexes répondant à diverses applications (énergie, santé par exemple).
Concepts
Dans sa phase actuelle, la biologie de synthèse vise surtout à rendre le génie biologique plus simple et rapide, plus accessible et moins onéreux par usage extensif de principes d'ingénierie (standardisation, automation, conception assistée par ordinateur…) ayant fait leurs preuves dans d’autres domaines plus matures tels le génie civil ou l’électronique. Elle doit pour cela relever des défis d'ingénierie propres au « substrat biologique », alors que les principes de fonctionnement des systèmes biologiques ou l'évolution ne sont pas encore tous compris.
Modifier le vivant ou en détourner certaines fonctions pose aussi des questions philosophiques et éthiques nouvelles et complexes. Notamment sur le fait de savoir s'il est possible ou souhaitable d'assimiler les êtres vivants à des « machines » et avec quelles conséquences, ou concernant la question de la brevetabilité du vivant ou de ses produits, et plus généralement de la propriété intellectuelle appliquée au vivant.
Histoire
Première génération : les années 1900
- Au début du XXe siècle, les travaux de Charles Darwin sur l'évolution sont complétés par la re-découverte des lois de l'hérédité de Mendel. Certains biologistes, tels Hugo de Vries, suggèrent de contrôler et de diriger les mécanismes de l'évolution en laboratoire pour créer de nouvelles formes de vie présentant des propriétés nouvelles.
- On peut retracer l'apparition du terme lui-même à 1910 et 1912, quand Stéphane Leduc (1853–1939), un biologiste français, publia son ouvrage intitulé Théorie physico-chimique de la vie et générations spontanées (1910)[2] et La biologie de synthèse (1912), dans lequel il affirme que pour tester la validité de nos connaissances en biologie, la fabrication, ou « synthèse », doit succéder à l'analyse[3] : « Quand on est arrivé à connaître le mécanisme physique de la production d'un objet ou d'un phénomène, (...) il devient possible (...) de reproduire l'objet ou le phénomène, la science est devenue de synthèse. La biologie est une science comme les autres, (...) elle doit être successivement descriptive, analytique et de synthèse. »
- À la même époque, Jacques Loeb, un physiologiste allemand installé aux États-Unis, est sans doute le plus grand promoteur de l'émergence d'une technologie basée sur la biologie.
Naissance du génie génétique : les années 1970-1980
- La naissance de la biotechnologie « moderne » est généralement datée en 1973 par l'invention des techniques d'ADN recombinant par Stanley Cohen et Herbert Boyer.
- La première synthèse de gène en 1977 a lieu dans le laboratoire de Boyer. Ce travail donne lieu à l'insuline et à la somatostatine recombinantes.
- En 1978, le prix Nobel de médecine est décerné à Werner Arber, Daniel Nathans et Hamilton O. Smith pour la découverte des enzymes de restriction et leur application aux problèmes de génétique moléculaire. Dans un éditorial du journal Gene, Wacław Szybalski (en) écrivait : « Le travail sur les nucléases de synthèse nous permet non seulement de construire aisément les molécules d’ADN recombinant et d’analyser les gènes individuels, mais nous a aussi mené à une nouvelle ère de la biologie de synthèse où non seulement les gènes existants sont décrits et analysés, mais où aussi de nouvelles configurations génétiques peuvent être construites et évaluées. »[4].
- L'invention de la réaction en chaîne par polymérase PCR en 1984 par Kary Mullis révolutionne la biologie moléculaire et le génie génétique.
Renaissance : les années 2000
À la fin des années 90, certains informaticiens se tournant vers la biologie comme Tom Knight au MIT, sont frustrés du manque d'organisation et de méthode du domaine. S'inspirant d'autres sciences de l'ingénieur, Knight introduit le concept standard biologique en créant des BioBricks[5], des composants biologiques aux diverses fonctions pouvant être assemblés par un protocole standardisé. Durant la même période, à l'université de Berkeley, Roger Brent, Robert Carlson, Drew Endy et Adam Arkin parmi d'autres, posent les bases de ce qu'ils appellent alors biologie « intentionnelle » ou « constructive », qui deviendra biologie de synthèse par la suite. Par opposition au génie génétique contemporain qu'ils considèrent comme majoritairement aléatoire, ils prônent une approche rationnelle inspirée des méthodes de sciences de l'ingénieur plus matures pour la conception et la construction de systèmes biologiques aux fonctions prévisibles, et ce de manière robuste.
Le congrès Synthetic Biology 1.0 organisé au MIT en 2004 marquera l'acte de naissance « officiel » de la biologie de synthèse contemporaine.
Le premier organisme utilisant un génome synthétique
Des généticiens ont recherché quel était le plus petit organisme connu pouvant être cultivé en souche pure dans un environnement dépourvu de stress et fournissant les nutriments nécessaires. Cela était nécessaire pour pouvoir étudier, reproduire (et éventuellement utiliser ou breveter) un génome correspondant approximativement à l'ensemble minimal de gènes indispensables à la vie et à la reproduction d'une bactérie[6].
La bactérie correspondant le mieux à ces critères était à l'époque Mycoplasma genitalium dont le génome (482 gènes codant des protéines, avec 580 kb[7]) était aussi le plus petit connu parmi toutes les espèces connues et pouvant être cultivées[6].
Son génome présente en outre peu de redondance génomique et cette bactérie (parasite obligatoire à niche écologique restreinte) possède un métabolisme minimal[6], et elle commençait à être assez bien connue.
Les gènes essentiels du génome de cette bactéries ont été identifiés à partir de la fin des années 1990 : sur 482 gènes codant des protéines, 382 sont indispensables ; pour 28 % d'entre eux la fonction de la protéine codée est inconnue[6]. L'interruption de certains a accéléré la croissance de la bactérie[6].
- En 2007, c'est donc à partir de Mycoplasma genitalium que Craig Venter a « fabriquée » Mycoplasma laboratorium, la première bactérie dite synthétique, c'est-à-dire entièrement reconstruite par génie génétique autour d'un chromosome de synthèse (Chromosome artificiel bactérien).
Cette bactérie survit (a priori peut-être éternellement si élevée dans un environnement dépourvu de stress) avec un nombre de gènes inférieur à ce qu'on pensait antérieurement nécessaire. Mushegian and Koonin en comparant les génomes de 2 bactéries aux petits génomes - Haemophilus influenzae (Gram-negative) et Mycoplasma genitalium (gram-positive) - avaient supposé que les 256 gènes orthologues (communs à ces 2 bactéries) étaient un nombre proche du nombre minimal de gènes nécessaire à la vie bactérienne[8]. Gil et son équipe (3) avaient ensuite proposé le chiffre de 206 gènes codant des protéines, sur la base de l'analyse de plusieurs bactéries endosymbiotes.
Le caractère artificiel de cette bactérie pose de nouvelles questions de bioéthique, d'autant que cette bactérie est un parasite (urogénital) obligatoire et un pathogène pour l'homme (maladie sexuellement transmissible). - En 2010, le premier organisme contenant un génome intégralement fabriqué par l'homme est décrit dans la Revue Science. Il s'agit d'une souche de Mycoplasma capricolum (en) dont le génome a été retiré et est remplacé par le génome « JCVI-syn1.0 » conçu par l'équipe de Craig Venter, donnant naissance à une souche de Mycoplasma laboratorium de type Mycoplasma mycoides (en) pour laquelle Craig Venter a fait une demande de brevet. Ce génome a été créé par la synthèse de 1 078 oligonucléotides de 1 080 paires de bases, ces 1 078 fragments ont été assemblés en 109 fragments de 10 080 paires de bases, eux-mêmes assemblés en 11 fragments de 100 000 paires de bases finalement réunis au sein du génome circulaire de 1 077 947 paires de bases[9],[10].
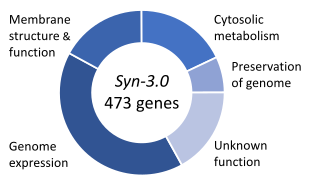
- En 2016, la même équipe a découpé le génome de Mycoplasma mycoides en différents éléments et les a ensuite réassemblés en éliminant les gènes non indispensables, par essai et erreur. Ils sont ainsi arrivés à une cellule synthétique de seulement 473 gènes, dont 41 % interviennent dans la régulation du génome, 18 % dans la structuration et le fonctionnement de la membrane, 17 % dans le métabolisme et 7 % dans la préservation du code génétique : restent 17 % de gènes apparemment indispensables mais dont on ne connaît pas la fonction[11].
En France
En 2010, l'iSSB[12] (Institute of Systems and Synthetic Biology) est le premier laboratoire de recherche en biologie de synthèse grâce à l'aide de Genopole, de l'Université d'Évry-Val-d'Essonne et du CNRS.
En 2010, le mSSB[13] (master in Systems and Synthetic Biology) est le premier master en Biologie de synthèse (université Paris-Saclay et université d'Évry-Val-d'Essonne).
Un site « Biologie de synthèse » a été ouvert par le gouvernement en 2011[14] qui promeut ce volet de la biologie, peu après qu'un rapport de l'OPECST a porté sur la faisabilité de certaines applications[15] qui porte notamment sur les « enjeux de la gouvernance, ils touchent respectivement à des positions diamétralement opposées sur l'appréciation et la gestion des risques, à l'adéquation du droit de la propriété intellectuelle et des brevets, à la question de l'opportunité d'un débat public — en particulier sur l'acceptation sociale de ces avancées — et aux problèmes de formation et de financement de la recherche »[15]
C'est à la suite des recommandations portées par deux rapports plus récents[16],[17], que l'observatoire de la biologie de synthèse[18] a été créé pour répondre au souci du ministère de l'Enseignement Supérieur et de la Recherche de suivre le développement de la biologie de synthèse et de favoriser la tenue d'un débat équilibré et argumenté au sein de la société.
Méthodes
Les méthodes de biologie moléculaire et plus généralement les concepts guidant le génie génétique n'ont pratiquement pas changé depuis les années 1970, limitant la construction de circuits génétiques complexes et rendant chaque projet de génie génétique herculéen. De plus, aucune stratégie globale d'investissement sur les technologies de base permettant la fabrication rapide et fiable de composants génétiques à bas coût ou la mise en place de librairies de composants biologiques extensivement caractérisés pouvant être réutilisés de façon générique n'a à ce jour été mise en place. De ce fait, le génie génétique s'apparente encore à de l'artisanat plutôt qu'à une véritable science de l'ingénieur. Par exemple, les concepts-clés de standardisation des composants biologiques utilisés, d'« abstraction hiérarchique » et de « découplage » devraient permettre l'utilisation de ces composants dans des systèmes synthétiques de plus en plus complexes[réf. nécessaire].
Applications
Mieux comprendre la biologie
Les biologistes cherchant à connaître le fonctionnement des systèmes vivants naturels, certains ont proposé de vérifier leur compréhension actuelle du vivant en construire un exemplaire synthétique (ou une version) d’un système vivant. Le travail avant-gardiste de Michael Elowitz[19] sur les répressilateurs[20] est un exemple d’une telle approche : Elowitz avait conçu un modèle mathématique du fonctionnement d'un oscillateur génétique dans les cellules vivantes. Pour le vérifier, il construisit une molécule d’ADN contenant le circuit selon son modèle, la plaça dans des bactéries et analysa le comportement du circuit. De légères différences entre ce qu’il attendait et ce qu’il observa mirent en lumière qu’une nouvelle discipline scientifique valait d’être créée. De tels travaux utilisent beaucoup de mathématiques pour prédire et modéliser la dynamique des systèmes biologiques avant de les reconstruire ou d'en construire de nouveaux expérimentalement. Une large palette de méthodes mathématiques ont été utilisées avec une précision variable, les plus utilisées pour décrire les interactions entre molécules et les réactions enzymatiques étant les équations différentielles ordinaires et les équations différentielles stochastiques. La théorie des graphes et les réseaux booléens ont été aussi utilisés. Parmi les exemples probants, citons les travaux d’Adam Arkin[21] et Alexander van Oudenaarden[22] ; voir aussi le numéro spécial de PBS Nova sur la vie artificielle[23].
Ingénierie du vivant
Les ingénieurs du domaine des biotechnologies voient la biologie comme une technologie de base. La biologie de synthèse inclut une large redéfinition et extension de la biotechnologie, avec le but d’être capable de concevoir et construire des systèmes biologiques fabriqués qui traitent l’information, manipulent les éléments chimiques, produisent de l’énergie ou des molécules (nouveaux polymères, aliments, médicaments...) susceptible d'améliorer l'environnement voire l'homme. Un des aspects qui distingue la biologie de synthèse du génie génétique conventionnel est sa forte attention à développer des technologies fondamentales rendant le génie biologique plus facile et plus fiable.
Dans les années 2000 -2010, trois façons de faire du neuf dans le Vivant (metabolic design pour les anglophones) semblent biotechnologiquement possibles[24] :
- à partir de l'inanimé prébiotique, c'est-à-dire en utilisant des acides aminés nouveaux construit à partir de « briques élémentaires » disponibles ou à créer.
- de manière parabiotique (à côté du vivant, en le modifiant), en considérant que les conditions initiales et l'évolution sont imparfaites et qu'on peut les améliorer. C'est notamment le cas de la xénobiologie.
- de manière post-biotique ; il s'agit alors de « corriger », « améliorer » ou transformer les créatures vivantes pour en faire de nouvelles formes de vie « utiles » et « captives » (non susceptibles de pulluler) ; c'est un des projets de la biologie de synthèse. Craig Venter a ainsi aux États-Unis lancé une entreprise de fabrication de génomes artificiels, visant la reprogrammation de cellules bactériennes par automates de synthèse d'ADN. On sait déjà « écrire » de nouvelles phrases d'ADN, mais on ne sait pas aujourd'hui prédire si ces instructions artificielles seront lues et exprimées par la cellule, ni si l'organisme ainsi modifié sera viable.
Chimie, biochimie et néocodage du vivant
Du point de vue de la chimie organique, les systèmes biologiques sont des systèmes physiques composés de matériaux chimiques. Il y a environ cent ans, la chimie passa de l’étude des matériaux chimiques naturels à la conception et l’élaboration de nouveaux matériaux chimiques. Cette transition inaugura le domaine de la chimie de synthèse. Dans le même esprit, certains aspects de la biologie de synthèse peuvent être vus comme une extension et une application de la chimie de synthèse à la biologie, et inclure des travaux allant de la création de nouveaux matériaux biochimiques jusqu’à l’étude des origines de la vie. Les groupes d’Eric Kool (en)[25] à Stanford, de Steven A. Benner (en)[26] à l’université de Floride, de Carlos Bustamante (en)[27] à Berkeley et de Jack Szostak[28] à Harvard sont de bons exemples de cette tradition. Parmi les exemples de biologie de synthèse figurent les travaux pionniers de Tim Gardner et Jim Collins (en) sur une construction d'un interrupteur génétiquement encodé[29], le registre des pièces biologiques standardisées ou « Biobriques »[30], et la compétition internationale des machines génétiquement modifiées[31].
Ingénierie métabolique
Une application phare de la biologie de synthèse est la construction de voies métaboliques responsables de la synthèse de composés d'intérêts tels médicaments, biocarburants ou polymères. Un bon exemple est le travail du laboratoire de Denis Pompon au CNRS en France qui fut le premier à réussir le transfert d'une voie métabolique de mammifères dans la levure: la voie de biosynthèse de l'hydrocortisone à partir d'un sucre simple dans le milieu de culture de la levure remaniée[32],[33] en y intégrant huit gènes de mammifères impliqués dans lavoie de biosynthèse des hormones stéroïdes et en modifiant quatre gènes de levure pour éliminer des réactions parasites induites par l'introduction et l'expression de ces gènes étrangers. Huit ans plus tard, Jay Keasling à l'université de Berkeley a reconstitué chez la levure la voie de synthèse d'un composé antimalarique, l'artemisinine, en y intégrant les gènes des enzymes de la plante produisant la substance dans la nature[34]. Ce sont, pour le moment, les deux seuls exemples de procédés d'ingénierie métaboliques industrialisés basés sur l'utilisation de la levure de boulangerie.
Réécriture génétique
Les « réécrivains » sont des biologistes de synthèse souhaitant vérifier l’idée que, puisque les systèmes biologiques naturels sont si compliqués et pas nécessairement optimaux, ils feraient mieux de reconstruire le système naturel qui nous intéresse à partir de zéro, afin de fournir des engineered surrogates (substituts artificiels) plus faciles à comprendre et avec lesquels l’interaction soit plus facile. Ils tirent leur inspiration du réusinage, une procédure parfois utilisée pour améliorer les logiciels. Drew Endy et son groupe[35] ont effectué quelques travaux préliminaires sur la réécriture[36]. Les oligonucléotides récoltés depuis une puce à ADN fabriquée par photolithographie ou jet d’encre DNA chip combinés avec les corrections d’erreurs d’ADN permettent des changements de codons à vaste échelle dans les systèmes génétiques pour améliorer l’expression génétique ou incorporer des amino-acides novateurs[37]. Tout comme dans l’exemple ci-dessus du T7, ceci favorise une approche ex nihilo de la synthèse.
Avancées dans la réécriture
En 2019, plusieurs articles concernant la réécriture ont été publiées, notamment une sur la synthèse de quatre bases artificielles qui peuvent s'insérer dans l'ADN sans perturber sa structure thermodynamique[38] ainsi qu'une autre sur le recodage entier du génome d'E. coli[39].
La synthèse du Hachimoji a pu démontrer que la nature dispose de plusieurs façons de fabriquer l'ADN et de stocker et transmettre l'information génétique[38]. Les chercheurs au Japon ont fait plusieurs expériences démontrant que l'addition de ces bases ne perturbent pas la structure de l'ADN et que les séquences contenant ces bases peuvent subir un certain taux de mutation et donc peuvent évoluer comme toute autre séquence d'ADN naturelle. De plus, cette addition artificielle permet aussi de potentiellement augmenter la densité d'informations contenues dans une molécule d'ADN, (avec plus de bases à disposition il existe plus de choix de combinaisons et donc plus d'information peut se trouver dans une même espace) ce qui pourrait donner des formes de vie encore plus complexes. Surtout intéressant pour les domaines tels que l'astrobiologie, cette synthèse permet d'élargir les critères de recherche de la vie extraterrestre.
Le recodage du génome complet de la bactérie E. coli[39] avait pour but de démontrer qu'un organisme n'a pas besoin de 64 codons (dont la plupart qui sont redondants) pour être fonctionnel. Les chercheurs ont alors réécrit tout le génome mais en n'utilisant que 61 codons, ce qui libère 3 codons déjà existants dans l'organisme qui peuvent être utilisés pour coder des acides aminés synthétiques et donc la création de nouvelles protéines. Cette étude a alors montré qu'il est possible de prendre avantage de la redondance déjà existante dans le code génétique pour l'intégration d'acides aminés artificiels et que la simplification du génome n'affecte pas la survie de l'organisme.
Exemples d'applications
Nouveaux objectifs de la biologie de synthèse appliquée : Les médias et la littérature spécialisée citent notamment :
- la construction de nouvelles voies métaboliques « sur-mesure » présentée comme nécessaire à une industrie chimique moins polluante et moins consommatrices d'énergie[24] ;
- la diversification et l'optimisation des utilisations du carbone, de l'azote, de l'hydrogène.. (en consommant moins d'énergie). 25 % du tonnage de fixation de l'azote sur la planète serait déjà anthropique (industrie, engrais) et cette activité est très consommatrice d'énergie et de ressources peu renouvelables ;
- création d'habitats industriels synthétiques et biosynthétisés : avec des organismes utilisés seuls ou collectivement, mais de manière contrôlée, pour produire des molécules ou objets[24] ;
- trouver des alternatives aux OGM actuels qui posent trop de problèmes de risques de pollution génétique. Ici les « bifurcations évolutives » fortes sont présentées comme empêchant la pollution génétique (par exemple par Philippe Marlière qui cite l'exemple du CEA à Évry qui vise la conception et création de bactéries modifiées pour capter le carbone du CO2 via le formaldéhyde, ou par d'autres voies autres que les voies métaboliques habituelles, avec l'espoir de fixer le CO2 d'une manière énergétiquement plus rentable[24].
- Certains[24] veulent — à partir du langage génétique connu, réputé « universel » — produire et rendre disponible et commercialisables des batteries d'instructions génétiques faciles à réassembler dans des organismes existants (bactéries en général, car relativement faciles à élever en bioréacteurs) ou nouveaux.
Ils voudraient utiliser l'exaptation (notion de « potentiel adaptatif » inventée par Stephen Jay Gould pour décrire la capacité du vivant à détourner au cours de l'évolution une fonction ou un organe pour un autre usage ; par exemple : la plume des dinosaures a probablement eu une fonction de protection thermique de l'animal ou de signal visuel avant de devenir la plume qui a permis la maîtrise du vol). Il existerait alors des risques significatifs et potentiellement graves de pollution génétique (voir paragraphe sur la sécurité).
Sécurité technique, environnementale et sanitaire
Cette ingénierie se présente comme pouvant peut-être contribuer à relever de grands défis environnementaux, tels que le dérèglement climatique, le manque d'eau propre, une agriculture, sylviculture optimisées mais les organismes synthétiques peuvent également présenter un risque élevé pour les écosystèmes naturels et la santé publique.
Les systèmes vivants font en effet preuve de fortes capacités d'adaptation et de mutation. Produire des formes de vies artificielles, ou des processus métaboliques inconnus dans la nature invite donc à la prudence et à l'application du principe de précaution. En termes de biosécurité, les promoteurs de la biologie de synthèse proposent généralement de combiner au moins trois types de verrous[24] ;
- le confinement trophique (ou nutritionnel) : il s'agirait de créer un organisme nécessitant des substances rares ou inconnues dans la nature pour survivre (vitamines artificielles ou catalyseurs non présent dans la nature ou n'intervenant pas ou peu dans le vivant comme le Fluor, ou le silicium[réf. nécessaire]..)
- le confinement évolutif : il s'agit de créer des organismes modifiés, recombinants (par exemple des bactéries « reprogrammées » et conçues pour être mal adaptées à la survie autonome dans la nature. Pour réduire les coûts de production, il est déjà relativement facile de massivement produire des organismes neufs recombinants (organismes reprogrammés) sans équivalents naturels. S'ils sont très différents et fortement altérés, ils sont supposés peu viables dans le milieu naturel. L'utilisation de tels organismes, dits losers (ex. : bactérie « amoindries » rendues si peu compétitives face aux formes « sauvages » qu'elles ne survivront que cultivées en condition optimale artificiellement entretenue par l'homme). Ceci a l'inconvénient d'un coût d'entretien du milieu et de la population considérés.
Philippe Marlière note cependant qu'il suffirait ensuite de soumettre cette masse d'organismes à un processus de type sélection naturelle pour sélectionner les individus et souches les plus aptes à survivre (possible avec bactéries grâce à leur taux rapide de reproduction). mais cette solution pose à nouveau la question des risques de fuites dans le milieu naturel d'un organisme ou d'un élément génomique devenu « compétitif », voire supercompétitif (« gagnant » ou winner) . - le confinement sémantique (grille de lecture génétique, changement de codons). Certains proposent de créer un nouveau code génétique avec des gènes et/ou des bases (parce que synthétiques) ne pouvant se mélanger avec notre ADN ou celui d'organismes sauvages. Une autre piste est de produire des organismes artificiels ne pouvant pas spontanément échanger de gènes entre eux ou avec d'autres[24].
On a aussi envisagé pour les OGM classiques de construire des organismes programmés pour se suicider ou ne pas pouvoir se reproduire (ex : gène terminator, inhibition de la reproduction...).
Dans les conditions scientifiques et techniques actuelles, aucun de ces trois « verrous » n'offre à lui seul de protection absolue, certaine ou définitive[24], car on ne peut exclure d'adaptations inattendues à la suite de mutations spontanées, ou échanges de gènes avec d'autres organismes sauvages, artificiels ou modifiés. Selon les promoteurs de leur utilisation la combinaison de ces trois barrières pourrait renforcer la sécurité face à des phénomènes d'adaptation biologique (un peu à la manière d'une trithérapie, mais même la trithérapie a ses limites puisqu'un variant semblant résistant du virus HIV/SIDA est assez rapidement apparu en Amérique du Nord à la de suite la trithérapie développée pour bloquer les adaptations de ce virus à ARN, qui comme celui de la grippe ne cesse de muter).
Les applications biotechnologiques nouvelles intègrent de plus en plus de précautions[réf. nécessaire], car les adaptations récentes et parfois spectaculaires de nombreux microbes aux antibiotiques, de plantes aux désherbants, de champignons aux fongicides, d'insectes aux insecticides) et les problèmes nosocomiaux croissant et persistant ont montré ou confirmé les puissantes capacités évolutives et adaptatives du vivant. Par ailleurs, le confinement total peut rarement être garanti, surtout si ces usages se généralisent.
Technologies clés
Il s'agit notamment des techniques de lecture et d'écriture de l'ADN (séquençage et fabrication), dont les prix et performances s'améliorent de façon exponentielle (Kurzweil, 2001).
Des mesures précises du comportement des systèmes synthétiques (dans diverses conditions) sont également nécessaires à la modélisation précise et au contrôle des systèmes biologiques
La conception et modélisation assistées par ordinateur (CAO...) sont aussi au cœur de la biologie synthétique.
Séquençage
Les biologistes de synthèse utilisent les méthodes de séquençage de l'ADN de plusieurs façons : d'abord en utilisant les résultats du séquençage de génomes à grande échelle qui sont une mine d'informations sur les organismes d'origine naturelle. Les biologistes de synthèse en tirent des modèles et molécules les aidant à construire des composants biologiques et des circuits génétiques. Les biologistes de synthèse utilisent aussi le séquençage de manière extensive pour vérifier l'exactitude de la séquence d'ADN des systèmes qu'ils fabriquent. Enfin un séquençage rapide, économique et fiable peut aussi faciliter la détection et l'identification rapides des systèmes et d'organismes de synthèse.
Fabrication
La biologie de synthèse est aujourd'hui limitée par le temps et les efforts (et donc le coût) associés a la fabrication de séquences d'ADN composant les circuits génétiques. Pour accélérer le cycle de conception, fabrication, d'essais et de reconception, la synthèse de novo d'ADN et l'assemblage de fragments d'ADN, processus communément appelés la synthèse de gènes, doivent devenir plus rapides, plus fiables, et moins onéreux.
En 2002 après 2 ans de travail, des chercheurs du SUNY Stony Brook ont annoncé avoir synthétisé les bases de la séquence publiée du génome du poliovirus (7741 pb), qui est le premier organisme synthétique déclaré[40]. En 2003, le génome du bactériophage Phi X 174 (5386 pb) a été assemblé en environ deux semaines par l'Institut de Craig Venter[41]. En 2006, la même équipe a construit et breveté le génome synthétique d'une nouvelle « bactérie minimale » Mycoplasma laboratorium et travaille à obtenir son fonctionnement dans une cellule vivante[42],[43]
En 2007, plusieurs sociétés offraient la synthèse des séquences génétiques jusqu'à 2000 paires de bases, pour un prix d'environ 1 $ par paire de bases et d'un temps de fabrication de moins de deux semaines[44]. En 2009 le prix a chuté à moins de 0,50 $ par paire de base tout en étant plus rapide. Ces prix sont jugés inférieurs au coût du clonage par des techniques conventionnelles, et le système commun de tarification des entreprises rend pratique et économiquement intéressant pour les chercheurs la conception et la commande de plusieurs variantes de la même séquence pour identifier des gènes ou des protéines avec des performances optimisées (meilleur usage de codons selon l'organisme par exemple).
Modélisation
Les modèles mathématiques aident à concevoir de circuits génétiques et des systèmes biologiques en permettant de mieux prédire le comportement du système et de l'optimiser avant sa fabrication. La biologie de synthèse cherche améliorer les modèles de réactions enzymatiques et d'interactions moléculaires, et à mieux comprendre le comportement des systèmes multi-composants. Récemment, des modèles multi-échelle de circuits génétiques mettant l'accent sur les applications de la biologie de synthèse ont été développés. Des simulations aident à modéliser les interactions biomoléculaires en jeu dans les processus de transcription, de traduction, et diverses régulation, et guident la conception de systèmes synthétiques[45].
Caractérisation
Des mesures quantitatives précises du comportement des systèmes biologiques sont essentielles pour mieux comprendre le fonctionnement des systèmes biologiques et appuyer la production de modèles et leur validation. Les différences entre les prévisions du modèle et les mesures expérimentales du système aident à identifier les lacunes dans la compréhension et à expliquer pourquoi les systèmes biosynthétiques ont souvent un comportement imprévu. Des technologies de mesures cinétiques en parallèle complètent utilement la microscopie et la cytométrie de flux. La microfluidique devrait bientôt permettre de faire un grand nombre de mesures en parallèle, à moindre coût et plus vite.
Questions sociales et éthiques
En plus des nombreux défis techniques, le vaste potentiel de la biologie de synthèse inquiète aussi les bioéthiciens quant à ses potentiels abus par des États voyous et/ou des terroristes[46],[47] ou simplement par inconscience. Tout comme la physique nucléaire a mené aux traitements des cancers par radiation, mais aussi aux armes nucléaires, la biologie de synthèse pourrait améliorer la lutte contre des maladies endémiques telles que la malaria, qui tue tous les ans des millions de personnes, mais aussi mener par exemple à un organisme vivant nouveau dans la biosphère et contre lequel le système immunitaire humain ou animal pourrait être impuissant (si cet organisme vivant s'avère virulent). Un organisme non-dangereux pour la santé humaine peut qu'il devient invasif en certains milieux dégrader ou détruire des écosystèmes vitaux pour l'humanité.
Certains promoteurs et généticiens utilisant ces techniques avancent qu'ils peuvent ou pourraient utiliser divers moyens de contention et de contrôle des nouveaux organismes ou gènes qu'ils créeraient et des suggestions détaillées apparaissent pour les licences et la surveillance[48] des diverses phases de la synthèse des gènes et du génome, mais d'autres généticiens (et une partie de la population) n'ont pas confiance dans ces systèmes de contrôle ou de défense, et craignent des accidents ou des abus. Des discussions en ligne sont en cours, détaillées et ouvertes, sur les problèmes sociétaux, par exemple à l'OpenWetWare[49]. Certains auteurs[46] estiment que la bioéthique doit élargir son champ à ces applications et préoccupations nouvelles, en examinant non seulement le moyen par lequel la connaissance scientifique est produite, mais aussi les types de connaissances à rechercher, diffuser et débattre.
De nombreux romans ou films de science fiction, et le jeu de rôles Transhuman Space ont popularisé le concept d'androïdes pour partie artificiels et de bioroïdes (introduits par (GURPS Cyberpunk), des androïdes biologiques créés par biogenèse (mot employé par le jeu de rôle pour désigner la biologie de synthèse)[pertinence contestée]
Le projet européen SYNBIOSAFE[50] porte sur les aspects éthiques et de sécurité de la biologie de synthèse, alors que déjà sont développés des projets de création d'acide nucléique artificiel.
En 2009, le président de la Commission européenne (José Barsoso) a demandé un avis du Groupe européen d'éthique des sciences et nouvelles technologies (GEE) sur l'éthique de la biologie de synthèse (GEE, 2009)[51], en précisant dans sa requête que « le débat sur la légitimité de nouvelles formes de vie a porté principalement sur les questions de sécurité, et une étude sur les implications morales, légales et sociales qui peuvent dériver de cette utilisation spécifique de la biotechnologie manque toujours. », mais, l'Europe soutient aussi financièrement un projet dit « 3NA » (acronyme anglais de « 3e forme d'acide nucléique ») de création d'un acide nucléique artificiel différent de l'ADN et de l'ARN. Le CEA y participe avec des laboratoires anglais et belges. Les promoteurs de ce projet estiment qu'il s'agit aussi d'un moyen de rendre les biotechnologies plus sûres que les OGM actuels, en limitant les risques qu'ils contaminent l'environnement, d'autres espèces ou des espèces parentes[24].
Le sous-domaine de l'alimentation est également questionné. La méthionine, par exemple, très utilisée dans l'alimentation animale, fait l'objet d'une attention particulière de la part des promoteurs des biotechnologies qui cherchent à la synthétiser à moindre coût[52], avec un soutien financier et stratégique de l'Europe[53] et non sans susciter des craintes en matière d'éthique environnementale[54], en raison des risques[55] lié aux biotechnologies et d'une demande sociale inexistante, voire opposée à l'artificialisation de la production de nourriture[56],[57].
Notes et références
- (en) Clyde A. Hutchison, Ray-Yuan Chuang, Vladimir N. Noskov, Nacyra Assad-Garcia, Thomas J. Deerinck, Mark H. Ellisman, John Gill, Krishna Kannan et Bogumil J. Karas, « Design and synthesis of a minimal bacterial genome », Science, vol. 351, no 6280, , aad6253 (ISSN 0036-8075, PMID 27013737, DOI 10.1126/science.aad6253, lire en ligne)
- Théorie physico-chimique de la vie et générations spontanées, S. Leduc, 1910
- La biologie de synthèse, étude de biophysique, S. Leduc, 1912
- Gene 1978, 4, p. 181
- En anglais, ce néologisme s'écrit avec deux majuscules. Voir en:BioBrick.
- John I. Glass, Nacyra Assad-Garcia, Nina Alperovich, Shibu Yooseph, Matthew R. Lewis, Mahir Maruf, Clyde A. Hutchison, Hamilton O. Smith et Craig Venter Essential genes of a minimal bacterium ; 18 novembre 2005; PNAS
- Séquençage déposé dans la base de donnée GenBank database (pour Mycoplasma genitalium G37 ATCC 33530 genomic sequence ; accession no. L43967).
- Fraser, C. M., Gocayne, J. D., White, O., Adams, M. D., Clayton, R. A., Fleischmann, R. D., Bult, C. J., Kerlavage, A. R., Sutton, G., Kelley, J. M., et al. (1995) The Minimal Gene Complement of Mycoplasma genitalium Science 270, 397-403. Abstract/FREE Full Text
- (en) Daniel G. Gibson, John I. Glass, Carole Lartigue, Vladimir N. Noskov, Ray-Yuan Chuang, Mikkel A. Algire, Gwynedd A. Benders, 2 Michael G. Montague, Li Ma, Monzia M. Moodie, Chuck Merryman, Sanjay Vashee, Radha Krishnakumar, Nacyra Assad-Garcia, Cynthia Andrews-Pfannkoch, Evgeniya A. Denisova, Lei Young, Zhi-Qing Qi, Thomas H. Segall-Shapiro, Christopher H. Calvey, Prashanth P. Parmar, Clyde A. Hutchison, III, Hamilton O. Smith, J. Craig;, « Creation of a Bacterial Cell Controlled by a Chemically Synthesized Genome », Science, (DOI 10.1126/science.1190719, lire en ligne)
- Hervé Morin, « Création d'une cellule "synthétique" », sur Le Monde.fr,
- Sean Bailly, « Le génome réduit à sa plus simple expression », Pour la science, no 463, , p. 6-7.
- Site officiel de l'iSSB
- Site officiel du mSSB
- Science.gouv, Lancement du site "Biologie de synthèse", 22/06/2011
- Sénat, OPECST, Geneviève Fioraso, députée, rapporteure, Les enjeux de la biologie de synthèse - Présentation de l'étude de faisabilité, 15/02/11
- Biologie de synthèse : conditions d'un dialogue avec la société, étude de l’Institut Francilien Recherche Innovation Société (IFRIS), commandée par le Ministère de l'Enseignement supérieur et de la Recherche.
- Biologie de synthèse : développements, potentialités et défis, rapport du groupe de travail créé au MESR par le secteur scientifique « bio-ressources, écologie, agronomie » et animé par François Képès.
- Site officiel de l'observatoire de la biologie de synthèse
- The Elowitz Lab sur caltech.edu
- A synthetic oscillatory network of transcriptional regulators. Nature 2000, sur Pubmed.
- Adam Arkin home page
- van Oudenaarden Lab
- numéro spécial de PBS Nova sur la vie artificielle
- Conférence filmée faite pour l'Université de tous les savoirs et intitulée « Comment et pourquoi faire des formes de vie nouvelle »
- Eric Kool
- Steven Benner
- Carlos Bustamante
- Jack Szostak
- construction d'un interrupteur génétiquement encodé
- registre des pièces biologiques standardisées
- intercollegiate Genetically Engineering Machine competition (iGEM)
- Duport C et al. Nature Biotechnology (1998) Feb 18(2): 186-189. Self-sufficient biosynthesis of pregnenolone and progesterone in engineered yeast
- Szezbara F.M. et al. Nature Biotechnology (2003) Feb 21(2): 143-149. Total biosynthesis of hydrocortisone from a simple carbon source in yeast
- Production of the antimalarial drug precursor artemisinic acid in engineered yeast. Ro et al., Nature 440, 940-943 (13 avril 2006)
- groupe de Drew Eddy
- par exemple, Refactoring Bacteriophage T7
- voir les projets de cellule de synthèse du laboratoire de George Church
- (en) Steven A. Benner, Millie M. Georgiadis, John SantaLucia et Andrew D. Ellington, « Hachimoji DNA and RNA: A genetic system with eight building blocks », Science, vol. 363, no 6429, , p. 884–887 (ISSN 0036-8075 et 1095-9203, PMID 30792304, DOI 10.1126/science.aat0971, lire en ligne, consulté le )
- (en) Julius Fredens, Kaihang Wang, Daniel de la Torre et Louise F. H. Funke, « Total synthesis of Escherichia coli with a recoded genome », Nature, vol. 569, no 7757, , p. 514–518 (ISSN 0028-0836 et 1476-4687, DOI 10.1038/s41586-019-1192-5, lire en ligne, consulté le )
- Couzin J (2002). "Virology. Active poliovirus baked from scratch". Science. 297 (5579): 174–5. doi:10.1126/science.297.5579.174b . .
- Smith, Hamilton O.; Hutchison, Clyde A.; Pfannkoch, Cynthia; Venter, J. Craig (2003). "Generating a Synthetic Genome by Whole Genome Assembly: ΦX174 Bacteriophage from Synthetic Oligonucleotides" . Proceedings of the National Academy of Sciences. 100 (26): 15440–5. Bibcode:2003PNAS..10015440S . doi:10.1073/pnas.2237126100 . JSTOR 3149024 . PMC 307586 free to read.
- (en) Nicholas Wade, « Scientists Transplant Genome of Bacteria », The New York Times, (ISSN 0362-4331, lire en ligne, consulté le )
- (en) DG Gibson, Benders GA, Andrews-Pfannkoch C, Denisova EA, Baden-Tillson H, Zaveri J, Stockwell TB, Brownley A, Thomas DW, Algire MA, Merryman C, Young L, Noskov VN, Glass JI, Venter JC, Hutchison CA 3rd et Smith HO, « Complete chemical synthesis, assembly, and cloning of a Mycoplasma genitalium genome », Science, vol. 319, no 5867, , p. 1215–20 (PMID 18218864, DOI 10.1126/science.1151721)
- Pollack, Andrew (2007-09-12). "How Do You Like Your Genes ? Biofabs Take Orders". The New York Times. ISSN 0362-4331 . Consulté 2007-12-28.
- Y. N. Kaznessis, (2007) "Models for Synthetic Biology", BMC Systems Biology, 2007, 1:47 doi:10.1186/1752-0509-1-47 [1]
- Thomas Douglas et Julian Savulescu ; Global medical ethics Synthetic biology and the ethics of knowledge J Med Ethics 2010;36:687-693 doi:10.1136/jme.2010.038232 ; résumé
- (New Scientist, 12 novembre 2005)
- A Synthetic Biohazard Non-proliferation Proposal harvard.edu
- OpenWetWare
- SYNBIOSAFE
- CTA, La biologie de synthèse et l’éthique : bâtir la confiance publique, 05/05/2011
- Gouvernement français, 2011 Investissements d'avenir - Biotechnologies et bioressources - PROJET SYNTHACS Ce projet propose une alternative à l’utilisation de la pétrochimie pour produire, à partir de biomasse renouvelable, la méthionine, l’un des composants essentiels de l’alimentation animale.
- EUROPE - DG RECHERCHE - Biotechnologies, Agriculture and Food -2009< 19th Meeting of the EC-US Task Force on Biotechnology ResearchAu sommaire: The importance of Synthetic Biology activities in a EuropeanBiotechnology Programme
- CONFEDERATION SUISSE Biologie synthétique - Biologie synthétique : réflexions éthiques, rapport de la CENH Mai 2010
- EUROPE - DG ENVIRONNEMENT >- Synthetic biology ethics: managing risks without limiting benefits, mars 2011]
- BBSRC New report reveals public’s views on synthetic biology, 14/06/2010
- VIVAGOVEILLE Au sommaire : Ne pas ouvrir la boîte de Pandore de la biologie synthétique, juin 2010
Voir aussi
Articles connexes
Bibliographie
- Sénat français, Les enjeux de la biologie synthétique - présentation de l'étude de faisabilité, Comptes rendus de l'Office parlementaire d'évaluation des choix scientifiques et technologiques ; 15/02/2011
- Commission européenne, (2016) Synthetic biology and biodiversity ; Science for Environment Policy (2016) ; Future Brief 15. Produced for the European Commission DG Environment by the Science Communication Unit, UWE, Bristol. (http://ec.europa.eu/science-environment-policy), PDF, 36 pages
Liens externes
- Entretiens vidéo avec des experts et des ONG (en anglais)
- L'ABC de la biologie de synthèse, CNRS, 2015
- Conférence filmée Comment et pourquoi faire des formes de vie nouvelle, de Philippe Marlière, l'un des promoteurs de la biologie de synthèse (vidéo de 79 min)
- Commentaire critique de l'article de François Képès, “La biologie de synthèse : vers une ingénierie du vivant”, paru dans le magazine scientifique Pour la Science no 440 de juin 2014: “François Képès, rationalisateur des machines vivantes”.
- Site de l'Observatoire français de la biologie de synthèse (créé en 2012)
- Site gouvernemental français biologie de synthèse (créé en 2011)
