Phyla bactériens
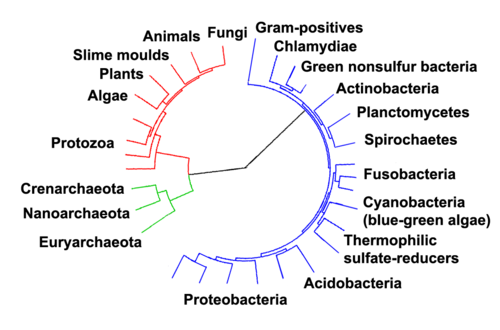
Les phyla bactériens constituent les principaux taxons, au niveau du phylum, du domaine des Eubacteria.
Dans la classification scientifique du vivant établie par Linné[2], chaque souche bactérienne est assignée à une espèce (nomenclature binominale), correspondant à un niveau inférieur dans la hiérarchie des groupes taxonomiques. À l'heure actuelle, le système de méga-classification le plus accepté dans la communauté scientifique correspond au modèle à trois domaines, basé sur la phylogénétique moléculaire. Dans ce système, les bactéries font partie du domaine Bacteria[3] et le "phylum" est le rang taxinomique inférieur au domaine, maintenant que le rang "règne" est devenu désuet dans la taxonomie bactérienne actuelle[4],[A 1]. Lorsque la nomenclature bactérienne était gérée par le Code Botanique, on utilisait le terme "division", mais aujourd'hui la nomenclature bactérienne (à l'exception des cyanobactéries) est gérée par le Code Bactériologique et on préfère le terme « phylum ».
Dans ce schéma de classification, les Bacteria sont (non-officiellement)[A 2] subdivisées en 30 phyla avec chacun au moins un représentant cultivé en laboratoire[5],[6],[7]. Chaque clade majeur de bactéries ne pouvant pas, à l'heure actuelle, être cultivé en laboratoire, est connu seulement et de façon assez indirecte par la métagénomique, c'est-à-dire l'analyse d'échantillons en vrac issus de l'environnement. Si l'on inclut les phyla candidats dans ces clades possibles, le nombre de phyla atteint 52 voire plus. Ainsi, le nombre de phyla principaux n'a cessé d'augmenter, partant de 12 en 1987, atteignant 30 en 2014, et dépassant 50 si l'on inclut les phyla candidats[8]. Le nombre total a été estimé à plus de 1000 phyla bactériens, dont plus de la moitié reste donc à découvrir[9].
À la base du clade Bacteria, près du dernier ancêtre commun universel de tous les êtres vivants, certains scientifiques pensent qu'il y aurait un ordre d'embranchement défini, alors que d'autres scientifiques, comme Norman Pace, pensent qu'il y avait au départ une grande polytomie, c'est-à-dire un événement de spéciations multiples simultanées[10].
Phylogénétique moléculaire
[modifier | modifier le code]Historiquement, la phylogénie était déduite et la taxonomie établie à partir des études morphologiques des êtres vivants. Récemment, le développement de la phylogénétique moléculaire a été utilisée pour permettre une meilleure élucidation de la relation évolutive des espèces en analysant leurs séquences d'ADN et de protéines, par exemple leur ADN ribosomal[11]. L'absence de caractéristiques morphologiques facilement accessibles, comme celles présentes chez les animaux et les plantes, a entravé les premiers efforts de classification et a donné lieu à une classification erronée, déformée et confuse, comme l'a noté Carl Woese avec l'exemple de l'étymologie attribuée au genre Pseudomonas qui correspond ironiquement à sa taxonomie, soit "fausse unité"[12].
Première subdivision
[modifier | modifier le code]
En 1987, Carl Woese, considéré comme le précurseur de la révolution en phylogénie moléculaire, a divisé les Eubacteria en 11 divisions basées sur les séquences d'ARN ribosomal 16S (SSU)[12],[14] :
- bactéries pourpres et apparentées (plus tard renommées Proteobacteria[15])
- sous-division alpha (bactéries pourpres non-sulfureuses, rhizobacteria, Agrobacterium, Rickettsiae, Nitrobacter)
- sous-division beta (Rhodocyclus, certaines Thiobacillus, Alcaligenes, Spirillum, Nitrosovibrio)
- sous-division gamma (bactéries entériques, pseudomonadales fluorescentes, bactéries pourpres sulfureuses, Legionella, certaines Beggiatoa)
- sous-division delta (sulfo- et sulfato-réductrices comme Desulfovibrio, myxobactéries, Bdellovibrio)
- bactéries à Gram positif[A 3]
- espèces à fort taux en GC (plus tard renommées Actinobacteria[16]) comme Actinomyces, Streptomyces, Arthrobacter, Micrococcus, Bifidobacterium
- espèces à faible taux en GC (plus tard renommées Firmicutes[16]) comme Clostridium, Peptococcus, Bacillus, Mycoplasma
- espèces photosynthétiques (Heliobacteria)
- espèces à Gram négatif avec paroi cellulaire (Megasphaera, Sporomusa)
- Cyanobactéries et chloroplastes (Aphanocapsa, Oscillatoria, Nostoc, Synechococcus, Gloeobacter, Prochloron)
- Spirochètes et apparentés
- Spirochètes (Spirochaeta, Treponema, Borrelia)
- Leptospiras (Leptospira, Leptonema)
- bactéries vertes sulfureuses (Chlorobium, Chloroherpeton)
- Bacteroides, Flavobacteria et apparentés (plus tard renommés Bacteroidetes)
- Bacteroides (Bacteroides, Fusobacterium)
- groupe des Flavobacterium (Flavobacterium, Cytophaga, Saprospira, Flexibacter)
- Planctomyces et apparentés (plus tard renommés Planctomycetes)
- groupes des Planctomyces (Planctomyces, Pasteuria [sic][A 4])
- Thermophiles (Isocystis pallida)
- Chlamydiae (Chlamydia psittaci, Chlamydia trachomatis)
- Micrococci radiorésistants et apparentés (qu'on appelle aujourd'hui communément Deinococcus-Thermus[7] ou Thermi)[A 5]
- groupe Deinococcus (Deinococcus radiodurans)
- Thermophiles (Thermus aquaticus)
- bactéries vertes non sulfureuses et apparentées (plus tard renommées Chloroflexi[17])
- groupe des Chloroflexus (Chloroflexus, Herpetosiphon)
- groupe des Thermomicrobium (Thermomicrobium roseum)
- Thermotogae (Thermotoga maritima)
Nouveaux phyla cultivés
[modifier | modifier le code]De nouvelles espèces ont été cultivées avec succès depuis 1987 (date de la publication de la méta-analyse de Woese), qui sont suffisamment différentes pour garantir leur appartenance à un nouveau phylum. La plupart sont thermophiles et souvent également chimiolithoautotrophes, comme les Aquificae, qui oxydent l'hydrogène gazeux. D'autres non thermophiles, comme les Acidobacteria, un phylum ubiquitaire avec des physiologies divergentes, ont été découvertes, dont certaines sont chimiolithotrophes, comme Nitrospira (qui oxyde le nitrile) ou Leptospirillum (qui oxyde le fer)[8] ; certain phyla proposés n'apparaissent cependant pas dans la base de données LPSN car ils ne sont pas suffisamment décrits ou attendent d'être validés, ou encore suscitent un débat pour savoir s'ils n'appartiennent pas à un phylum déjà existant. Par exemple le genre Caldithrix, comprenant les espèces C. palaeochoryensis[18] et C. abyssi[19], est considéré comme appartenant aux Deferribacteres[7]. Cependant, il ne partage que 81% de similarité avec les autres Deferribacteres (les espèces appartenant au genre Deferribacter et apparentées) et n'est considéré comme un phylum à part entière que par Rappé et Giovannoni[8]. De plus, la place du genre Geovibrio dans le phylum Deferribacteres est sujet à débat[7].
Phyla non cultivés et métagénomique
[modifier | modifier le code]Avec l'avènement de méthodes pour analyser l'ADN environnemental (la métagénomique), l'ARNr 16S d'un très grand nombre d'espèces non découvertes a été analysé, prouvant qu'il y a plusieurs phyla entiers qui n'ont pas de représentant cultivable connu et qu'au sein de certains phyla il manque des représentants cultivables de subdivisions majeures telles que les Verrucomicrobia et les Chloroflexi[8]. Le terme Candidatus est employé pour les propositions de nouvelles espèces pour lesquelles le manque d'information à leur sujet les empêche d'être validées, comme lorsque les seules informations dont on dispose sont des données de séquence ADN, même si le génome entier a été séquencé[20],[21]. Lorsque les espèces en question sont membres d'un phylum entier, on appelle ce dernier une division candidate (ou un phylum candidat), et en 2003 cela concernait 26 des 52 phyla découverts[8]. En 1998, Hungenholtz et Pace définissent un phylum candidat comme un groupe de séquences d'ARNr 16S partageant moins de 85% de similarité avec les phyla déjà existants et décrits[22]. Plus récemment, on a proposé de diminuer ce seuil de similarité à 75%[9]. Avant 1998 on connaissait trois phyla candidats, avant l'établissement du seuil à 85% défini par Hungenholtz et Pace :
- le groupe OS-K (provenant de la source Octopus du Parc National du Yellowstone)
- le Groupe Marin A (provenant de l'océan Pacifique)
- le Groupe Termite 1 (provenant du tube digestif du termite Reticulitermes speratus, aujourd'hui classifié dans les Elusimicrobia)[23]
Depuis lors, plusieurs autres phyla candidats ont été identifiés[8] :
- OP1, OP3, OP5 (aujourd'hui Caldiserica), OP8, OP9 (aujourd'hui Atribacteria)[23], OP10 (aujourd'hui Armatimonadetes), OP11 (bassin en obsidienne, Parc National du Yellowstone)
- WS2, WS3, WS5, WS6 (aquifère contaminé de Wurtsmith)
- SC3 et SC4 (provenant de sol aride)
- VadinBE97 (aujourd'hui Lentisphaerae)
- NC10 (provenant de grottes inondées, rizières, zones intertidales etc)
- BRC1 (provenant de sol brut et racines de riz)
- ABY1 (provenant de sédiments)
- Guyamas1 (provenant de cheminées hydrothermales)
- GN01, GN02, GN04 (provenant d'un tapis microbien hypersalin à Guerrero Negro)
- NKB19 (provenant de boue activée)
- SBR1093 (provenant de boue activée)
- TM6 et TM7 (Torf, Mittlere Schicht, littéralement "couche tourbe, intermédiaire")
Depuis lors, un phylum candidat appelé Poribacteria a été découvert, vivant en symbiose avec les éponges et faisant l'objet de nombreuses recherches actuellement[24],[A 6]. Un autre phylum candidat, les Tectomicrobia, a également été découvert, vivant en symbiose avec les éponges[25]. Enfin, on a proposé que Nitrospina gracilis, ayant été pendant longtemps sans affiliation phylogénétique, appartienne à un nouveau phylum, celui des Nitrospinae[26].
Parmi les autres phyla candidats faisant l'objet de recherches, on peut citer le TM7[27], les génomes d'organismes ayant été séquencés (draft genomes en anglais)[28], le WS6[29] et le Groupe Marin A[8].
Deux espèces du phylum candidat OP10, aujourd'hui appelé Armatimonadetes, ont été récemment cultivées avec succès : Armatimonas rosea isolée du rhizoplane d'un roseau d'un lac japonais[30], et Chthonomonas calidirosea provenant d'un isolat d'un sol géothermiquement chauffé à la Porte des Enfers (Tikitere, Nouvelle-Zélande)[31].
Une espèce du phylum candidat OP5, Caldisericum exile, a également été cultivée avec succès, ce qui a donné lieu à l'appellation de ce phylum Caldiserica[32].
Le phylum candidat VadinBE97 est désormais connu sous le nom de Lentispherae depuis que les espèces Lentisphaera araneosa et Victivallis vadensis ont été cultivées avec succès[32].
Plus récemment, on a donné à plusieurs phyla candidats des noms provisoires malgré le fait qu'ils n'aient pas de représentants encore cultivables[33] :
- le phylum candidat ACD58 a été renommé Berkelbacteria[34]
- le phylum candidat CD12 (également connu comme phylum candidat BHI80-139) a été renommé Aerophobetes
- le phylum candidat EM19 a été renommé Calescamantes
- le phylum candidat GN02 (également connu comme phylum candidat BD1-5) a été renommé Gracilibacteria
- le phylum candidat KSB3 a été renommé Modulibacteria[35]
- le phylum candidat NKB19 a été renommé Hydrogenedentes
- le phylum candidat OctSpa1-106 a été renommé Fervidibacteria
- le phylum candidat OD1 a été renommé Parcubacteria
- le phylum candidat OP1 a été renommé Candidatus Acetothermia puis renommé Candidatus Acetithermota
- le phylum candidat OP3 a été renommé Omnitrophica
- le phylum candidat OP8 a été renommé Aminicenantes
- le phylum candidat OP9 (également connu comme phylum candidat JS1) a été renommé Atribacteria
- le phylum candidat OP11 a été renommé Microgenomates
- le phylum candidat PER a été renommé Perigrinibacteria[36]
- le phylum candidat SAR406 (également connu comme phylum candidat Groupe Marin A) a été renommé Marinimicrobia
- le phylum candidat SR1 a été renommé Absconditabacteria[37]
- le phylum candidat TM6 a été renommé Dependentiae[38]
- le phylum candidat TM7 a été renommé Saccharibacteria[39]
- le phylum candidat WS3 a été renommé Latescibacteria
- le phylum candidat WWE1 a été renommé Cloacimonetes
- le phylum candidat WWE3 a été renommé Katanobacteria[37]
- le phylum candidat ZB1 a été renommé Ignavibacteriae[40]
Malgré ces lignages non officiellement reconnus (en raison de l'augmentation sans cesse du nombre de séquences appartenant à des phyla non décrits), la base de données Arb-Silva liste 67 phyla, dont 37 phyla candidats (Acetothermia, Aerophobetes, Aminicenantes, aquifer1, aquifer2, Atribacteria, Calescamantes, CKC4, Cloacimonetes, GAL08, GOUTA4, Gracilibacteria, Fermentibacteria (Hyd24-12)[41], Hydrogenedentes, JL-ETNP-Z39, Kazan-3B-09, Latescibacteria, LCP-89, LD1-PA38, Marinimicrobia, Microgenomates, OC31, Omnitrophica, Parcubacteria, PAUC34f, RsaHF231, S2R-29, Saccharibacteria, SBYG-2791, SHA-109, SM2F11, SR1, TA06, TM6, WCHB1-60, WD272, and WS6)[42] ; le Ribosomal Database Project 10, liste 49 phyla, dont 20 phyla candidats (Acetothermia, Aminicenantes, Atribacteria, BRC1, Calescamantes, Cloacimonetes, Hydrogenedentes, Ignavibacteriae, Latescibacteria, Marinimicrobia, Microgenomates, Nitrospinae, Omnitrophica, Parcubacteria, Poribacteria, SR1, Saccharibacteria, WPS-1, WPS-2, and ZB3)[43] ; enfin, le NCBI liste 120 phyla, dont 90 phyla candidats (AC1, Acetothermia, Aerophobetes, Aminicenantes, Atribacteria, Berkelbacteria, BRC1, CAB-I, Calescamantes, CPR1, CPR2, CPR3, EM 3, Fervidibacteria, GAL15, GN01, GN03, GN04, GN05, GN06, GN07, GN08, GN09, GN10, GN11, GN12, GN13, GN14, GN15, Gracilibacteria, Fermentibacteria (Hyd24-12), Hydrogenedentes, JL-ETNP-Z39, KD3-62, kpj58rc, KSA1, KSA2, KSB1, KSB2, KSB3, KSB4, Latescibacteria, Groupe Marin A, Marinimicrobia, Microgenomates, MSBL2, MSBL3, MSBL4, MSBL5, MSBL6, NC10, Nitrospinae, NPL-UPA2, NT-B4, Omnitrophica, OP2, OP4, OP6, OP7, OS-K, Parcubacteria, Peregrinibacteria, Poribacteria, RF3, Saccharibacteria, SAM, SBR1093, Sediment-1, Sediment-2, Sediment-3, Sediment-4, SPAM, SR1, TA06, TG2, TM6, VC2, WOR-1, WOR-3, WPS-1, WPS-2, WS1, WS2, WS4, WS5, WS6, WWE3, WYO, ZB3, and Zixibacteria)[44].
Superphyla
[modifier | modifier le code]Malgré le manque de clarté de l'ordre d'embranchement pour la plupart des phyla bactériens, plusieurs groupes de phyla possèdent un regroupement clair et sont appelés "superphyla".
Sphingobacteria
[modifier | modifier le code]Les Sphingobacteria (anciennement appelé « groupe des FCB ») comprend les Bacteroidetes, le genre non classé Caldithrix, les Chlorobi, le phylum candidat Cloacimonetes, les Fibrobacteres, les Gemmatimonadales, le phylum candidat Ignivibacteriae, le phylum candidat Latescibacteria, le phylum candidat Marinimicrobia et le phylum candidat Zixibacteria[33],[35].
Planctobacteria
[modifier | modifier le code]Les Planctobacteria (anciennement appelé « groupe des PVC ») comprend les Chlamydiae, les Lentisphaerae, le phylum candidat Omnitrophica, les Planctomycetes, le phylum candidat Poribacteria, et les Verrucomicrobia[33],[35].
Patescibacteria
[modifier | modifier le code]Le superphylum proposé des Patescibacteria comprend le phylum candidat Gracilibacteria, les Microgenomates, les Parcubacteria et les Saccharibacteria[33],[35], et probablement les Dependentiae[38]. On a récemment proposé que ces mêmes phyla candidats, ainsi que les phyla candidats Berkelbacteria, CPR2, CPR3, Kazan, Perigrinibacteria, SM2F11, WS6, et WWE3, appartiennent au groupe plus large des CPR[36]. Pour compliquer les choses, il a été suggéré que plusieurs de ces phyla seraient en fait eux-mêmes des superphyla (voir la section sur les superhyla cryptiques ci-dessous).
Terrabacteria
[modifier | modifier le code]Le superphylum proposé des Terrabacteria[45] comprend les Actinobacteria, Cyanobacteria, Deinococcus–Thermus, Chloroflexi, Firmicutes, et le phylum candidat OP10[33],[35],[45],[46].
Proteobacteria comme superphylum
[modifier | modifier le code]Il a été proposé que plusieurs classes du phylum des Proteobacteria soient classifiées comme des phyla à part entière, ce qui ferait des Proteobacteria un superphylum[9]. Il a récemment été proposé que la classe des Epsilonproteobacteria soit combinée avec l'ordre des Desulfurellales pour créer le nouveau phylum des Epsilonbacteraeota[47].
Superphyla cryptiques
[modifier | modifier le code]Il a été suggéré que plusieurs phyla candidats (Microgenomates, Omnitrophica, Parcubacteria et Saccharibacteria) et plusieurs phyla acceptés (Elusimicrobia, Caldiserica, et Armatimonadetes) soient en fait des superphyla ayant été incorrectement décrits comme phyla parce qu'il n'existe pas de règles définissant un phylum bactérien. Par exemple, il a été suggéré que le phylum candidat des Microgenomates soit en fait un superphylum qui englobe 28 phyla subordonnés et que le phylum des Elusimicrobia soit en fait un superphylum qui englobe 7 phyla subordonnés[9].
Vue d'ensemble des phyla actuels
[modifier | modifier le code]En , il existait 30 phyla appartenant au domaine Bacteria et acceptés par la LPSN[7]. Il n'existe pas de règles définies pour la nomenclature des phyla bactériens. Il a été proposé que le suffixe "-bacteria" soit utilisé dans chaque nom de phylum[48], mais en général le nom d'un phylum est le pluriel du genre-type, à l'exception des Firmicutes, Cyanobacteria et Proteobacteria dont le nom ne provient pas d'un nom de genre (alors que par exemple le nom Actinobacteria provient du genre Actinomyces).
Acidobacteria
[modifier | modifier le code]Les Acidobacteria (toutes à Gram négatif) constituent le phylum bactérien le plus abondants dans la plupart des sols, mais ses membres sont en majorité encore non cultivés. De plus, ils sont phénotypiquement assez divers et comprennent non seulement des acidophiles, mais aussi des non acidophiles[49]. Généralement, ses membres ont une division lente, un métabolisme lent en conditions de faibles apports en nutriments, et peuvent tolérer des fluctuations dans le niveau d'hydratation du sol dans lesquels ils vivent[49].
Actinobacteria
[modifier | modifier le code]Les Actinobacteria constituent un phylum[50] composé de bactéries qui sont toutes à Gram positif, dont beaucoup sont des productrices notables de métabolites secondaires. Il n'existe que deux phyla comprenant uniquement des bactéries à Gram positif, le second étant les Firmicutes ; les Actinobacteria possèdent généralement un taux de GC plus élevé et sont parfois appelé "bactéries à Gram positif à fort GC". Parmi les genres ou espèces notables on trouve Streptomyces (producteur d'antibiotiques), Cutibacterium acnes (commensale de la peau odorante) et Propionibacterium freudenreichii (vivant dans les trous de l'emmental).
Aquificae
[modifier | modifier le code]Les Aquificae[51] (à Gram négatif) contiennent seulement 14 genres (dont Aquifex et Hydrogenobacter). Les espèces sont hyperthermophiles et chimiolithotrophes (sulfureuses). D'après certaines études, cela pourrait être un des phyla les plus anciens.
Armatimonadetes
[modifier | modifier le code]Ce phylum de bactéries à Gram négatif comprend les espèces-type Armatimonas rosea[30] et Chthonomonas calidirosea[31].
Bacteroidetes
[modifier | modifier le code]Les Bacteroidetes[52] (à Gram négatif) font partie du superphylum des FCB ou Sphingobacteria. Certaines espèces sont des pathogènes opportunistes, alors que d'autres sont les bactéries commensales du tube digestif humain les plus communes. Elles sont devenues célèbres en dehors de la communauté scientifique à travers le mythe urbain de la poudre bactérienne pour perdre du poids[53].
Caldiserica
[modifier | modifier le code]Ce phylum était préalablement connu sous le nom de phylum candidat OP5. Il a pour unique représentant l'espèce Caldisericum exile[32].
Chlamydiae
[modifier | modifier le code]Le phylum des Chlamydiae[54] (à Gram négatif faible) appartient au superphylum des PVC ou Planctobacteria. Il se compose à l'heure actuelle de seulement 6 genres correspondant à des pathogènes intracellulaires obligatoires ayant un cycle de vie complexe. Parmi les espèces on trouve Chlamydia trachomatis (qui cause la chlamydiose)[55].
Chlorobi
[modifier | modifier le code]Les Chlorobi[56] font partie du superphylum des Sphingobacteria. On y trouve seulement 7 genres de bactéries photoautotrophes anaérobes obligatoires, appelées familièrement bactéries vertes sulfureuses. Le cœur de réaction photosynthétique chez les Chlorobi et les Chloroflexi (un autre groupe photosynthétique, voir ci-dessous) est formé par une structure appelée chlorosome[57] par opposition aux phycobilisomes des cyanobactéries (qui sont également photosynthétiques).
Chloroflexi
[modifier | modifier le code]Les Chloroflexi[58] constituent un phylum hétérogène comprenant des thermophiles et des halorespireurs ; on les appelle communément bactéries vertes non-sulfureuses.
Ce phylum[59] comprend seulement trois espèces : Chrysiogenes arsenatis[60], Desulfurispira natronophila[61] et Desulfurispirillum alkaliphilum[62].
Cyanobacteria
[modifier | modifier le code]Également connues sous le nom de (micro-) algues bleues ou algues vertes, les cyanobactéries[63] constituent le principal clade photosynthétique dont on pense qu'il a causé l'oxygénation de l'atmosphère terrestre[64].
On y trouve des bactéries en forme de bâtonnets à Gram négatif, basant leur métabolisme en anaérobiose à partir de fer, de manganèse ou de nitrate. Elles peuvent également produire de l'énergie par la fermentation. Le genre-type est Deferribacter.
Les espèces communément admises au sein de ce phylum sont Deinococcus radiodurans (polyextrêmophile[67]) et Thermus aquaticus[68] ; on utilise la polymérase de cette dernière dans les PCR[69].
Dictyoglomi
[modifier | modifier le code]Ce phylum comprend des bactéries à Gram négatif, thermophiles et chimioorganotrophes, dont le genre type Dictyoglomus[70] suscite un intérêt tout particulier parce qu'il produit l'enzyme xylanase capable de digérer le xylane, un hétéropolymère du xylose. On utilise cette enzyme dans le pré-traitement de la pulpe de bois lors de la fabrication du papier[71] car elle permet d'obtenir des niveaux de blancheur comparables au traitement classique en utilisant beaucoup moins de chlorine.
Elusimicrobia
[modifier | modifier le code]Ce phylum correspond à l'ancien phylum candidat Groupe Termite 1[71]. Ce sont des symbiontes du tube digestif des termites et des fourmis. L'espèce-type est Elusimicrobium minutum[23].
Membre du superphylum des Sphingobacteria, ce phylum comprend la plupart des bactéries habitant le rumen des ruminants, leur permettant de dégrader la cellulose des plantes qu'ils ingèrent[73]. Le genre Fibrobacter (seul genre connu du phylum à ce jour) a été retiré en 1988 du genre Bacteroides auquel il appartenait avant[74].
Ce phylum[75] comprend des espèces à faible taux de GC, Gram positif et formant pour la plupart d'entre elles des spores. On les répartit en 3 classes : les Bacilli[76] comprenant par exemple le genre Bacillus[77] (le pathogène B. anthracis et B. subtilis utile en biotechnologie, etc), les bactéries lactiques acides[78] (comme Lactobacillus casei utilisée dans le yaourt, Oenococcus oeni utilisée en fermentation malolactique, ou encore le pathogène Streptococcus pyogenes) et la classe des Clostridia[7] composée principalement d'espèces saprophytes sulfato-réductrices, dont le genre Clostridium (avec les pathogènes C. difficile, C. tetani, C. botulinum et la bactérie utilisée en biotechnologie C. acetobutylicum).
Ce phylum[79] comprend des bacilles à Gram négatif non sporulantes anaérobes obligatoires. Découvertes dès le XIXe siècle[80], on y trouve de nombreux pathogènes causant des nécroses tissulaires, un travail prématuré et des ulcères tropicaux.
Membre du superphylum des Sphingobacteria, il doit son nom à l'espèce-type Gemmatimonas aurantiaca qui compose environ 2% des bactéries du sol et faisant partie du top 9 des phyla trouvés dans le sol[81].
Anciennement clade VadinBE97[82], ce phylum fait partie du superphylum des Planctobacteria.
On y trouve de nombreux genres mais le plus représenté est Nitrospira.
Membre du superphylum des Planctobacteria, on y trouve des bactéries aquatiques issues de saumures, de mer et d'eau douce. Elles se reproduisent par bourgeonnement. Le métabolisme de l'anammox ne se retrouve que chez des bactéries appartenant à ce phylum[85].
Ce phylum contient la majorité des espèces connues[5], telles que Escherichia coli et Pseudomonas aeruginosa.
Les bactéries appartenant à ce phylum sont connues pour leur compartimentalisation, leur forme caractéristique hélicoïdale enroulée[86] et on y trouve des espèces comme Borrelia burgdorferi qui cause la maladie de Lyme[87].
Les membres de ce phylum sont à Gram négatif avec deux parois, anaérobes obligatoires, en forme de bâtonnets ; certains sont des commensaux humains[88].
Tenericutes
[modifier | modifier le code]On y trouve la classe des Mollicutes, qu'on associait auparavant au phylum des Firmicutes[89]. Malgré leurs parents à Gram positif à une seule paroi, les bactéries de ce groupe ne possèdent pas de peptidoglycane. Un genre notable est Mycoplasma[86].
C'est un phylum[90] composé de seulement trois genres appartenant à la même famille des Thermodesulfobacteriaceae : Caldimicrobium, Thermodesulfatator et Thermodesulfobacterium[91]. Les membres de ce phylum sont des thermophiles sulfato-réducteurs.
On y trouve des bactéries vertes non sulfureuses thermophiles vivant dans des environnements généralement éloignés des activités humaines[93]. Elles constituent des sujets de recherche intéressants car elles sont capables d'avoir une croissance améliorée en présence d'antibiotiques et d'oxyder le monoxyde de carbone[94].
Thermotogae
[modifier | modifier le code]Les Thermotogae[95] constituent un phylum dont les membres possèdent une enveloppe externe inhabituelle qu'on appelle la toga et sont pour la plupart des fermenteurs hyperthermophiles anaérobes obligatoires.
Ce phylum appartient au superphylum des Planctobacteria. Tout comme les espèces du genre Planctomycetes, ses membres possèdent plan cellulaire compartimenté avec un nucléoïde condensé et les ribosomes du pirellulosome (englobé par la membrane intracytoplasmique) et le paryphoplasme forment un compartiment entre la membrane intracytoplasmique et la membrane plasmique[96].
Ordre d'embranchement
[modifier | modifier le code]L'ordre d'embranchement des phyla bactériens n'est pas clair[10]. Diverses études arrivent à différents résultats en raison des différents jeux de données et méthodes utilisés. Par exemple, dans les études utilisant le 16S et quelques autres séquences, les Thermotogae et les Aquificae apparaissent comme les phyla les plus anciens[97], alors que dans d'autres études phylogénétiques, Firmicutes est le phylum le plus ancien[98].
Voir aussi
[modifier | modifier le code]Notes
[modifier | modifier le code]- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Bacterial phyla » (voir la liste des auteurs).
- Des éditions précédentes de Brock Biology of Microorganisms appelaient les phyla des "règnes".
- Pour des raisons historiques, les taxons supérieurs au rang de classe ne sont pas soumises au règles du Code Bactériologique (révision de 1990), et en conséquence il n'existe pas de nomenclature "officielle", mais il existe plusieurs autorités dans le domaine, comme le Manuel de Bactériologie Systématique de Bergey, qui contient un plan taxonomique, et la revue scientifique International Journal of Systematic Bacteriology/International Journal of Systematic and Evolutionary Microbiology (IJSB/IJSEM), dans laquelle on trouve le dépôt de la Liste des Noms de Procaryotes ayant le Statut de Nomenclature en Règle (LPSN en anglais).
- Jusqu'à récemment, on a pensé que seuls les Firmicutes et les Actinobacteria étaient à Gram positif. Cependant, le phylum candidat TM7 pourrait également être à Gram positif. En revanche, les Chloroflexi possèdent une bicouche unique mais apparaissent Gram négatif (à quelques exceptions près).
- Pasteuria est maintenant assignée au phylum des Bacilli et non plus au phylum des Planctomycetes.
- Il a été proposé d'appeler ce clade Xenobacteria ou Hadobacteria (ce dernier nom est considéré illégitime).
- La divergence des principales lignées bactériennes est antérieure à celles des éponges.
Références
[modifier | modifier le code]- Francesca D. Ciccarelli, Tobias Doerks, Christian von Mering et Christopher J. Creevey, « Toward automatic reconstruction of a highly resolved tree of life », Science (New York, N.Y.), vol. 311, no 5765, , p. 1283–1287 (ISSN 1095-9203, PMID 16513982, DOI 10.1126/science.1123061, lire en ligne, consulté le )
- (la) Carl von Linné, Systemae Naturae, sive regna tria naturae, systematics proposita per classes, ordines, genera & species, (lire en ligne)
- C. R. Woese, O. Kandler et M. L. Wheelis, « Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya », Proceedings of the National Academy of Sciences of the United States of America, vol. 87, no 12, , p. 4576–4579 (ISSN 0027-8424, PMID 2112744, DOI 10.1073/pnas.87.12.4576, lire en ligne, consulté le )
- (en) pubmeddev et no authors, « International Code of Nomenclature of Bacteria: Bacteriological Code, 1990 Revision - PubMed - NCBI », sur www.ncbi.nlm.nih.gov (consulté le )
- (en) M. Madigan et J. Martinko, Brock Biology of Microorganisms, San Francisco, Pearson/Benjamin Cummings, , 1061 p. (ISBN 978-0-13-232460-1)
- (en) G. M. Garrity, T. G. Lilburn, J. R. Cole, S. H. Harrison, J. Euzéby et B. J. Tindall, « Taxonomic Outline of the Bacteria and Archaea, Release 7.7 », sur taxonomicoutline.org, Taxonomic Outline of Bacteria and Archaea,
- J. P. Euzéby, « List of Bacterial Names with Standing in Nomenclature: a Folder Available on the Internet », International Journal of Systematic and Evolutionary Microbiology,, vol. 47, no 2, , p. 590–592 (ISSN 1466-5026, DOI 10.1099/00207713-47-2-590, lire en ligne, consulté le )
- Michael S. Rappé et Stephen J. Giovannoni, « The uncultured microbial majority », Annual Review of Microbiology, vol. 57, , p. 369–394 (ISSN 0066-4227, PMID 14527284, DOI 10.1146/annurev.micro.57.030502.090759, lire en ligne, consulté le )
- Pablo Yarza, Pelin Yilmaz, Elmar Pruesse et Frank Oliver Glöckner, « Uniting the classification of cultured and uncultured bacteria and archaea using 16S rRNA gene sequences », Nature Reviews. Microbiology, vol. 12, no 9, , p. 635–645 (ISSN 1740-1534, PMID 25118885, DOI 10.1038/nrmicro3330, lire en ligne, consulté le )
- Norman R. Pace, « Mapping the tree of life: progress and prospects », Microbiology and molecular biology reviews: MMBR, vol. 73, no 4, , p. 565–576 (ISSN 1098-5557, PMID 19946133, PMCID 2786576, DOI 10.1128/MMBR.00033-09, lire en ligne, consulté le )
- G. J. Olsen, C. R. Woese et R. Overbeek, « The winds of (evolutionary) change: breathing new life into microbiology », Journal of Bacteriology, vol. 176, no 1, , p. 1–6 (ISSN 0021-9193, PMID 8282683, DOI 10.1128/jb.176.1.1-6.1994, lire en ligne, consulté le )
- C. R. Woese, « Bacterial evolution », Microbiological Reviews, vol. 51, no 2, , p. 221–271 (ISSN 0146-0749, PMID 2439888, lire en ligne, consulté le )
- F. Schluenzen, A. Tocilj, R. Zarivach et J. Harms, « Structure of functionally activated small ribosomal subunit at 3.3 angstroms resolution », Cell, vol. 102, no 5, , p. 615–623 (ISSN 0092-8674, PMID 11007480, DOI 10.1016/s0092-8674(00)00084-2, lire en ligne, consulté le )
- (en) L. Holland, « Carl Woese In Forefront Of Bacterial Evolution Revolution », sur The Scientist Magazine®, (consulté le )
- E. Stackebrandt, R. G. E. Murray et H. G. Trüper, « Proteobacteria classis nov., a Name for the Phylogenetic Taxon That Includes the “Purple Bacteria and Their Relatives” », International Journal of Systematic and Evolutionary Microbiology,, vol. 38, no 3, , p. 321–325 (ISSN 1466-5026, DOI 10.1099/00207713-38-3-321, lire en ligne, consulté le )
- ERKO STACKEBRANDT, FRED A. RAINEY et NAOMI L. WARD-RAINEY, « Proposal for a New Hierarchic Classification System, Actinobacteria classis nov. », International Journal of Systematic and Evolutionary Microbiology,, vol. 47, no 2, , p. 479–491 (ISSN 1466-5026, DOI 10.1099/00207713-47-2-479, lire en ligne, consulté le )
- (en) D. R. Boone et R. W. Castenholz, The Archaea and the Deeply Branching and Phototrophic Bacteria, New York, Springer, , 2e éd., 722 p. (ISBN 978-0-387-98771-2, lire en ligne), p. 721
- Margarita L. Miroshnichenko, Tatyana V. Kolganova, Stefan Spring et Nikolay Chernyh, « Caldithrix palaeochoryensis sp. nov., a thermophilic, anaerobic, chemo-organotrophic bacterium from a geothermally heated sediment, and emended description of the genus Caldithrix », International Journal of Systematic and Evolutionary Microbiology, vol. 60, no Pt 9, , p. 2120–2123 (ISSN 1466-5026, PMID 19854873, DOI 10.1099/ijs.0.016667-0, lire en ligne, consulté le )
- (en) pubmeddev, « Caldithrix abyssi gen. nov., sp. nov., a nitrate-reducing, thermophilic, anaerobic bacterium isolated from a Mid-Atlantic Ridge hydrothermal vent, ... - PubMed - NCBI », sur www.ncbi.nlm.nih.gov (consulté le )
- R. G. Murray et K. H. Schleifer, « Taxonomic notes: a proposal for recording the properties of putative taxa of procaryotes », International Journal of Systematic Bacteriology, vol. 44, no 1, , p. 174–176 (ISSN 0020-7713, PMID 8123559, DOI 10.1099/00207713-44-1-174, lire en ligne, consulté le )
- Wilhelm Frederiksen, « Judicial Commission of the International Committee on Systematic Bacteriology », International Journal of Systematic and Evolutionary Microbiology,, vol. 45, no 1, , p. 195–196 (ISSN 1466-5026, DOI 10.1099/00207713-45-1-195, lire en ligne, consulté le )
- P. Hugenholtz, C. Pitulle, K. L. Hershberger et N. R. Pace, « Novel division level bacterial diversity in a Yellowstone hot spring », Journal of Bacteriology, vol. 180, no 2, , p. 366–376 (ISSN 0021-9193, PMID 9440526, lire en ligne, consulté le )
- Oliver Geissinger, Daniel P. R. Herlemann, Erhard Mörschel et Uwe G. Maier, « The Ultramicrobacterium “Elusimicrobium minutum” gen. nov., sp. nov., the First Cultivated Representative of the Termite Group 1 Phylum », Applied and Environmental Microbiology, vol. 75, no 9, , p. 2831–2840 (ISSN 0099-2240, PMID 19270135, PMCID 2681718, DOI 10.1128/AEM.02697-08, lire en ligne, consulté le )
- Lars Fieseler, Matthias Horn, Michael Wagner et Ute Hentschel, « Discovery of the novel candidate phylum "Poribacteria" in marine sponges », Applied and Environmental Microbiology, vol. 70, no 6, , p. 3724–3732 (ISSN 0099-2240, PMID 15184179, DOI 10.1128/AEM.70.6.3724-3732.2004, lire en ligne, consulté le )
- Micheal C. Wilson, Tetsushi Mori, Christian Rückert et Agustinus R. Uria, « An environmental bacterial taxon with a large and distinct metabolic repertoire », Nature, vol. 506, no 7486, , p. 58–62 (ISSN 1476-4687, PMID 24476823, DOI 10.1038/nature12959, lire en ligne, consulté le )
- Sebastian Lücker, Boris Nowka, Thomas Rattei et Eva Spieck, « The Genome of Nitrospina gracilis Illuminates the Metabolism and Evolution of the Major Marine Nitrite Oxidizer », Frontiers in Microbiology, vol. 4, , p. 27 (ISSN 1664-302X, PMID 23439773, PMCID 3578206, DOI 10.3389/fmicb.2013.00027, lire en ligne, consulté le )
- P. Hugenholtz, B. M. Goebel et N. R. Pace, « Impact of culture-independent studies on the emerging phylogenetic view of bacterial diversity », Journal of Bacteriology, vol. 180, no 18, , p. 4765–4774 (ISSN 0021-9193, PMID 9733676, lire en ligne, consulté le )
- Yann Marcy, Cleber Ouverney, Elisabeth M. Bik et Tina Lösekann, « Dissecting biological "dark matter" with single-cell genetic analysis of rare and uncultivated TM7 microbes from the human mouth », Proceedings of the National Academy of Sciences of the United States of America, vol. 104, no 29, , p. 11889–11894 (ISSN 0027-8424, PMID 17620602, PMCID 1924555, DOI 10.1073/pnas.0704662104, lire en ligne, consulté le )
- M. A. Dojka, J. K. Harris et N. R. Pace, « Expanding the known diversity and environmental distribution of an uncultured phylogenetic division of bacteria », Applied and Environmental Microbiology, vol. 66, no 4, , p. 1617–1621 (ISSN 0099-2240, PMID 10742250, DOI 10.1128/aem.66.4.1617-1621.2000, lire en ligne, consulté le )
- Hideyuki Tamaki, Yasuhiro Tanaka, Hiroaki Matsuzawa et Mizuho Muramatsu, « Armatimonas rosea gen. nov., sp. nov., of a novel bacterial phylum, Armatimonadetes phyl. nov., formally called the candidate phylum OP10 », International Journal of Systematic and Evolutionary Microbiology, vol. 61, no Pt 6, , p. 1442–1447 (ISSN 1466-5034, PMID 20622056, DOI 10.1099/ijs.0.025643-0, lire en ligne, consulté le )
- Kevin C.-Y. Lee, Peter F. Dunfield, Xochitl C. Morgan et Michelle A. Crowe, « Chthonomonas calidirosea gen. nov., sp. nov., an aerobic, pigmented, thermophilic micro-organism of a novel bacterial class, Chthonomonadetes classis nov., of the newly described phylum Armatimonadetes originally designated candidate division OP10 », International Journal of Systematic and Evolutionary Microbiology, vol. 61, no Pt 10, , p. 2482–2490 (ISSN 1466-5034, PMID 21097641, DOI 10.1099/ijs.0.027235-0, lire en ligne, consulté le )
- Koji Mori, Kaoru Yamaguchi, Yayoi Sakiyama et Tetsuro Urabe, « Caldisericum exile gen. nov., sp. nov., an anaerobic, thermophilic, filamentous bacterium of a novel bacterial phylum, Caldiserica phyl. nov., originally called the candidate phylum OP5, and description of Caldisericaceae fam. nov., Caldisericales ord. nov. and Caldisericia classis nov », International Journal of Systematic and Evolutionary Microbiology, vol. 59, no Pt 11, , p. 2894–2898 (ISSN 1466-5026, PMID 19628600, DOI 10.1099/ijs.0.010033-0, lire en ligne, consulté le )
- Christian Rinke, Patrick Schwientek, Alexander Sczyrba et Natalia N. Ivanova, « Insights into the phylogeny and coding potential of microbial dark matter », Nature, vol. 499, no 7459, , p. 431–437 (ISSN 1476-4687, PMID 23851394, DOI 10.1038/nature12352, lire en ligne, consulté le )
- Kelly C. Wrighton, Cindy J. Castelle, Michael J. Wilkins et Laura A. Hug, « Metabolic interdependencies between phylogenetically novel fermenters and respiratory organisms in an unconfined aquifer », The ISME journal, vol. 8, no 7, , p. 1452–1463 (ISSN 1751-7370, PMID 24621521, PMCID 4069391, DOI 10.1038/ismej.2013.249, lire en ligne, consulté le )
- Yuji Sekiguchi, Akiko Ohashi, Donovan H. Parks et Toshihiro Yamauchi, « First genomic insights into members of a candidate bacterial phylum responsible for wastewater bulking », PeerJ, vol. 3, , e740 (ISSN 2167-8359, PMID 25650158, PMCID 4312070, DOI 10.7717/peerj.740, lire en ligne, consulté le )
- Christopher T. Brown, Laura A. Hug, Brian C. Thomas et Itai Sharon, « Unusual biology across a group comprising more than 15% of domain Bacteria », Nature, vol. 523, no 7559, , p. 208–211 (ISSN 1476-4687, PMID 26083755, DOI 10.1038/nature14486, lire en ligne, consulté le )
- Laura A. Hug, Brett J. Baker, Karthik Anantharaman et Christopher T. Brown, « A new view of the tree of life », Nature Microbiology, vol. 1, , p. 16048 (ISSN 2058-5276, PMID 27572647, DOI 10.1038/nmicrobiol.2016.48, lire en ligne, consulté le )
- Yun Kit Yeoh, Yuji Sekiguchi, Donovan H. Parks et Philip Hugenholtz, « Comparative Genomics of Candidate Phylum TM6 Suggests That Parasitism Is Widespread and Ancestral in This Lineage », Molecular Biology and Evolution, vol. 33, no 4, , p. 915–927 (ISSN 1537-1719, PMID 26615204, PMCID 4776705, DOI 10.1093/molbev/msv281, lire en ligne, consulté le )
- Mads Albertsen, Philip Hugenholtz, Adam Skarshewski et Kåre L. Nielsen, « Genome sequences of rare, uncultured bacteria obtained by differential coverage binning of multiple metagenomes », Nature Biotechnology, vol. 31, no 6, , p. 533–538 (ISSN 1546-1696, PMID 23707974, DOI 10.1038/nbt.2579, lire en ligne, consulté le )
- Olga A. Podosokorskaya, Vitaly V. Kadnikov, Sergey N. Gavrilov et Andrey V. Mardanov, « Characterization of Melioribacter roseus gen. nov., sp. nov., a novel facultatively anaerobic thermophilic cellulolytic bacterium from the class Ignavibacteria, and a proposal of a novel bacterial phylum Ignavibacteriae », Environmental Microbiology, vol. 15, no 6, , p. 1759–1771 (ISSN 1462-2920, PMID 23297868, DOI 10.1111/1462-2920.12067, lire en ligne, consulté le )
- Rasmus Hansen Kirkegaard, Morten Simonsen Dueholm, Simon Jon McIlroy et Marta Nierychlo, « Genomic insights into members of the candidate phylum Hyd24-12 common in mesophilic anaerobic digesters », The ISME journal, vol. 10, no 10, , p. 2352–2364 (ISSN 1751-7370, PMID 27058503, PMCID 5030696, DOI 10.1038/ismej.2016.43, lire en ligne, consulté le )
- (en) The ARB development Team, « Arb-Silva Browser », sur Arb-Silva
- (en) « Hierarchy Browser », sur Ribosomal database project
- (en) « Taxonomy Browser », sur NCBI
- Fabia U. Battistuzzi, Andreia Feijao et S. Blair Hedges, « A genomic timescale of prokaryote evolution: insights into the origin of methanogenesis, phototrophy, and the colonization of land », BMC evolutionary biology, vol. 4, , p. 44 (ISSN 1471-2148, PMID 15535883, DOI 10.1186/1471-2148-4-44, lire en ligne, consulté le )
- Fabia U. Battistuzzi et S. Blair Hedges, « A major clade of prokaryotes with ancient adaptations to life on land », Molecular Biology and Evolution, vol. 26, no 2, , p. 335–343 (ISSN 1537-1719, PMID 18988685, DOI 10.1093/molbev/msn247, lire en ligne, consulté le )
- David W. Waite, Inka Vanwonterghem, Christian Rinke et Donovan H. Parks, « Comparative Genomic Analysis of the Class Epsilonproteobacteria and Proposed Reclassification to Epsilonbacteraeota (phyl. nov.) », Frontiers in Microbiology, vol. 8, , p. 682 (ISSN 1664-302X, PMID 28484436, PMCID 5401914, DOI 10.3389/fmicb.2017.00682, lire en ligne, consulté le )
- (en) R. G. E. Murray, « The higher taxa, or, a place for everything...? », Bergey's manual of systematic bacteriology, 1re série, vol. 1, , p. 31-34
- Anna Kielak, Agata S. Pijl, Johannes A. van Veen et George A. Kowalchuk, « Phylogenetic diversity of Acidobacteria in a former agricultural soil », The ISME journal, vol. 3, no 3, , p. 378–382 (ISSN 1751-7370, PMID 19020558, DOI 10.1038/ismej.2008.113, lire en ligne, consulté le )
- Jacqueline A. Servin, Craig W. Herbold, Ryan G. Skophammer et James A. Lake, « Evidence excluding the root of the tree of life from the actinobacteria », Molecular Biology and Evolution, vol. 25, no 1, , p. 1–4 (ISSN 1537-1719, PMID 18003601, DOI 10.1093/molbev/msm249, lire en ligne, consulté le )
- Emma Griffiths et Radhey S. Gupta, « Molecular signatures in protein sequences that are characteristics of the phylum Aquificae », International Journal of Systematic and Evolutionary Microbiology, vol. 56, no Pt 1, , p. 99–107 (ISSN 1466-5026, PMID 16403873, DOI 10.1099/ijs.0.63927-0, lire en ligne, consulté le )
- (en) Bergey's Manual of Systematic Bacteriology : Volume 4 : The Bacteroidetes, Spirochaetes, Tenericutes (Mollicutes), Acidobacteria, Fibrobacteres, Fusobacteria, Dictyoglomi, Gemmatimonadetes, Lentisphaerae, Verrucomicrobia, Chlamydiae, and Planctomycetes, Springer-Verlag, coll. « Bergey's Manual of Systematic Bacteriology », , 949 p. (ISBN 978-0-387-95042-6, lire en ligne)
- S. H. Duncan, G. E. Lobley, G. Holtrop et J. Ince, « Human colonic microbiota associated with diet, obesity and weight loss », International Journal of Obesity (2005), vol. 32, no 11, , p. 1720–1724 (ISSN 1476-5497, PMID 18779823, DOI 10.1038/ijo.2008.155, lire en ligne, consulté le )
- Barbara S. Sixt, Alexander Siegl, Constanze Müller et Margarete Watzka, « Metabolic features of Protochlamydia amoebophila elementary bodies--a link between activity and infectivity in Chlamydiae », PLoS pathogens, vol. 9, no 8, , e1003553 (ISSN 1553-7374, PMID 23950718, PMCID 3738481, DOI 10.1371/journal.ppat.1003553, lire en ligne, consulté le )
- Cherilyn Elwell, Kathleen Mirrashidi et Joanne Engel, « Chlamydia cell biology and pathogenesis », Nature Reviews. Microbiology, vol. 14, no 6, , p. 385–400 (ISSN 1740-1534, PMID 27108705, PMCID 4886739, DOI 10.1038/nrmicro.2016.30, lire en ligne, consulté le )
- Donald A. Bryant et Niels-Ulrik Frigaard, « Prokaryotic photosynthesis and phototrophy illuminated », Trends in Microbiology, vol. 14, no 11, , p. 488–496 (ISSN 0966-842X, PMID 16997562, DOI 10.1016/j.tim.2006.09.001, lire en ligne, consulté le )
- Gert T. Oostergetel, Herbert van Amerongen et Egbert J. Boekema, « The chlorosome: a prototype for efficient light harvesting in photosynthesis », Photosynthesis Research, vol. 104, nos 2-3, , p. 245–255 (ISSN 1573-5079, PMID 20130996, PMCID 2882566, DOI 10.1007/s11120-010-9533-0, lire en ligne, consulté le )
- Philip Hugenholtz et Erko Stackebrandt, « Reclassification of Sphaerobacter thermophilus from the subclass Sphaerobacteridae in the phylum Actinobacteria to the class Thermomicrobia (emended description) in the phylum Chloroflexi (emended description) », International Journal of Systematic and Evolutionary Microbiology,, vol. 54, no 6, , p. 2049–2051 (ISSN 1466-5026, DOI 10.1099/ijs.0.03028-0, lire en ligne, consulté le )
- (en) « 'The All-Species Living Tree' Project: 16S rRNA-based LTP release 123 (full tree) » [PDF], sur Silva Comprehensive Ribosomal RNA Database
- J. M. Macy, K. Nunan, K. D. Hagen et D. R. Dixon, « Chrysiogenes arsenatis gen. nov., sp. nov., a new arsenate-respiring bacterium isolated from gold mine wastewater », International Journal of Systematic Bacteriology, vol. 46, no 4, , p. 1153–1157 (ISSN 0020-7713, PMID 8863450, DOI 10.1099/00207713-46-4-1153, lire en ligne, consulté le )
- (en) D. Y. Sorokin et G. Muyzer, « Desulfurispira natronophila gen. nov. sp. nov.: an obligately anaerobic dissimilatory sulfur-reducing bacterium from soda lakes », Extremophiles, vol. 14, no 4, , p. 349–355 (ISSN 1433-4909, PMID 20407798, PMCID PMC2898105, DOI 10.1007/s00792-010-0314-7, lire en ligne, consulté le )
- (en) D. Yu. Sorokin, M. Foti, B. J. Tindall et G. Muyzer, « Desulfurispirillum alkaliphilum gen. nov. sp. nov., a novel obligately anaerobic sulfur- and dissimilatory nitrate-reducing bacterium from a full-scale sulfide-removing bioreactor », Extremophiles, vol. 11, no 2, , p. 363–370 (ISSN 1433-4909, DOI 10.1007/s00792-006-0048-8, lire en ligne, consulté le )
- Aharon Oren, « A proposal for further integration of the cyanobacteria under the Bacteriological Code », International Journal of Systematic and Evolutionary Microbiology,, vol. 54, no 5, , p. 1895–1902 (ISSN 1466-5026, DOI 10.1099/ijs.0.03008-0, lire en ligne, consulté le )
- (en) G. C. Dismukes, V. V. Klimov, S. V. Baranov et Yu N. Kozlov, « The origin of atmospheric oxygen on Earth: The innovation of oxygenic photosynthesis », Proceedings of the National Academy of Sciences, vol. 98, no 5, , p. 2170–2175 (ISSN 0027-8424 et 1091-6490, PMID 11226211, DOI 10.1073/pnas.061514798, lire en ligne, consulté le )
- (en) H. Huber et K.O. Stetter, Bergey's Manual of Systematic Bacteriology, vol. 1 : The Archaea and the deeply branching and phototrophic Bacteria, New York, Springer-Verlag, , 2e éd., « Family I. Deferribacteraceae fam. nov. », p. 465-466
- (en) E. Griffiths et R. S. Gupta, « Identification of signature proteins that are distinctive of the Deinococcus-Thermus phylum. », International Microbiology, 3e série, vol. 10, (lire en ligne [PDF])
- « The World’s Toughest Bacterium », sur www.genomenewsnetwork.org (consulté le )
- (en) Thomas D. Brock et Hudson Freeze, « Thermus aquaticus gen. n. and sp. n., a Nonsporulating Extreme Thermophile », Journal of Bacteriology, vol. 98, no 1, , p. 289–297 (ISSN 0021-9193 et 1098-5530, PMID 5781580, lire en ligne, consulté le )
- R. K. Saiki, D. H. Gelfand, S. Stoffel et S. J. Scharf, « Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase », Science (New York, N.Y.), vol. 239, no 4839, , p. 487–491 (ISSN 0036-8075, PMID 2448875, DOI 10.1126/science.2448875, lire en ligne, consulté le )
- TAKASHI SAIKI, YASUHIKO KOBAYASHI, KIYOTAKA KAWAGOE et TERUHIKO BEPPU, « Dictyoglomus thermophilum gen. nov., sp. nov., a Chemoorganotrophic, Anaerobic, Thermophilic Bacterium », International Journal of Systematic and Evolutionary Microbiology,, vol. 35, no 3, , p. 253–259 (ISSN 1466-5026, DOI 10.1099/00207713-35-3-253, lire en ligne, consulté le )
- (en) Marjaana Rättö, Indra M. Mathrani, Birgitte Ahring et Liisa Viikari, « Application of thermostable xylanase of Dictyoglomus sp. in enzymatic treatment of kraft pulps », Applied Microbiology and Biotechnology, vol. 41, no 1, , p. 130–133 (ISSN 1432-0614, DOI 10.1007/BF00166094, lire en ligne, consulté le )
- Radhey S. Gupta, « The phylogeny and signature sequences characteristics of Fibrobacteres, Chlorobi, and Bacteroidetes », Critical Reviews in Microbiology, vol. 30, no 2, , p. 123–143 (ISSN 1040-841X, PMID 15239383, DOI 10.1080/10408410490435133, lire en ligne, consulté le )
- (en) Emma Ransom-Jones, David L. Jones, Alan J. McCarthy et James E. McDonald, « The Fibrobacteres: an Important Phylum of Cellulose-Degrading Bacteria », Microbial Ecology, vol. 63, no 2, , p. 267–281 (ISSN 1432-184X, DOI 10.1007/s00248-011-9998-1, lire en ligne, consulté le )
- LARRY MONTGOMERY, BERDENA FLESHER et DAVID STAHL, « Transfer of Bacteroides succinogenes (Hungate) to Fibrobacter gen. nov. as Fibrobacter succinogenes comb. nov. and Description of Fibrobacter intestinalis sp. nov. », International Journal of Systematic and Evolutionary Microbiology,, vol. 38, no 4, , p. 430–435 (ISSN 1466-5026, DOI 10.1099/00207713-38-4-430, lire en ligne, consulté le )
- N. E. GIBBONS et R. G. E. MURRAY, « Proposals Concerning the Higher Taxa of Bacteria », International Journal of Systematic and Evolutionary Microbiology,, vol. 28, no 1, , p. 1–6 (ISSN 1466-5026, DOI 10.1099/00207713-28-1-1, lire en ligne, consulté le )
- (en) Wolfgang Ludwig, Karl-Heinz Schleifer et William B. Whitman, « Bacilli class. nov. », dans Bergey's Manual of Systematics of Archaea and Bacteria, American Cancer Society, (ISBN 9781118960608, DOI 10.1002/9781118960608.cbm00033, lire en ligne), p. 1–1
- (de) F. Cohn, « Untersuchungen über Bacterien. », Beiträge zur Biologie der Pflanzen, 2e série, vol. 1, , p. 127-224
- (en) Lactic acid bacteria, New York: Dekker, S. Salminen et A. Von Wright, , p. 199-225
- K. W. Bennett et A. Eley, « Fusobacteria: new taxonomy and related diseases », Journal of Medical Microbiology, vol. 39, no 4, , p. 246–254 (ISSN 0022-2615, PMID 8411084, DOI 10.1099/00222615-39-4-246, lire en ligne, consulté le )
- N. M. Ovchinnikov, « [Centenary of the discovery of the causative agent of gonorrhea] », Vestnik Dermatologii I Venerologii, no 1, , p. 3–7 (ISSN 0042-4609, PMID 105505, lire en ligne, consulté le )
- Mariam Fawaz, « Revealing the Ecological Role of Gemmatimonadetes Through Cultivation and Molecular Analysis of Agricultural Soils », Masters Theses, (lire en ligne, consulté le )
- Jang-Cheon Cho, Kevin L. Vergin, Robert M. Morris et Stephen J. Giovannoni, « Lentisphaera araneosa gen. nov., sp. nov, a transparent exopolymer producing marine bacterium, and the description of a novel bacterial phylum, Lentisphaerae », Environmental Microbiology, vol. 6, no 6, , p. 611–621 (ISSN 1462-2912, PMID 15142250, DOI 10.1111/j.1462-2920.2004.00614.x, lire en ligne, consulté le )
- (en) George M. Garrity, John G. Holt, Eva Spieck et Eberhard Bock, « Phylum BVIII. Nitrospirae phy. nov. », dans Bergey’s Manual® of Systematic Bacteriology: Volume One : The Archaea and the Deeply Branching and Phototrophic Bacteria, Springer New York, (ISBN 9780387216096, DOI 10.1007/978-0-387-21609-6_25, lire en ligne), p. 451–464
- Cheryl Jenkins, Vishram Kedar et John A. Fuerst, « Gene discovery within the planctomycete division of the domain Bacteria using sequence tags from genomic DNA libraries », Genome Biology, vol. 3, no 6, , research0031.1 (ISSN 1474-760X, DOI 10.1186/gb-2002-3-6-research0031, lire en ligne, consulté le )
- Mike S. M. Jetten, Laura van Niftrik, Marc Strous et Boran Kartal, « Biochemistry and molecular biology of anammox bacteria », Critical Reviews in Biochemistry and Molecular Biology, vol. 44, nos 2-3, , p. 65–84 (ISSN 1040-9238, PMID 19247843, DOI 10.1080/10409230902722783, lire en ligne, consulté le )
- (en) Sherris Medical Microbiology : An Introduction to Infectious Diseases, McGraw Hill, K. J. Ryan et C. G. Ray, , 4e éd., 979 p. (ISBN 978-0-8385-8529-0)
- Kit Tilly, Patricia A. Rosa et Philip E. Stewart, « Biology of infection with Borrelia burgdorferi », Infectious Disease Clinics of North America, vol. 22, no 2, , p. 217–234, v (ISSN 0891-5520, PMID 18452798, PMCID 2440571, DOI 10.1016/j.idc.2007.12.013, lire en ligne, consulté le )
- Hélène Marchandin, Audrey Damay, Laurent Roudière et Corinne Teyssier, « Phylogeny, diversity and host specialization in the phylum Synergistetes with emphasis on strains and clones of human origin », Research in Microbiology, vol. 161, no 2, , p. 91–100 (ISSN 1769-7123, PMID 20079831, DOI 10.1016/j.resmic.2009.12.008, lire en ligne, consulté le )
- (en) Wolfgang Ludwig, Karl-Heinz Schleifer et William B. Whitman, « Revised road map to the phylum Firmicutes », dans Bergey’s Manual® of Systematic Bacteriology: Volume Three The Firmicutes, Springer New York, (ISBN 9780387684895, DOI 10.1007/978-0-387-68489-5_1, lire en ligne), p. 1–13
- (en) T. J. Vick, J. A. Dodsworth, K. C. Costa et E. L. Shock, « Microbiology and geochemistry of Little Hot Creek, a hot spring environment in the Long Valley Caldera », Geobiology, vol. 8, no 2, , p. 140–154 (ISSN 1472-4669, DOI 10.1111/j.1472-4669.2009.00228.x, lire en ligne, consulté le )
- Christian Jeanthon, Stéphane L'Haridon, Valérie Cueff et Amy Banta, « Thermodesulfobacterium hydrogeniphilum sp. nov., a thermophilic, chemolithoautotrophic, sulfate-reducing bacterium isolated from a deep-sea hydrothermal vent at Guaymas Basin, and emendation of the genus Thermodesulfobacterium. », International Journal of Systematic and Evolutionary Microbiology,, vol. 52, no 3, , p. 765–772 (ISSN 1466-5026, DOI 10.1099/00207713-52-3-765, lire en ligne, consulté le )
- (en) George M. Garrity, John G. Holt et Jerome J. Perry, « Phylum BVII. Thermomicrobia phy. nov. », dans Bergey’s Manual® of Systematic Bacteriology: Volume One : The Archaea and the Deeply Branching and Phototrophic Bacteria, Springer New York, (ISBN 9780387216096, DOI 10.1007/978-0-387-21609-6_24, lire en ligne), p. 447–450
- Karen M. Houghton, Xochitl C. Morgan, Kirill Lagutin et Andrew D. MacKenzie, « Thermorudis pharmacophila sp. nov., a novel member of the class Thermomicrobia isolated from geothermal soil, and emended descriptions of Thermomicrobium roseum, Thermomicrobium carboxidum, Thermorudis peleae and Sphaerobacter thermophilus », International Journal of Systematic and Evolutionary Microbiology, vol. 65, no 12, , p. 4479–4487 (ISSN 1466-5034, PMID 26374291, DOI 10.1099/ijsem.0.000598, lire en ligne, consulté le )
- C. E. King et G. M. King, « Thermomicrobium carboxidum sp. nov., and Thermorudis peleae gen. nov., sp. nov., carbon monoxide-oxidizing bacteria isolated from geothermally heated biofilms », International Journal of Systematic and Evolutionary Microbiology,, vol. 64, no 8, , p. 2586–2592 (ISSN 1466-5026, DOI 10.1099/ijs.0.060327-0, lire en ligne, consulté le )
- (en) Robert Huber et Michael Hannig, « Thermotogales », dans The Prokaryotes: Volume 7: Proteobacteria: Delta, Epsilon Subclass, Springer New York, (ISBN 9780387307473, DOI 10.1007/0-387-30747-8_38, lire en ligne), p. 899–922
- Kuo-Chang Lee, Richard I Webb, Peter H Janssen et Parveen Sangwan, « Phylum Verrucomicrobia representatives share a compartmentalized cell plan with members of bacterial phylum Planctomycetes », BMC Microbiology, vol. 9, , p. 5 (ISSN 1471-2180, PMID 19133117, PMCID 2647929, DOI 10.1186/1471-2180-9-5, lire en ligne, consulté le )
- (en) Nicholas C. Butzin, Pascal Lapierre, Anna G. Green et Kristen S. Swithers, « Reconstructed Ancestral Myo-Inositol-3-Phosphate Synthases Indicate That Ancestors of the Thermococcales and Thermotoga Species Were More Thermophilic than Their Descendants », PLOS ONE, vol. 8, no 12, , e84300 (ISSN 1932-6203, PMID 24391933, PMCID PMC3877268, DOI 10.1371/journal.pone.0084300, lire en ligne, consulté le )
- Alejandra Moreno-Letelier, Gabriela Olmedo-Alvarez, Luis E. Eguiarte et Valeria Souza, « Divergence and Phylogeny of Firmicutes from the Cuatro Ciénegas Basin, Mexico: A Window to an Ancient Ocean », Astrobiology, vol. 12, no 7, , p. 674–684 (ISSN 1531-1074, PMID 22920517, PMCID 3426897, DOI 10.1089/ast.2011.0685, lire en ligne, consulté le )
Articles connexes
[modifier | modifier le code]- Taxonomie bactérienne
- Nomenclature bactérienne
- Liste d'espèces eubactériennes dont le génome est séquencé
Liens externes
[modifier | modifier le code]- Site du LPSN (Liste des noms de procaryotes ayant le statut de nomenclature en règle, List of Prokaryotic names with Standing in Nomenclature en anglais)
- Base de données Arb-Silva
- Site du Ribosomal Database Project 10
