Filoviridae
| Rang ' royaume ' non reconnu | Riboviria |
|---|---|
| Règne | Orthornavirae |
| Embranchement | Negarnaviricota |
| Sous-embr. | Haploviricotina |
| Classe | Monjiviricetes |
| Ordre | Mononegavirales |
La famille des Filoviridae, ou filovirus, appartient à l'ordre des Mononegavirales (mononégavirus) regroupant les virus à ARN monocaténaire non segmenté à polarité négative. Les filovirus sont ainsi dénommés car ils présentent une apparence filamenteuse.
La plupart d'entre eux (mais il existe peu d'exceptions[4]) sont des agents infectieux responsables de fièvres hémorragiques[5] aiguës particulièrement létales, notamment :
- le virus Ebola, responsable de la maladie à virus Ebola ;
- le virus Marburg, responsable de la fièvre hémorragique de Marburg.
Taxinomie
Ils appartiennent à l'ordre des Mononegavirales, comprenant les virus à ARN monocaténaire non segmenté à polarité négative. Initialement classés parmi les rhabdovirus, les filovirus forment aujourd'hui une famille distincte et seraient en réalité plus proches des paramyxovirus[6], parmi lesquels on trouve notamment les virus des oreillons et de la rougeole.
On connaît cinq virus distincts[7],[8], que l'ICTV rattache chacun à l'une des cinq espèces du genre Ebolavirus. Cependant, la taxinomie des filovirus est récente et continue d'évoluer au gré des avancées phylogénétiques, d'où une relative confusion entre les différentes dénominations retenues selon les auteurs[9]. Un usage bien ancré dans les laboratoires fait du virus Ebola une désignation synonyme du genre Ebolavirus décliné en cinq sous-types de virus[10],[11], tandis que la nomenclature adoptée par l'ICTV, faisant du virus Ebola le virus de l'espèce type du genre Ebolavirus, n'a pas encore été ratifiée.

On distingue :
- le virus Ebola proprement dit (EBOV), de l'espèce ebolavirus Zaïre (autrefois ZEBOV), ou sous-type Ebola Zaïre, identifié pour la première fois en 1976 au Zaïre[12] (aujourd'hui République démocratique du Congo) — c'est le plus virulent des cinq virus, à l'origine de l'épidémie de 2014 en Afrique de l'Ouest[13] ;
- le virus Soudan (SUDV), de l'espèce ebolavirus Soudan (autrefois SEBOV), ou sous-type Ebola Soudan, endémique au Soudan du Sud et en Ouganda[14] ;
- le virus Reston (RESTV), de l'espèce ebolavirus Reston (autrefois REBOV), ou sous-type Ebola Reston, identifié en 1983 dans la région de Reston, aux États-Unis[15] ;
- le virus Forêt de Taï (TAFV), de l'espèce ebolavirus Forêt de Taï, autrefois ebolavirus Côte d'Ivoire (CIEBOV), ou sous-type Ebola Forêt de Taï (ou encore Ebola Côte d'Ivoire), identifié en 1994 dans le parc national de Taï, en Côte d'Ivoire, aux confins de la Guinée et du Liberia[16] ;
- le virus Bundibugyo (BDBV), de l'espèce ebolavirus Bundibugyo (autrefois BEBOV), ou sous-type Ebola Bundibugyo, identifié en 2008 dans la région de Bundibugyo, en Ouganda[17].
Virions
Les filovirus sont, comme leur nom l'indique, des particules virales d'apparence filamenteuse. La capside en elle-même présente une apparence filamenteuse en U, en 6, en crochet ou en bâtonnet, et peut être ramifiée[9].
Par ailleurs ce sont des virus enveloppés -- la capside n'est pas nue mais recouverte d'une membrane lipidique. Les virions sont également caractérisés par la présence d'une glycoprotéine fortement glycosylée constituée de deux sous-unités différentes et formant des trimères insérés dans l'enveloppe virale en formant des projections de 7 nm espacées d'environ 10 nm[9].
(tomographie électronique)
Génomique
Le nom des espèces virales validé par l'ICTV a sensiblement évolué depuis l'identification de ces virus[19],[20],[21],[22],[23],[24]. La taxonomie de la famille peut encore évoluer au fur et à mesure des études génétiques et de l'éventuelle découverte de nouveaux membres de la famille [25], en zone tropicale, mais aussi en zone tempérée (un virus proche d'Ebola a par exemple été trouvé en Europe[26]).
Ces virus possèdent un génome de 19 kilobases, plus long que la plupart des mononégavirus (dont le génome mesure entre 8 et 16 kb). Leur ARN génomique contient un ou plusieurs chevauchements de gènes. Les gènes de ce génome sont organisés selon le schéma 3'-UTR–NP–VP35–VP40–GP–VP30–VP24–L–5'-UTR ; la protéine VP24 est spécifique aux filovirus tandis que la protéine VP30 est partiellement analogue à une protéine uniquement exprimée chez les pneumovirus[9].
| Virus | Taille de l'ARN génomique |
Taux de GC | Réf. |
|---|---|---|---|
| Ebola | 18,96 kb | 41,1 % | [1] |
| Soudan | 18,88 kb | 41,3 % | [2] |
| Reston | 18,89 kb | 40,6 % | [3] |
| Forêt de Taï | 18,93 kb | 42,3 % | [4] |
| Bundibugyo | 18,94 kb | 42,0 % | [5] |
Les variations génétiques entre les différents virus du genre Ebolavirus peuvent être illustrées par la comparaison de la taille des différents génomes et leurs taux de GC respectifs (voir tableau ci-contre à droite).[réf. nécessaire]
Par comparaison, parmi les autres filovirus, le virus Marburg, du genre Marburgvirus, a un génome long de 19,11 kb avec un taux de GC de 38,8 %[27] tandis que le virus Lloviu, du genre Cuevavirus, a un génome long de 18,93 kb avec un taux de GC de 46,0 %[28].
Pouvoir pathogène
Ces virus se développent dans l’organisme de certains animaux (en particulier chiroptères et primates -- non-humain comme humains [29]).
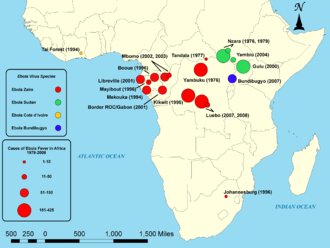
- virus Ebola en rouge ;
- virus Soudan en vert ;
- virus Forêt de Taï en orange ;
- virus Bundibugyo en bleu.
La nature pathogène des différents filovirus, qu'il s'agisse du genre Ebolavirus ou du genre Marburgvirus, est très semblable dans la mesure où ces virus ont tous été associés à des flambées de fièvres hémorragiques chez l'homme et les autres primates avec des symptômes identiques. Ils diffèrent en revanche du point de vue génétique, avec une séquence nucléotidique pouvant varier de 30 à 40 % d'une souche à l'autre, ce qui se traduit par une sévérité très différente entre les pathologies induites chez l'homme par ces différents virus — la létalité peut ainsi être nulle chez les humains pour le virus Reston, mais approcher 90 % pour le virus Ebola — bien que des facteurs environnementaux puissent également expliquer ces différences.[réf. nécessaire]
Le virus Reston a été isolé en 1989 chez des macaques crabiers aux Philippines. Présent également en Chine, il est moins pathogène chez les primates non humains et l'on pensait qu'il n'affectait pas les humains jusqu'à ce qu'on identifie une transmission du porc à l'homme en 2009.[réf. nécessaire]Le virus Bundibugyo, découvert en 2008, s’apparente davantage au virus Forêt de Taï[7], mais est plus virulent que ce dernier.
Évolution
Pour savoir si les filovirus sont réellement source d'un risque pandémique [31], il est utile de connaitre l'histoire de ces virus et de déterminer et comprendre leurs capacités évolutives et adapatatives (par le jeu des mutations) sur le long et moyen terme, et sur le court terme lors d'une épidémie, ou d'évaluer leur capacité d'adaptation à d'autres environnement et à d'autres hôtes[32].
Selon les données génétiques disponibles, les virus des groupes Marburg (marburgvirus) et Ebola (ebolavirus) du Soudan partageraient un ancêtre commun récent (aux échelles de l'évolution); ils seraient nés de cet ancêtre approximativement 700 ans avant nos jours pour le premier groupe et il y a 850 ans environ pour le second. Et les membres connus de la famille des Filoviridae (dont le virus Lloviu récemment décrit) partageraient un ancêtre commun vieux d'environ 10 000 ans (fin de la dernière glaciation).[réf. nécessaire]
Selon Serena A. Carroll & al., comprendre le passé et le paléoenvironnement de ces virus pourraient aider à mieux comprendre le rôle des espèces réservoir et à mieux combattre ou prévenir les épidémies humaines en comprenant mieux les mécanismes d'émergence, l'évolution de la pathogénicité des virus et des risques pour la santé publique et la santé environnementale (écoépidémiologie).[réf. nécessaire]
En particulier, il est important de comprendre si et comment des animaux sauvages (viande de brousse, singes destinés à l'expérimentation animale...) ou certains animaux d'élevage ou domestiqués susceptibles de faire l'objet d'échanges internationaux peuvent être porteurs, voire porteurs sains de virus de cette famille, et comment ils lui résistent si c'est le cas, y compris par exemple pour le porc par exemple[33] qui est de plus en plus génétiquement homogénéisé dans le monde, un vecteur potentiel de nombreux virus.
Liste des genres et des espèces
Selon l’ICTV (version de juillet 2019[3]) :
- genre Cuevavirus
- espèce Lloviu cuevavirus
- genre Dianlovirus
- espèce Mengla dianlovirus
- genre Ebolavirus
- espèce Bombali ebolavirus
- espèce Bundibugyo ebolavirus
- espèce Reston ebolavirus
- espèce Sudan ebolavirus
- espèce Tai Forest ebolavirus
- espèce Zaire ebolavirus
- genre Marburgvirus
- espèce Marburg marburgvirus
- genre Striavirus
- espèce Xilang striavirus
- genre Thamnovirus
- espèce Huangjiao thamnovirus
Notes et références
- (en) « Microscopie électronique en transmission d'un virus Ebola », CDC, États-Unis
- ICTV. International Committee on Taxonomy of Viruses. Taxonomy history. Published on the Internet https://talk.ictvonline.org/., consulté le 5 février 2021
- (en) « Virus Taxonomy: 2019 Release », ICTV, (consulté le ).
- Sanchez A, Geisbert TW, Feldmann H. 2007. Filoviridae: Marburg and Ebola Viruses, p. 1409–1448. In Knipe DM, Howley PM (ed), Fields virology. Lippincott Williams and Wilkins, Philadelphia, PA.
- McKee, K., & Khan, A. S. (1998) Hemorrhagic fever viruses belonging to the families Arenaviridae, Filoviridae, and Bunyaviridae. Infectious Diseases, 2249-65.
- (en) Anthony Sanchez, Michael P. Kiley, Hans-Dieter Klenk et Heinz Feldmann, « Sequence analysis of the Marburg virus nucleoprotein gene: comparison to Ebola virus and other non-segmented negative-strand RNA viruses », Journal of General Virology, vol. 73, no 2, , p. 347-357 (PMID 1538192, DOI 10.1099/0022-1317-73-2-347, lire en ligne)
- (en) Jonathan S. Towner, Tara K. Sealy, Marina L. Khristova, César G. Albariño, Sean Conlan, Serena A. Reeder, Phenix-Lan Quan, W. Ian Lipkin, Robert Downing, Jordan W. Tappero, Samuel Okware, Julius Lutwama, Barnabas Bakamutumaho, John Kayiwa, James A. Comer, Pierre E. Rollin, Thomas G. Ksiazek et Stuart T. Nichol, « Newly Discovered Ebola Virus Associated with Hemorrhagic Fever Outbreak in Uganda », PLoS Pathogens, vol. 4, no 11, , e1000212 (PMID 19023410, PMCID 2581435, DOI 10.1371/journal.ppat.1000212, lire en ligne)
- (en) Ayato Takada et Yoshihiro Kawaoka, « The pathogenesis of Ebola hemorrhagic fever », Trends in Microbiology, vol. 9, no 10, , p. 506-511 (PMID 11597453, DOI 10.1016/S0966-842X(01)02201-6, lire en ligne)
- (en) Jens H. Kuhn, Stephan Becker, Hideki Ebihara, Thomas W. Geisbert, Karl M. Johnson, Yoshihiro Kawaoka, W. Ian Lipkin, Ana I. Negredo, Sergey V. Netesov, Stuart T. Nichol, Gustavo Palacios, Clarence J. Peters, Antonio Tenorio, Viktor E. Volchkov et Peter B. Jahrling, « Proposal for a revised taxonomy of the family Filoviridae: classification, names of taxa and viruses, and virus abbreviations », Archives of Virology, vol. 155, no 12, , p. 2083-2103 (PMID 21046175, PMCID 3074192, DOI 10.1007/s00705-010-0814-x, lire en ligne)
- « Avis du Haut Conseil de la santé publique relatif à la conduite à tenir autour des cas suspects de maladie Ebola » [PDF], hat Conseil de la santé publique (France),
- « Infections par les Filovirus, les Arenavirus et les Hantavirus » [PDF] — Pages 723 et suivantes de l'ouvrage du Collège des Universitaires de Maladies Infectieuses et Tropicales (CMIT).
- (en) S. Pattyn, G.vander Groen, W. Jacob, P. Piot et G. Courteille, « Isolation of Marburg-like virus from a case of hæmorrhagic fever in Zaire », The Lancet, vol. 309, no 8011, , p. 573-574 (PMID 65663, DOI 10.1016/S0140-6736(77)92002-5, lire en ligne)
- (en) Sylvain Baize, Delphine Pannetier, Lisa Oestereich, Toni Rieger, Lamine Koivogui, N'Faly Magassouba, Barrè Soropogui, Mamadou Saliou Sow, Sakoba Keïta, Hilde De Clerck, Amanda Tiffany, Gemma Dominguez, Mathieu Loua, Alexis Traoré, Moussa Kolié, Emmanuel Roland Malano, Emmanuel Heleze, Anne Bocquin, Stephane Mély, Hervé Raoul, Valérie Caro, Dániel Cadar, Martin Gabriel, Meike Pahlmann, Dennis Tappe, Jonas Schmidt-Chanasit, Benido Impouma, Abdoul Karim Diallo, Pierre Formenty, Michel Van Herp et Stephan Günther, « Emergence of Zaire Ebola Virus Disease in Guinea — Preliminary Report », The New England Journal of Medicine, (PMID 24738640, DOI 10.1056/NEJMoa1404505, lire en ligne)
- « Ebola haemorrhagic fever in Sudan, 1976. Report of a WHO/International Study Team », Bulletin of the World Health Organization, vol. 56, no 2, , p. 247–270 (ISSN 0042-9686, PMID 307455, PMCID 2395561, lire en ligne, consulté le )
- P. B. Jahrling, T. W. Geisbert, D. W. Dalgard et E. D. Johnson, « Preliminary report: isolation of Ebola virus from monkeys imported to USA », Lancet (London, England), vol. 335, no 8688, , p. 502–505 (ISSN 0140-6736, PMID 1968529, DOI 10.1016/0140-6736(90)90737-p, lire en ligne, consulté le )
- B. Le Guenno, P. Formenty, P. Formentry et M. Wyers, « Isolation and partial characterisation of a new strain of Ebola virus », Lancet (London, England), vol. 345, no 8960, , p. 1271–1274 (ISSN 0140-6736, PMID 7746057, DOI 10.1016/s0140-6736(95)90925-7, lire en ligne, consulté le )
- Jonathan S. Towner, Tara K. Sealy, Marina L. Khristova et César G. Albariño, « Newly discovered ebola virus associated with hemorrhagic fever outbreak in Uganda », PLoS pathogens, vol. 4, no 11, , e1000212 (ISSN 1553-7374, PMID 19023410, PMCID 2581435, DOI 10.1371/journal.ppat.1000212, lire en ligne, consulté le )
- (en) David Goodsell, « Ebola Virus Proteins » [PDF], sur rcsb.org/pdb, (DOI 10.2210/rcsb_pdb/mom_2014_10)
- (en) « ICTV Taxonomy History for Zaire ebolavirus », ICTV
- (en) « ICTV Taxonomy History for Sudan ebolavirus », ICTV
- (en) « ICTV Taxonomy History for Reston ebolavirus », ICTV
- (en) « ICTV Taxonomy History for Tai Forest ebolavirus », ICTV
- (en) « ICTV Taxonomy History for Bundibugyo ebolavirus », ICTV
- « Maladie à virus Ebola », OMS,
- Barrette RW, Xu L, Rowland JM, McIntosh MT. 2011. Current perspectives on the phylogeny of Filoviridae. Infect. Genet. Evol. 11(7):1514–1519.CrossRef
- Negredo A, Palacios G, Vazquez-Moron S, Gonzalez F, Dopazo H, Molero F, Juste J, Quetglas J, Savji N, de la Cruz Martinez M, Herrera JE, Pizarro M, Hutchison SK, Echevarria JE, Lipkin WI, Tenorio A. (2011) Discovery of an ebolavirus-like filovirus in Europe. PLoS Pathog. 7:e1002304. doi:10.1371/journal.ppat.1002304.
- (en) Marburg marburgvirus sur le site Genome du NCBI.
- (en) Lloviu cuevavirus sur le site Genome du NCBI.
- Ryabchikova, E. I., Kolesnikova, L. V., & Netesov, S. V. (1999) Animal pathology of filoviral infections. Current topics in microbiology and immunology, 235, 145.
- (en) « Ebola Hemorrhagic Fever Distribution Map », Centers for Disease Control and Prevention
- Martina B et Osterhaus A (2009) Filoviruses" : a real pandemic threat ? ; EMBO Mol Med. (2009) 1 (1): 10-18
- Rodriguez LL, De Roo A, Guimard Y, Trappier SG, Sanchez A, Bressler D, Williams AJ, Rowe AK, Bertolli J, Khan AS, Ksiazek TG, Peters CJ, Nichol ST (1999) Persistence and genetic stability of Ebola virus during the outbreak in Kikwit, Democratic Republic of the Congo, 1995. J. Infect. Dis. 179(Suppl 1):S170–S176.
- Barrette RW, Metwally SA, Rowland JM, Xu L, Zaki SR, Nichol ST, Rollin PE, Towner JS, Shieh W-J, Batten B, Sealy TK, Carrillo C, Moran KE, Bracht AJ, Mayr GA, Sirios-Cruz M, Catbagan DP, Lautner EA, Ksiazek TG, White WR, McIntosh M (2009) Discovery of swine as a host for the Reston ebolavirus. Science 325:204–206.Abstract/FREE Full Text
Références biologiques
- (en) Référence ICTV : Filoviridae (consulté le )
- (en) Référence NCBI : Filoviridae (taxons inclus) (consulté le )
Voir aussi
Articles connexes
Liens externes
(en) « Ressources scientifiques pour les recherches sur les Filovirus », sur FILOVIR (consulté le )
Bibliographie
- (en) Hans-Dieter Klenk, Marburg and Ebola Viruses. Current Topics in Microbiology and Immunology, vol. 235, Berlin, Allemagne, Springer-Verlag, , 225 p. (ISBN 978-3-540-64729-4)
- (en) Hans-Dieter Klenk et Heinz Feldmann, Ebola and Marburg Viruses — Molecular and Cellular Biology, Wymondham, Norfolk, Royaume-Uni, Horizon Bioscience, , 369 p. (ISBN 978-0-9545232-3-7, lire en ligne)
- (en) Jens H. Kuhn, Filoviruses — A Compendium of 40 Years of Epidemiological, Clinical, and Laboratory Studies. Archives of Virology Supplement, vol. 20, Vienna, Autriche, Springer, , 413 p. (ISBN 978-3-211-20670-6)
- (en) Elena I. Ryabchikova et Barbara B. Price, Ebola and Marburg Viruses — A View of Infection Using Electron Microscopy, Columbus, Ohio, États-Unis, Battelle Press, , 211 p. (ISBN 978-1-57477-131-2)
- Beer, B., Kurth, R., & Bukreyev, A. (1999). Characteristics of filoviridae: Marburg and Ebola viruses. Naturwissenschaften, 86(1), 8-17 (résumé [6])
- Carroll, S. A., Towner, J. S., Sealy, T. K., McMullan, L. K., Khristova, M. L., Burt, F. J.... & Nichol, S. T. (2013). Molecular evolution of viruses of the family Filoviridae based on 97 whole-genome sequences. Journal of virology, 87(5), 2608-2616.
- Ignatyev, G. M. (1998). Immune response to filovirus infections. Current topics in microbiology and immunology, 235, 205-217.
- Kuhn, J. H., Bao, Y., Bavari, S., Becker, S., Bradfute, S., Brister, J. R.... & Nichol, S. T. (2013). Virus nomenclature below the species level: a standardized nomenclature for natural variants of viruses assigned to the family Filoviridae. Archives of virology, 158(1), 301-311 (résumé [7])
- Kiley, M. P., Bowen, E. T. W., Eddy, G. A., Isaäcson, M., Johnson, K. M., McCormick, J. B.... & Wulff, H. (1982). Filoviridae: a taxonomic home for Marburg and Ebola viruses ?. Intervirology, 18(1-2), 24-32 (résumé : [8].
- Zeller, H., & Bouloy, M. (2000). Infections by viruses of the families Bunyaviridae and Filoviridae. Revue scientifique et technique (International Office of Epizootics), 19(1), 79-91 (résumé : [9]




