Succinate déshydrogénase
| Succinate déshydrogénase | ||
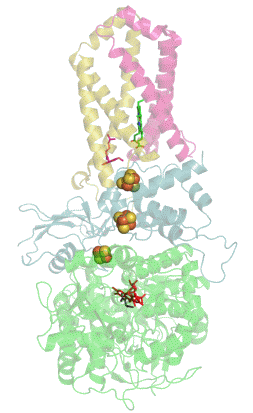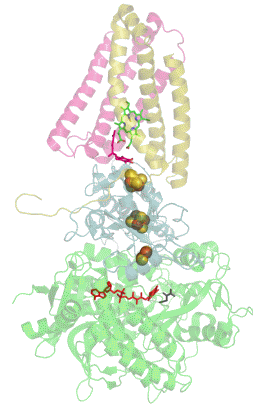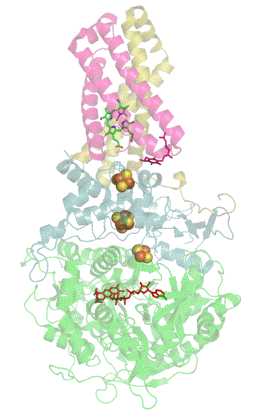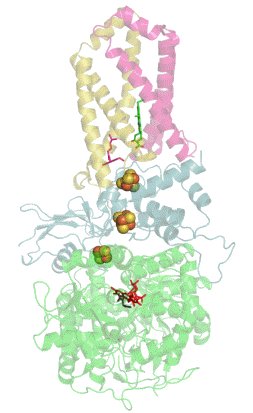 Structure tridimensionnelle d'une succinate déshydrogénase d'E. coli montrant l'emplacement des cofacteurs : on reconnaît de bas en haut le FAD, trois centres fer-soufre [2Fe-2S], [4Fe-4S] et [3Fe-4S], l'ubiquinone, et le groupe héminique (PDB 1NEK[1]). Les sous-unités sont identifiées par une couleur : SdhA en vert, SdhB en bleu, SdhC en rose et SdhD en jaune. | ||
| Caractéristiques générales | ||
|---|---|---|
| Nom approuvé | Complexe succinate déshydrogénase | |
| Symbole | SDH | |
| N° EC | 1.3.5.1 | |
| Gène SDHA Sous-unité A, flavoprotéine | ||
| Homo sapiens | ||
| Locus | 5p15.33 | |
| Masse moléculaire | 72 692 Da[2] | |
| Nombre de résidus | 664 acides aminés[2] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
| Gène SDHB Sous-unité B, protéine fer-soufre | ||
| Homo sapiens | ||
| Locus | 1p36.13 | |
| Masse moléculaire | 31 630 Da[2] | |
| Nombre de résidus | 280 acides aminés[2] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
| Gène SDHC Sous-unité C, protéine membranaire | ||
| Homo sapiens | ||
| Locus | 1q23.3 | |
| Masse moléculaire | 18 610 Da[2] | |
| Nombre de résidus | 169 acides aminés[2] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
| Gène SDHD Sous-unité D, protéine membranaire | ||
| Homo sapiens | ||
| Locus | 11q23.1 | |
| Masse moléculaire | 17 043 Da[2] | |
| Nombre de résidus | 159 acides aminés[2] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
La succinate déshydrogénase (SDH), ou complexe II de la chaîne respiratoire, également appelée succinate-coenzyme Q réductase (SQR), est une oxydoréductase membranaire qui catalyse la réaction :
Cette enzyme est présente chez un très grand nombre d'êtres vivants. On la trouve ainsi dans la membrane plasmique des bactéries et dans la membrane mitochondriale interne des eucaryotes, notamment des mammifères. Elle présente la particularité d'intervenir à la fois dans le cycle de Krebs et dans la chaîne de transport d'électrons de la phosphorylation oxydative[3]. Elle constitue le deuxième point d'entrée des électrons à haut potentiel de transfert dans la respiration cellulaire et assure le transfert de électrons du succinate de la matrice mitochondriale vers la coenzyme Q10 dissoute dans la bicouche lipidique. Chez l'humain un blocage de la succinate déshydrogénase est connue pour entrainer des maladies graves (maladies neurodégénératives ou des tumeurs), avec un spectre très large de symptômes typiques des maladies mitochondriales[4],[5].
| N° EC | EC |
|---|---|
| N° CAS | |
| Cofacteur(s) | FAD ; Fe-S |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
Structure et fonctionnement[modifier | modifier le code]
La plupart des succinate déshydrogénases de bactéries et de mitochondries sont constituées de quatre sous-unités : deux sous-unités hydrophiles et deux sous-unités hydrophobes. Les deux premières sont la flavoprotéine SdhA et la protéine fer-soufre SdhB, situées dans la matrice mitochondriale, tandis que les deux dernières sont désignées par SdhC et SdhD et forment l'ancrage membranaire de l'enzyme. La SdhA contient le cofacteur flavine adénine dinucléotide (FAD) ainsi que le site de liaison au succinate, tandis que la SdhB contient trois centres fer-soufre : [2Fe-2S], [4Fe-4S] et [3Fe-4S]. Il existe deux isoformes distinctes de la sous-unité SdhA chez l'homme (sous-unités Fp de type I et de type II), présentes également chez Ascaris suum (en) et Caenorhabditis elegans[6]. Les sous-unités SdhC et SdhD forment un complexe membranaire avec le cytochrome b comprenant six hélices transmembranaires, un hème b et un site de liaison à l'ubiquinone. On trouve également une molécule de cardiolipine et une molécule de phosphatidyléthanolamine occupant le volume hydrophobe situé sous l'hème dans les sous-unités SdhC et SdhD.
-
(en) Schéma des flux d'électrons à travers le complexe II (PDB 1YQ3[7]), illustrant la position des différents cofacteurs : le FAD, les trois centres fer-soufre, l'hème et l'ubiquinone. La matrice mitochondriale est en haut, la membrane mitochondriale interne est en bas.
Inhibiteurs[modifier | modifier le code]
Il existe deux classes distinctes d'inhibiteurs du complexe II : ceux qui bloquent la liaison à l'ubiquinone et ceux qui bloquent la liaison au succinate.
- Parmi les premiers, on trouve un pesticide comme la carboxine et ses dérivés (grande famille des SDHI, inhibiteur de la SDH) et le thénoyltrifluoroacétone. Ils sont utilisés en agriculture comme fongicides depuis la fin des années 1960. La carboxine a surtout été utilisée pour combattre les maladies provoquées par les basidiomycètes, telles que la rouille noire, ainsi que les maladies à Rhizoctonia, elle a ensuite été interdite pour l'utilisation en champs. Des dérivés de la carboxine ont été développés plus récemment avec un spectre plus large tels quel le boscalid, le penthiopyrad, le bixafen, le fluxapyroxad, le penflufen, l'isopyrazam, le flutolanil, le penthiopyrad, le Xemium, le fluopyram et l'isoflucypram[8]. Les inhibiteurs SDHi ne sont pas spécifique d'une espèce, ils peuvent bloquer la respiration d'organismes variés, allant des moisissures (ex: botrytis cinerea) aux vers de terres, abeilles, poissons, grenouilles et l'Homme}[9],[10],[11],[12].
Les SDHI sont des inhibiteurs qui rentrent en compétition avec les quinones ce qui aurait pour effet la production d'ions superoxyde O2.- et l'apparition d'un stress oxydant. Comme pour les antibiotiques avec les bactéries, les champignons (moisissures) sont capables de développer des résistances, ce qui rend les SDHI inefficaces avec le temps[13].
Certains mycètes importants en agriculture ne sont pas sensibles aux composés de la nouvelle génération d'inhibiteurs de la liaison du complexe II à l'ubiquinone[14].
- Parmi les seconds, on trouve des analogues du succinate tels que le malonate, mais aussi des métabolites du cycle de Krebs que sont le malate et l'oxaloacétate. Ce dernier est l'un des plus puissants inhibiteurs du complexe II, ce qui aurait un effet bénéfique en limitant la production d'ions superoxyde O2• – par transfert d'électrons inversé dans la NADH déshydrogénase (complexe I)[15].
Notes et références[modifier | modifier le code]
- (en) Victoria Yankovskaya, Rob Horsefield, Susanna Törnroth, César Luna-Chavez, Hideto Miyoshi, Christophe Léger, Bernadette Byrne, Gary Cecchini et So Iwata, « Architecture of Succinate Dehydrogenase and Reactive Oxygen Species Generation », Science, vol. 299, no 5607, , p. 700-704 (PMID 12560550, DOI 10.1126/science.1079605, Bibcode 2003Sci...299..700Y, lire en ligne)
- Les valeurs de la masse et du nombre de résidus indiquées ici sont celles du précurseur protéique issu de la traduction du gène, avant modifications post-traductionnelles, et peuvent différer significativement des valeurs correspondantes pour la protéine fonctionnelle.
- (en) Kayode S. Oyedotun et Bernard D. Lemire, « The quaternary structure of the Saccharomyces cerevisiae succinate dehydrogenase. Homology modeling, cofactor docking, and molecular dynamics simulation studies », Journal of Biological Chemistry, vol. 279, no 10, , p. 9424-9431 (PMID 14672929, DOI 10.1074/jbc.M311876200, lire en ligne)
- P.Bénit et al., Pathologies liées aux déficits du cycle de Krebs. Revue Francophone des laboratoires N° 501 2018. {{DOI: 10.1016/S1773-035X(18)30120-5}}
- S.Loublier et al., Les maladies mitochondriales : une médecine à part ? 2009. DOI 10.1016/j.immbio.2009.08.002
- (en) Eriko Tomitsuka, Hiroko Hirawake, Yu-ichi Goto, Masafumi Taniwaki, Shigeharu Harada et Kiyoshi Kita, « Direct Evidence for Two Distinct Forms of the Flavoprotein Subunit of Human Mitochondrial Complex II (Succinate-Ubiquinone Reductase) », Journal of Biochemistry, vol. 134, no 2, , p. 191-195 (PMID 12966066, DOI 10.1093/jb/mvg144, lire en ligne)
- (en) Li-shar Huang, Gang Sun, David Cobessi, Andy C. Wang, John T. Shen, Eric Y. Tung, Vernon E. Anderson et Edward A. Berry, « 3-Nitropropionic Acid Is a Suicide Inhibitor of Mitochondrial Respiration That, upon Oxidation by Complex II, Forms a Covalent Adduct with a Catalytic Base Arginine in the Active Site of the Enzyme », Journal of Biological Chemistry, vol. 281, no 9, , p. 5965-5972 (PMID 16371358, PMCID 1482830, DOI 10.1074/jbc.M511270200, lire en ligne)
- (en) Philippe Desbordes, Bernd Essigmann, Stephanie Gary et Oliver Gutbrod, « Isoflucypram, the first representative of a new succinate dehydrogenase inhibitor fungicide subclass: Its chemical discovery and unusual binding mode », Pest Management Science, vol. 76, no 10, , p. 3340–3347 (ISSN 1526-498X et 1526-4998, PMID 32506626, PMCID PMC7540001, DOI 10.1002/ps.5951, lire en ligne, consulté le )
- V. Graillot et al., Evidence of the in vitro genotoxicity of methyl-pyrazol pesticides in human cells. Mutat Res. 2012. DOI 10.1016/j.mrgentox.2012.05.014 lire en ligne
- Qian et al., Toxic effects of boscalid in adult zebrafish (Danio rerio) on carbohydrate and lipid metabolism. Environmental pollution
- S. Wu et al., Single and mixture toxicity of strobilurin and SDHI fungicides to Xenopus tropicalis embryos. Ecotoxicology and envirinmental safety
- LH Liao et al., behavioral responses of honey bees (Apis mellifera) to natural and synthetic xenobiotics in food, 2017, Sci Rep. DOI 10.1038/j.immbio.s41598-017-15066-5
- (en) Hervé F. Avenot et Themis J. Michailides, « Progress in understanding molecular mechanisms and evolution of resistance to succinate dehydrogenase inhibiting (SDHI) fungicides in phytopathogenic fungi », Crop Protection, vol. 29, no 7, , p. 643-651 (DOI 10.1016/j.cropro.2010.02.019, lire en ligne)
- (en) Tiphaine Dubos, Matias Pasquali, Friederike Pogoda, Angèle Casanova, Lucien Hoffmann et Marco Beyer, « Differences between the succinate dehydrogenase sequences of isopyrazam sensitive Zymoseptoria tritici and insensitive Fusarium graminearum strains », Pesticide Biochemistry and Physiology, vol. 105, no 1, , p. 28-35 (PMID 24238287, DOI 10.1016/j.pestbp.2012.11.004, lire en ligne)
- (en) Florian L. Muller, Yuhong Liu, Muhammad A. Abdul-Ghani, Michael S. Lustgarten, Arunabh Bhattacharya, Youngmok C. Jang et Holly Van Remmen, « High rates of superoxide production in skeletal-muscle mitochondria respiring on both complex I- and complex II-linked substrates », Biochemical Journal, vol. 409, no 2, , p. 491-499 (PMID 17916065, DOI 10.1042/BJ20071162, lire en ligne)


![(en) Schéma des flux d'électrons à travers le complexe II (PDB 1YQ3[7]), illustrant la position des différents cofacteurs : le FAD, les trois centres fer-soufre, l'hème et l'ubiquinone. La matrice mitochondriale est en haut, la membrane mitochondriale interne est en bas.](http://upload.wikimedia.org/wikipedia/commons/thumb/9/97/Succinate_Dehydrogenase_1YQ3_Electron_Carriers_Labeled.png/660px-Succinate_Dehydrogenase_1YQ3_Electron_Carriers_Labeled.png)