Complexe alpha-cétoglutarate déshydrogénase
| N° EC | EC |
|---|---|
| N° CAS | |
| Cofacteur(s) | TPP |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
| N° EC | EC |
|---|---|
| N° CAS |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
| N° EC | EC |
|---|---|
| N° CAS | |
| Cofacteur(s) | FAD |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
Le complexe α-cétoglutarate déshydrogénase, également appelé complexe oxoglutarate déshydrogénase (OGDC), est l'association de trois enzymes — une décarboxylase, une acyltransférase et une oxydo-réductase — agissant séquentiellement dans cet ordre pour catalyser la réaction :

+ CoA-SH + NAD+ → NADH + CO2 + 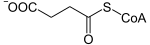
α-Cétoglutarate Succinyl-CoA
Ce complexe enzymatique intervient dans trois voies métaboliques : le cycle de Krebs, la dégradation de la lysine et le métabolisme du tryptophane. Il est structurellement apparenté au complexe pyruvate déshydrogénase et au complexe 3-méthyl-2-oxobutanoate déshydrogénase. Les trois enzymes constituant le complexe sont les suivantes :
Le mécanisme de cette réaction, qui fait intervenir successivement les enzymes E1, E2 et E3, chacune avec ses cofacteurs, est assez complexe, et peut être résumé par le schéma simplifié ci-dessous :

Mécanisme réactionnel du complexe α-cétoglutarate déshydrogénase (R = CH2-COO− sur ce schéma) :
- l'α-cétoglutarate déshydrogénase (E1) catalyse les étapes A et B avec la thiamine pyrophosphate (TPP),
- la dihydrolipoamide S-succinyltransférase (E2) catalyse l'étape C avec le lipoamide et la coenzyme A (CoA-SH),
- la dihydrolipoyl déshydrogénase (E3) catalyse les étapes D et E avec le FAD et le NAD+.
Régulation[modifier | modifier le code]
Le complexe α-cétoglutarate déshydrogénase est un point de régulation essentiel du cycle de Krebs. Il est inhibé par ses produits de réaction, c'est-à-dire par la succinyl-CoA et le NADH, et lorsque la charge énergétique de la cellule est élevée. En particulier, il est inhibé lorsque les ratios de concentrations [ATP] / [ADP], [NADH] / [NAD+] et [succinyl-CoA] / [CoA] sont élevés[4]. A contrario, l'ADP et les cations de magnésium Mg2+ et de calcium Ca2+ sont des activateurs allostériques du complexe[5]. Ces effecteurs allostériques agissent essentiellement sur l'enzyme E1, c'est-à-dire l'α-cétoglutarate déshydrogénase elle-même, mais les deux autres enzymes sont également susceptibles de régulation allostérique[4].
La régulation du complexe α-cétoglutarate déshydrogénase agit par ricochet sur la phosphorylation oxydative et la production d'ATP en modulant la quantité de coenzymes réduites, et notamment de NADH, présentes dans les cellules, et, plus précisément, dans les mitochondries chez les eucaryotes[6] : l'activation du complexe α-cétoglutarate déshydrogénase accélère la phosphorylation oxydative et accroît la production d'ATP en augmentant le flux d'électrons injectés dans la chaîne respiratoire à partir du NADH.
En activant la chaîne respiratoire, le complexe α-cétoglutarate déshydrogénase accroît la production de radicaux libres, ce qui peut conduire au stress oxydant. Ce complexe enzymatique est vraisemblablement capable de réagir à l'augmentation des radicaux libres dans la cellule en réduisant son activité, ce qui permet de limiter les dommages induits par les dérivés réactifs de l'oxygène[7]. Plus précisément, il peut subir une inhibition entièrement réversible en présence d'une forte concentration de radicaux libres, inhibition qui peut être totale dans les cas extrêmes[8]. On pense que l'inhibition transitoire du complexe mitochondrial provient de la glutathionylation (en) réversible du domaine de liaison à l'acide lipoïque de l'enzyme E2, c'est-à-dire de la dihydrolipoamide S-succinyltransférase[9]. La glutathionylation est une modification post-traductionnelle qui survient notamment en présence de radicaux libres et peut être levée sous l'effet antioxydant de la glutarédoxine[8]. Cette modification a pour effet de protéger l'acide lipoïque de l'oxydation.
Réponse au stress[modifier | modifier le code]
Le complexe α-cétoglutarate déshydrogénase joue un rôle dans la réponse cellulaire à l'exposition aiguë au stress. Son inhibition temporaire est en effet suivie d'un surcroît d'activité lorsqu'elle est levée, ce qui permet de compenser après-coup l'exposition à un stress aigu par une suractivité du complexe α-cétoglutarate déshydrogénase[10].
Les effets d'une exposition aiguë au stress sont généralement mieux tolérés par les cellules que ceux d'une exposition prolongée ou chronique, lorsque ces effets s'accumulent. La réactivation du complexe α-cétoglutarate déshydrogénase suivant une inhibition due au stress peut s'épuiser lorsque l'inhibition devient trop importante[10]. En particulier, le stress cellulaire peut conduire à un dérèglement de la biosynthèse du glutamate, qui, outre son rôle structurel comme acide aminé protéinogène, est également un neurotransmetteur. La neurotoxicité du glutamate dans le cerveau résulte de son accumulation à la suite d'épisodes de stress trop fréquents ou trop prolongés. Cette accumulation ne peut être résorbée lorsque la réaction du complexe α-cétoglutarate déshydrogénase cesse d'être efficace, et c'est là que les pathologies peuvent se développer.
Une complexe α-cétoglutarate déshydrogénase défectueux peut également sensibiliser la cellule à d'autres toxines susceptibles d'entraîner une neurodégénérescence[11].
Pathologie[modifier | modifier le code]
Dans la maladie métabolique de l'acidurie combinée malonique et méthylmalonique (CMAMMA) due à un déficit en ACSF3, la synthèse mitochondriale des acides gras (mtFASII) est altérée, ce qui constitue la réaction précurseur de la biosynthèse de l'acide lipoïque[12],[13]. Il en résulte un degré de lipoylation réduit d'enzymes mitochondriales importantes, telles que le complexe α-cétoglutarate déshydrogénase[13].
Notes et références[modifier | modifier le code]
- (en) René A.W. Frank, Amanda J. Price, Fred D. Northrop, Richard N. Perham et Ben F. Luisi, « Crystal Structure of the E1 Component of the Escherichia coli 2-Oxoglutarate Dehydrogenase Multienzyme Complex », Journal of Molecular Biology, vol. 368, no 3, , p. 639-651 (PMID 17367808, DOI 10.1016/j.jmb.2007.01.080, lire en ligne)
- (en) James E. Knapp, Donald Carroll, Janet E. Lawson, Stephen R. Ernst, Lester J. Reed et Marvin L. Hackert, « xpression, purification, and structural analysis of the trimeric form of the catalytic domain of the Escherichia coli dihydrolipoamide succinyltransferase », Protein Science, vol. 9, no 1, , p. 37-48 (PMID 10739245, PMCID 2144448, DOI 10.1110/ps.9.1.37, lire en ligne)
- (en) Ewa M. Ciszak, Anna Makal, Young S. Hong, Ananthalakshmy K. Vettaikkorumakankauv, Lioubov G. Korotchkina et Mulchand S. Patel, « How Dihydrolipoamide Dehydrogenase-binding Protein Binds Dihydrolipoamide Dehydrogenase in the Human Pyruvate Dehydrogenase Complex », Journal of Biological Chemistry, vol. 281, no 1, , p. 648-655 (PMID 16263718, DOI 10.1074/jbc.M507850200, lire en ligne)
- (en) Slawomir Strumilo, « Often ignored facts about the control of the 2-oxoglutarate dehydrogenase complex », Biochemistry and Molecular Biology Education, vol. 33, no 4, , p. 284-287 (DOI 10.1002/bmb.2005.49403304284, lire en ligne)
- (en) Feng Qi, Ranjan K. Pradhan, Ranjan K. Dash et Daniel A. Beard, « Detailed kinetics and regulation of mammalian 2-oxoglutarate dehydrogenase », BMC Biochemistry, vol. 12, , p. 53 (PMID 21943256, PMCID 3195097, DOI 10.1186/1471-2091-12-53, lire en ligne)
- (en) Laszlo Tretter et Vera Adam-Vizi, « Alpha-ketoglutarate dehydrogenase: a target and generator of oxidative stress », Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences, vol. 360, no 1464, , p. 2335-2345 (PMID 16321804, PMCID 1569585, DOI 10.1098/rstb.2005.1764, JSTOR 30041424, lire en ligne)
- (en) Aaron L. McLain, Pamela A. Szweda et Luke I. Szweda, « α-Ketoglutarate dehydrogenase: A mitochondrial redox sensor », Free Radical Research, vol. 45, no 1, , p. 29-36 (PMID 21110783, PMCID 3169906, DOI 10.3109/10715762.2010.534163, lire en ligne)
- (en) Aaron L. McLain, Peter J. Cormier, Michael Kinter et Luke I. Szweda, « Glutathionylation of α-ketoglutarate dehydrogenase: The chemical nature and relative susceptibility of the cofactor lipoic acid to modification », Free Radical Biology and Medicine, vol. 61, , p. 161-169 (PMID 23567190, PMCID 3883985, DOI 10.1016/j.freeradbiomed.2013.03.020, lire en ligne)
- (en) Milana A. B. Applegate, Kenneth M. Humphries et Luke I. Szweda, « Reversible Inhibition of α-Ketoglutarate Dehydrogenase by Hydrogen Peroxide: Glutathionylation and Protection of Lipoic Acid », Biochemistry, vol. 47, no 1, , p. 473-478 (PMID 18081316, DOI 10.1021/bi7017464, lire en ligne)
- (en) Anastasia Graf, Lidia Trofimova, Alexandra Loshinskaja, Garik Mkrtchyan, Anastasiia Strokina, Maxim Lovat, Adam Tylicky, Slawomir Strumilo, Lucien Bettendorff et Victoria I. Bunik, « Up-regulation of 2-oxoglutarate dehydrogenase as a stress response », The International Journal of Biochemistry & Cell Biology, vol. 45, no 1, , p. 175-189 (PMID 22814169, DOI 10.1016/j.biocel.2012.07.002, lire en ligne)
- (en) Gary E. Gibson, John P. Blass, M. Flint Beal et Victoria Bunik, « The α-ketoglutarate-dehydrogenase complex: a mediator between mitochondria and oxidative stress in neurodegeneration », Molecular Neurobiology, vol. 31, no 1, , p. 43-63 (PMID 15953811, DOI 10.1385/MN:31:1-3:043, lire en ligne)
- (en) Alina Levtova, Paula J. Waters, Daniela Buhas et Sébastien Lévesque, « Combined malonic and methylmalonic aciduria due to ACSF3 mutations: Benign clinical course in an unselected cohort », Journal of Inherited Metabolic Disease, vol. 42, no 1, , p. 107–116 (ISSN 0141-8955 et 1573-2665, DOI 10.1002/jimd.12032, lire en ligne)
- (en) Zeinab Wehbe, Sidney Behringer, Khaled Alatibi et David Watkins, « The emerging role of the mitochondrial fatty-acid synthase (mtFASII) in the regulation of energy metabolism », Biochimica et Biophysica Acta (BBA) - Molecular and Cell Biology of Lipids, vol. 1864, no 11, , p. 1629–1643 (DOI 10.1016/j.bbalip.2019.07.012, lire en ligne)




