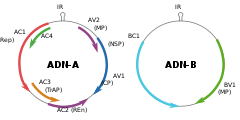Virus New Delhi des feuilles enroulées de la tomate
Begomovirus solanumdelhiense
| Groupe | Groupe II |
|---|---|
| Famille | Geminiviridae |
| Genre | Begomovirus |
Begomovirus solanumdelhiense, appelé le virus New Delhi des feuilles enroulées de la tomate (ToLCNDV, Tomato leaf curl New Delhi virus), est un phytovirus du genre Begomovirus (famille des Geminiviridae) originaire du sous-continent indien. C'est un virus à ADN à simple brin, dont le génome circulaire, bipartite, est constitué de deux composants (ADN-A et ADN-B) d'environ 2,5 à 2,7 kb. Le composant ADN-A peut se répliquer de manière autonome, cependant le composant ADN-B est nécessaire pour l'infection systémique des plantes[2].
Le virus New Delhi de la tomate est transmis principalement par l'aleurode du tabac, (Bemisia tabaci) selon le mode circulant et persistant[3]. Ce virus, isolé pour la première fois en 1995 sur la tomate, en Inde, affecte gravement les cultures de courgettes et concombres dans plusieurs pays méditerranéens depuis les années 2010.
Ce virus émergent est devenu en Inde l'agent pathogène le plus important pour les cultures de tomates. En quelques années, il a étendu de façon importante son aire de diffusion, jusqu'au Moyen-Orient et à la Méditerranée occidentale, et sa gamme d'hôtes qui comprend plus de 40 espèces, parmi lesquelles des cultures économiquement très importantes comme la tomate, la courgette, le soja, la pomme de terre ou le tabac[4]. Le ToLCNDV est associé dans des infections naturelles avec des ADN satellites (satellites alpha ou béta) qui peuvent moduler sa pathogenèse, y compris sa gamme d'hôtes[4].
Génome
[modifier | modifier le code]Le ToLCNDV est un bégomovirus, virus à ADN à simple brin. Il se caractérise par son génome bipartite, c'est-à-dire qu'il possède deux composants génomiques, nommés ADN-A et ADN-B, longs chacun d'environ 2800 nucléotides. L'ADN-A contient les gènes AV1 et AV2 dans le sens du brin viral (orientation positive) et AC1, AC2, AC3 et AC4 dans le sens complémentaire (orientation négative). L'ADN-B contient le gène BV1 dans le sens du brin viral et le gène BC1 dans le sens complémentaire. Dans les deux molécules (ADN-A et ADN-B), les gènes orientés dans le sens positif et dans le sens négatif sont séparés par une région intergénique contenant une région commune (CR) composée des mêmes séquences de nucléotides dans l'ADN-A et l'ADN-B. La principale caractéristique topologique de la région commune est une structure en épingle à cheveux avec un nonanucléotide (TAATATT/AC) qui couvre l'origine de réplication du brin viral (v-ori)[5],[6].
L'ADN-A du ToLCNDV code des protéines impliquées dans la réplication du virus : protéine Rep (replication‐associated protein,) d'initiation de la réplication (AC1), protéine REn (replication enhancer protein) activatrice de réplication (AC2), protéine TrAP (transcriptional activator protein) d'activation de la transcription (AC3). Le gène AV1 code une protéine d'enveloppe impliquée dans la transmission par les insectes. Les fonctions précises des deux gènes AV2 et AC4 restent incertaines mais ils sont impliqués dans le mouvement viral (AV2) et dans la pathogénicité (AC4).
L'ADN-B code une protéine de mouvement MP (BV1) et une protéine nucléaire navette NSP (BC1), impliquées dans les mouvements inter- et intracellulaires, respectivement[5],[6].
| Segment | Gène | Sens | Protéine codée | Fonction |
|---|---|---|---|---|
| ADN-A | AV1 | + | CP | protéine de capside, joue un rôle dans la transmission et le mouvement |
| AV2 | + | MP | protéine de mouvement | |
| AC1 | - | Rep | protéine de réplication du virus, interaction spécifique avec des séquences d'ADN de la région intergénique | |
| AC2 | - | REn | protéine activatrice de réplication | |
| AC3 | - | TrAP | protéine d'activation de la transcription | |
| AC4 | - | agit au niveau de l’expression des symptômes et du mouvement | ||
| ADN-B | BV1 | + | MP | protéine de mouvement |
| BC1 | - | NSP | protéine nucléaire navette |
Plantes-hôtes
[modifier | modifier le code]Le ToLCNDV affecte une large gamme de plantes-hôtes, principalement dans les familles des Solanaceae et Cucurbitaceae, et le nombre d'hôtes nouveaux pourrait augmenter. Parmi les hôtes signalés figurent notamment les suivants[2] :
- Solanaceae : Capsicum annuum (piment), Cestrum nocturnum (jasmin de nuit), Datura stramonium (datura), Nicotiana tabacum (tabac), Solanum lycopersicum (tomate), Solanum melongena (aubergine), Solanum nigrum (morelle noire), Solanum tuberosum (pomme de terre) ;
- Cucurbitaceae : Benincasa hispida (courge cireuse), Citrullus lanatus (pastèque), Coccinia grandis (courge écarlate), Cucumis melo (melon), Cucumis sativus (concombre), Cucurbita ficifolia (courge de Siam), Cucurbita maxima (potiron), Cucurbita moschata (courge musquée), Cucurbita pepo (courgette), Ecballium elaterium (concombre d'âne), Lagenaria siceraria (calebasse), Luffa aegyptiaca (luffa), Momordica charantia (melon amer), Trichosanthes cucumerina (concombre serpent) ;
- Malvaceae : Abelmoschus esculentus (gombo), Gossypium hirsutum (coton mexicain), Hibiscus cannabinus (chanvre du Deccan) ;
- Asteraceae : Eclipta prostrata (eclipta), Parthenium hysterophorus (fausse camomille), Sonchus oleraceus (laiteron) ;
- Autres familles : Daucus carota (carotte), Calotropis procera (pommier de Sodome), Catharanthus roseus (pervenche de Madagascar), Carica papaya (papayer), Chenopodium album (chénopode blanc), Convolvulus arvensis (liseron des champs), Sauropus androgynus, Cyamopsis tetragonoloba (guar), Glycine max (soja), Jasminum multiflorum (faux jasmin), Papaver somniferum (pavot somnifère), Saccharum edule, Rumex dentatus.
Symptômes
[modifier | modifier le code]Les symptômes d'une infection par le virus New Delhi de la tomate varient selon l'espèce de plante-hôte et les conditions de croissance. Parmi les symptômes courants, on peut noter un rabougrissement et une perte de vigueur très prononcée des plantes.
Chez la tomate, les plantes infectées présentent des symptômes typiques, notamment l'enroulement du bord des feuilles vers le haut ou vers le bas et le gaufrage du limbe, le jaunissement des nervures, des marbrures jaunes, des cloques sur les feuilles. Aux derniers stades de l'infection, les plantes paraissent rabougries avec des entre-nœuds raccourcis et présentent une nouaison altérée, entraînant la perte de la récolte[4].
Chez les courgettes et les melons, les jeunes feuilles montrent des symptômes d'enroulement et de recroquevillement, ainsi qu'un jaunissement internervaire avec le gonflement des nervures foliaires chez les concombres, et chez les melons une mosaïque sévère, qui peut affecter négativement la productivité et donc le rendement, ainsi que la qualité des produits. Il est probable que la gravité des symptômes dépende des espèces végétales, de l'environnement, des conditions de croissance et des infections mixtes potentielles avec d'autres virus végétaux, et même de la présence de bétasatellites associés au ToLCNDV. Cependant aucun bétasatellite n'a encore été trouvé dans les cultures de cucurbitacées du bassin méditerranéen[2],[7].
Distribution
[modifier | modifier le code]Détecté d'abord en Inde en 1995, le ToLCNDV s'est largement répandu au cours des années suivantes dans le Sud de l'Asie, principalement chez les Solanaceae et les Cucurbitaceae, dans le sous-continent indien (Bangla Desh, Sri Lanka, Pakistan), à l'Est (Thaïlande, Malaisie, Indonésie, Philippines, Taïwan) et à l'ouest (Iran). Le virus a été détecté en Iran en 2012 dans des cultures de melon (Cucumis melo)[8]. Il est ensuite apparu dans le bassin méditerranéen, où il a été détecté pour la première fois en Espagne en 2012 et les années suivantes dans d'autres pays comme l'Italie, la Tunisie, le Maroc et la Grèce. Il est également présent, en septembre 2020, en Afrique du Nord, aux Seychelles, au Portugal, dans les pays baltes (Lituanie, Estonie) et en Slovénie[2]. Le virus a été signalé pour la première fois dans le Sud de la France (régions PACA et Occitanie) en septembre 2020 dans des cultures de courgettes.
L'examen de séquences nucléotidiques a montré que la structure génétique des populations de ToLCNDV dans le bassin méditerranéen semble être génétiquement homogène avec des isolats appartenant au génotype nommé ToLCNDV-ES et assez différenciée des isolats de ToLCNDV signalés dans le sous-continent indien. Cela suggère qu'il est possible que cette population virale envahissante provienne d'une seule introduction[2]. La souche la plus répandue en Europe (ToLCNDV-ES) est particulièrement adaptée aux Cucurbitaceae, alors que les isolats signalés en Asie sont mieux adaptés aux cultures de Solanaceae. Ces derniers pourraient donc présenter un risque supplémentaire pour l'agriculture européenne[9].
Transmission
[modifier | modifier le code]La transmission horizontale (de plante à plante) de ce virus se fait principalement par un insecte vecteur hémiptère, Bemisia tabaci (l'aleurode du tabac ou mouche blanche de la patate douce). La transmission se fait selon le mode circulant et persistant, ce qui signifie que l'insecte vecteur fait l'acquisition du virus et peut rester virulifère tout au long de sa vie. La dispersion du ToLCNDV est associée au mouvement du vecteur. Localement, les aleurodes se déplacent rarement d'une plante à l'autre. A longue distance, leur dispersion peut être facilitée par le vent et l'activité humaine de propagation du matériel végétal. Il est possible que les populations d'aleurodes passent d'une culture à une autre, en particulier lorsqu'une culture est récoltée ou abandonnée[2],[7].
La transmission par les semences contaminées a été décrite pour ce virus au moins sur courgette (Cucurbita pepo, mais ce sujet reste controversé. Toutefois, ce mode de transmission par les semences a été signalé récemment chez la tomate pour une espèce de virus proche, le TYLCV (virus des feuilles jaunes en cuillère de la tomate), et des isolats proches se sont révélés transmissibles par les semences. L'expansion de la maladie peut aussi résulter du transport et de l'utilisation de plants infectés[7].
Le virus est limité au phloème végétal et Les plantes hôtes peuvent être infectées expérimentalement par inoculation mécanique[2].
Méthodes de lutte
[modifier | modifier le code]Comme c'est le cas de tous les phytovirus, il n'existe pas de méthode de lutte directe permettant de guérir les plantes infectées. La lutte repose essentiellement sur les traitements insecticides contre les insectes-vecteurs (aleurodes du tabac) ou sur la culture en serres étanches aux insectes, ainsi que sur des méthodes de prévention sanitaire (choix de plants sains, élimination des plantes infectées).
Il est nécessaire aussi d'éviter les cultivars les plus sensibles, mais on n'a pas encore (en 2020) identifié des cultivars résistants ou tolérants au ToLCNDV[10]. Toutefois, à ce jour (2020), quatre accessions seulement de Cucurbita moschata se sont révélées asymptomatiques ou présentant des symptômes bénins après une infection au ToLCNDV, et pourraient être des candidats potentiels comme sources de résistance au ToLCNDV chez les Cucurbitaceae. D'autre part, 13 accessions de tomate contenant des gènes Ty ont montré une résistance au ToLCNDV, avec une période de latence retardée, des symptômes bénins et une faible accumulation de virus en comparaison avec des génotypes sensibles. Cependant, ces accessions ne peuvent pas être utilisée directement en culture en raison de leurs caractéristiques agronomiques inacceptables[2],[11].
Notes et références
[modifier | modifier le code]- (en) « Virus Taxonomy: 2023 Release », ICTV, (consulté le ).
- (en) « Tomato leaf curl New Delhi virus (Tomato New Delhi virus) », sur Invasive Species Compendium (ISC), CABI (consulté le ).
- P. Gentit, « Le Tomato leaf curl New Delhi virus(ToLCNDV) », sur Direction régionale de l’alimentation, de l’agriculture et de la forêt (DRAAF) - PACA (consulté le ).
- (en) Enrique Moriones, Shelly Praveen & Supriya Chakraborty, « Tomato Leaf Curl New Delhi Virus: An Emerging Virus Complex Threatening Vegetable and Fiber Crops », Viruses, vol. 9, no 10, , p. 264 (DOI 10.3390/v9100264, lire en ligne).
- (en) Mazhar Hussain, Shahid Mansoor, Shazia Iram, Yusuf Zafar, and Rob W. Briddon, « The Hypersensitive Response to Tomato leaf curl New Delhi virus Nuclear Shuttle Protein Is Inhibited by Transcriptional Activator Protein », Molecular Plant-Microbe Interactions, vol. 20, no 12, , p. 1581–1588 (DOI doi:10.1094 / MPMI -20-12-1581, lire en ligne).
- (en) Zaidi SS, Martin DP, Amin I, Farooq M, « Tomato leaf curl New Delhi virus: a widespread bipartite begomovirus in the territory of monopartite begomoviruses », Molecular Plant Pathology, vol. 18, no 7, , p. 901-911 (PMID 27553982, PMCID PMC6638225, DOI 10.1111/mpp.12481, lire en ligne).
- « Tomato leaf curl New Delhi virus (ToLCNDV) - Alerte : premier signalement du ToLCNDV en France en septembre 2020 », sur E-phytia légumes, INRA (consulté le ).
- (en) Sara Yazdani-Khameneh, Samaneh Aboutorabi, Majid Shoori, Azin Aghazadeh, Parastoo Jahanshahi, Alireza Golnaraghi & Mojdeh Maleki, « Natural Occurrence of Tomato leaf curl New Delhi virus in Iranian Cucurbit Crops », Plant Pathol J., vol. 32, no 3, , p. 201–208 (DOI 10.5423/PPJ.OA.10.2015.0210, lire en ligne).
- (en) EFSA Panel on Plant Health (PLH), « Pest categorisation of tomato leaf curl New Delhi virus », EFSA Journal, John Wiley and Sons / European Food Safety Authority (EFSA), vol. 18, no 7, (lire en ligne).
- (en) « EPPO Alert List – Tomato leaf curl New Delhi virus », OEPP (consulté le ).
- (en) Akhtar, K. P., Afzal Akram, Najeeb Ullah, Saleem, M. Y., Muhammad Saeed, « Evaluation of Solanum species for resistance to Tomato leaf curl New Delhi virus using chip grafting assay », Scientia Horticulturae volume= 256, Elsevier B.V., (DOI 10.1016/j.scienta.2019.108646, lire en ligne).
Voir aussi
[modifier | modifier le code]Articles connexes
[modifier | modifier le code]Liens externes
[modifier | modifier le code]- Ressources relatives au vivant :
- (en) Référence NCBI : Tomato leaf curl New Delhi virus (taxons inclus) (consulté le )
- (en) EPPO Alert List – Tomato leaf curl New Delhi virus, OEPP.
- (fr) Tomato leaf curl New Delhi virus (ToLCNDV) - Alerte : premier signalement du ToLCNDV en France en septembre 2020 , INRA e-phytia Légumes.