Répétitions en tandem

En biochimie, les répétitions en tandem sont l'association, dans une protéine, d'au moins deux occurrences de motifs séquentiels ou de motifs structuraux semblables. Ces redondances sont générées par des duplications internes dans les séquences génomiques codantes et non codantes. La nature des unités ainsi répétées en tandem peut être extrêmement diverse, allant de la répétition d'un seul résidu d'acide aminé à la répétition de domaines de plus d'une centaine de résidus[1],[2].
-
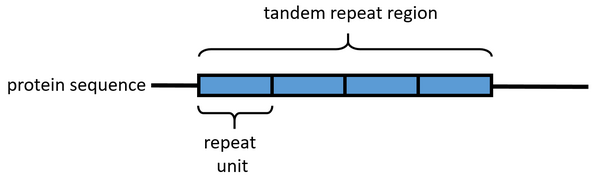
Représentation schématique d'une séquence protéique de répétitions en tandem.
Les répétitions en tandem sont omniprésentes dans les protéomes et se retrouvent dans au moins 14 % de toutes les protéines[3]. On les retrouve notamment dans près du tiers des protéines humaines et la moitié des protéines de Plasmodium falciparum et de Dictyostelium discoideum[3],[4]. Les répétitions en tandem d'unités courtes sont les plus fréquentes, notamment les répétitions d'un même résidu[3].
Environ la moitié des répétitions en tandem sont des régions ayant une conformation intrinsèquement désordonnée et naturellement dépliée[5],[6],[7], tandis que l'autre moitié présente une structure tridimensionnelle stable ayant une grande variété de formes et de fonctions[8],[9].
Les structures des répétitions en tandem peuvent être rangées dans cinq classes en fonction de la longueur des unités répétées[8] :
- agrégats cristallins formés par des répétitions d'unités d'un ou deux résidus, typiques de régions de faible complexité ;
- structures fibreuses stabilisées par des interactions inter-chaînes formées par des répétitions d'unités de trois à sept résidus ;
- structures linéaires formées de répétitions d'unités de 5 à 40 résidus prédominantes dans les domaines en solénoïde ;
- structures fermées formées de répétitions d'unités de 30 à 60 résidus ;
- structure en collier de perles formées de répétitions d'unités de plus de 50 résidus, suffisamment grandes pour se replier chacune en domaines stables.
Les protéines et structures suivantes sont des exemples bien connus ayant des répétitions en tandem : le collagène, qui joue un rôle clé dans l'arrangement de la matrice extracellulaire animale ; les superhélices, qui portent des fonctions structurelles et d'oligomérisation ; les répétitions riches en leucine, qui se lient spécifiquement à un certain nombre de protéines globulaires par leurs surfaces concaves ; les doigts de zinc, qui régulent l'expression de gènes en se liant à l'ADN.
Notes et références[modifier | modifier le code]
- (en) Jaap Heringa, « Detection of internal repeats: how common are they? », Current Opinion in Structural Biology, vol. 8, no 3, , p. 338-345 (PMID 9666330, DOI 10.1016/S0959-440X(98)80068-7, lire en ligne)
- (en) Miguel A. Andrade, Chris P. Ponting, Toby J. Gibson et Peer Bork, « Homology-based method for identification of protein repeats using statistical significance estimates », Journal of Molecular Biology, vol. 298, no 3, , p. 521-537 (PMID 10772867, DOI 10.1006/jmbi.2000.3684, lire en ligne)
- (en) Edward M. Marcotte, Matteo Pellegrini, Todd O. Yeates et David Eisenberg, « A census of protein repeats », Journal of Molecular Biology, vol. 293, no 1, , p. 151-160 (PMID 10512723, DOI 10.1006/jmbi.1999.3136, lire en ligne)
- (en) Marco Pellegrini, « Tandem repeats in proteins: prediction algorithms and biological role », Frontiers in Bioengineering and Biotechnology, vol. 3, , article no 143 (PMID 26442257, PMCID 4585158, DOI 10.3389/fbioe.2015.00143, lire en ligne)
- (en) Peter Tompa, « Intrinsically unstructured proteins evolve by repeat expansion », BioEssays, vol. 25, no 9, , p. 847-855 (PMID 12938174, DOI 10.1002/bies.10324, lire en ligne)
- (en) Michelle Simon et John M Hancock, « Tandem and cryptic amino acid repeats accumulate in disordered regions of proteins », Genome Biology, vol. 10, no 6, , article no R59 (PMID 19486509, PMCID 2718493, DOI 10.1186/gb-2009-10-6-r59, lire en ligne)
- (en) Julien Jorda, Bin Xue, Vladimir N. Uversky et Andrey V. Kajava, « Protein tandem repeats – the more perfect, the less structured », The FEBS Journal, vol. 277, no 12, , p. 2673-2682 (PMID 20553501, PMCID 2928880, DOI 10.1111/j.1742-464X.2010.07684.x, lire en ligne)
- (en) Andrey V. Kajava, « Tandem repeats in proteins: From sequence to structure », Journal of Structural Biology, vol. 179, no 3, , p. 279-288 (PMID 21884799, DOI 10.1016/j.jsb.2011.08.009, lire en ligne)