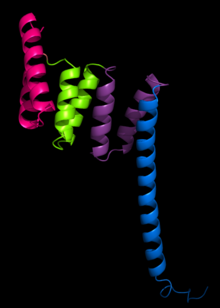
Tétratricopeptide
| Pfam | PF00515 |
|---|---|
| Clan Pfam | CL0020 |
| InterPro | IPR001440 |
| SCOP | 1a17 |
| SUPERFAMILY | 1a17 |
| CDD | cd00189 |

Une répétition tétratricopeptide (TPR), ou domaine tétratricopeptide, est un motif structurel présent dans une grande variété de protéines. Il consiste en une séquence de 34 résidus d'acides aminés organisée en tandem d'hélices α répétés de 3 à 16 fois[1] formant l'échafaudage structurant les interactions protéine-protéine et, souvent, l'assemblage de complexes protéiques. Ces répétitions forment un domaine solénoïde appelé domaine TPR. Parmi les protéines présentant de tels domaines, on trouve les sous-unités CDC16 (en), CDC23 (en) et CDC27 (en) du complexe de promotion de l'anaphase (APC), la sous-unité p67phox (en) de la NADPH oxydase, les immunophilines de liaison à la protéine Hsp90, les facteurs de transcription, l'inhibiteur de la protéine kinase R (en), le récepteur du signal de cible peroxysomale 1 (en) et les protéines d'importation mitochondriale.
Structure[modifier | modifier le code]
La première structure à avoir été déterminée est celle de la protéine phosphatase 5 (en). Sa résolution par cristallographie aux rayons X a permis d'identifier une paire d'hélices α antiparallèles[2]. Cette structure contient une répétition TPR de trois tandems formant une structure en solénoïde α.
Une structure TPR typiques est caractérisée par des interactions entre les hélices A et B du premier motif et l'hélice A’ du motif TPR suivant. Bien que la nature de ces interactions soit variable, les deux premières hélices du motif TPR présentent typiquement un angle d'empilement d'environ 24° au sein d'un motif isolé. Les répétitions de plus de trois motifs TPR produisent des superhélices droites caractérisées par une face convexe et une face concave, cette dernière intervenant généralement dans la liaison avec un ligand[1],[3].
En ce qui concerne la séquence peptidique de répétitions tétratricopeptide, on observe un mélange de grands et de petits résidus hydrophobes sans qu'aucune position soit totalement invariante. Certains résidus sont cependant généralement conservés, comme le tryptophane 4, la leucine 7, la glycine 8, la tyrosine 11, l'alanine 20, la phénylalanine 24, l'alanine 27 et la proline 32. Parmi ceux-ci, les résidus aux positions 8, 20 et 27 tendent à être davantage conservés. Plutôt qu'être occupées par un résidu spécifique, les autres positions sont occupées par un résidu préférentiellement petit, grand ou aromatique. Les résidus situés entre les hélices jouent un rôle essentiellement structurel tandis que les résidus situés entre motifs TPR adjacents jouent un rôle à la fois structurel et fonctionnel[1].
Peptides contenant le motif TPR[modifier | modifier le code]
La protéine adaptatrice Hop (en) assure l'association des protéines chaperonnes Hsp70 et Hsp90. Elle contient trois répétitions TPR ayant chacune sa spécificité de liaison particulière pour des peptides donnés. Le domaine TPR1 reconnaît l'extrémité C-terminale de Hsp70 tandis que le domaine TPR2 se lie au domaine C-terminal de Hsp90. Ces deux séquences C-terminales se terminent par un motif Glu–Glu–Val–Asp (EEVD) et les interactions sont de nature à la fois électrostatique et hydrophobe[1],[4].
La protéine PEX5 (en) interagit avec signal de cible peroxysomale à travers des motifs PTR : la plupart de ses contacts avec l'extrémité C-terminale du tripeptide signal se font avec la face concave des motifs TPR1, TPR2 et TPR3[5],[6].
La protéine NCF2 (en) (facteur cytosolique neutrophile 2) est une sous-unité indispensable de la NADPH oxydase dont les motifs TPR assurent l'assemblage avec les autres sous-unités du complexe enzymatique[7].
Notes et références[modifier | modifier le code]
- (en) Gregory L. Blatch et Michael Lässle, « The tetratricopeptide repeat: a structural motif mediating protein-protein interactions », BioEssays, vol. 21, no 11, , p. 932-939 (PMID 10517866, DOI 10.1002/(SICI)1521-1878(199911)21:11<932::AID-BIES5>3.0.CO;2-N, lire en ligne)
- (en) Amit K. Das, Patricia T. W. Cohen et David Barford, « The structure of the tetratricopeptide repeats of protein phosphatase 5: implications for TPR‐mediated protein–protein interactions », The EMBO Journal, vol. 17, no 5, , p. 1183-1552 (PMID 9482716, PMCID 1170467, DOI 10.1093/emboj/17.5.1192, lire en ligne)
- (en) Christopher G. M. Wilson, Tommi Kajander et Lynne Regan, « The crystal structure of NlpI. A prokaryotic tetratricopeptide repeat protein with a globular fold », The FEBS Journal, vol. 272, no 1, , p. 166-179 (PMID 15634341, DOI 10.1111/j.1432-1033.2004.04397.x, lire en ligne)
- (en) Clemens Scheufler, Achim Brinker, Gleb Bourenkov, Stefano Pegoraro, Luis Moroder, Hans Bartunik, F.Ulrich Hartl et Ismail Moarefi, « Structure of TPR Domain–Peptide Complexes. Critical Elements in the Assembly of the Hsp70–Hsp90 Multichaperone Machine », Cell, vol. 101, no 2, , p. 199-210 (PMID 10786835, DOI 10.1016/S0092-8674(00)80830-2, lire en ligne)
- (en) Gregory J. Gatto Jr., Brian V. Geisbrecht, Stephen J. Gould et Jeremy M. Berg, « Corrigendum: Peroxisomal targeting signal-1 recognition by the TPR domains of human PEX5 », Nature Structural Biology, vol. 9, , p. 788 (DOI 10.1038/nsb1002-788b, lire en ligne)
- (en) Gregory J. Gatto Jr., Brian V. Geisbrecht, Stephen J. Gould et Jeremy M. Berg, « Peroxisomal targeting signal-1 recognition by the TPR domains of human PEX5 », Nature Structural Biology, vol. 7, no 12, , p. 1091-1095 (PMID 11101887, DOI 10.1038/81930, lire en ligne)
- (en) Karine Lapouge, Susan J. M. Smith, Philip A. Walker, Steven J. Gamblin, Stephen J. Smerdon et Katrin Rittinger, « Structure of the TPR Domain of p67phox in Complex with Rac·GTP », Molecular Cell, vol. 6, no 4, , p. 899-907 (PMID 11090627, DOI 10.1016/S1097-2765(05)00091-2, lire en ligne)