ADP-ribosylation

L'ADP-ribosylation est une modification post-traductionnelle de certaines protéines consistant à leur adjoindre une ou plusieurs unités ADP-ribose[1],[2]. Il s'agit d'une modification réversible intervenant dans de nombreux processus des cellules vivantes, notamment la signalisation cellulaire, la réparation de l'ADN, la régulation de l'expression génétique (épigénétique) et l'apoptose[3],[4]. Certaines formes de cancer ont été associées à une ADP-ribosylation incorrecte[5]. Cette modification est également à la base du mécanisme d'action de certaines toxines bactériennes telles que la toxine cholérique et la toxine diphtérique[6].
Mécanisme catalytique[modifier | modifier le code]

La source d'ADP-ribose pour la plupart des enzymes qui assurent l'ADP-ribosylation des protéines est le NAD+, une coenzyme d'oxydoréduction. Au cours de cette réaction de transfert, la liaison N-osidique qui relie le résidu de nicotinamide au reste de la molécule de NAD+ est clivée, permettant la substitution nucléophile avec la chaîne latérale du résidu d'acide aminé cible de la protéine.
Les ADP-ribosyltransférases peuvent réaliser deux types de modifications : des mono(ADP-ribosylations), et des poly(ADP-ribosylations).
Mono(ADP-ribosylation)[modifier | modifier le code]
Les mono-ADP-ribosyltransférases catalysent généralement l'addition d'une unité ADP-ribose à l'extrémité de la chaîne latérale d'un résidu d'arginine à l'aide d'un motif R—S—EXE (Arg–//–Ser–//–Glu–Xaa–Glu) hautement conservé[7], dont les trois premiers résidus conservés forment la triade catalytique. La réaction commence par la formation d'un ion oxonium par rupture de la liaison entre le nicotinamide et le ribose, puis la chaîne latérale du résidu d'arginine cible agit comme un nucléophile pour attirer l'atome de carbone électrophile adjacent à l'ion oxonium. Pour que cela puisse se produire, l'arginine est déprotonée par un résidu de glutamate de l'enzyme, tandis qu'un autre résidu de glutamate conservé forme une liaison hydrogène avec l'un des hydroxyles du ribose pour faciliter la réaction. Le clivage de la liaison aboutit à la libération du nicotinamide, laissant un résidu d'ADP-ribose sur la protéine ADP-ribosylée.
Cette ADP-ribosylation peut notamment être annulée par une ADP-ribosylarginine hydrolase, qui libère de l'ADP-ribose et restitue la protéine dans son état initial ; le NAD lui-même, en revanche, n'est pas reformé.
Poly(ADP-ribosylation)[modifier | modifier le code]
Les poly(ADP-ribose) polymérases (PARP) sont présentes essentiellement chez les eucaryotes et catalysent le transfert de plusieurs unités ADP-ribose à des protéines cibles. La source d'unités ADP-ribose est le NAD, comme pour les mono(ADP-ribosylation)s. Elles utilisent une triade catalytique His-Tyr-Glu pour faciliter la liaison au NAD et le positionnement de l'extrémité de la chaîne d'ADP-ribose naissante sur la protéine cible. Le résidu de glutamate catalyse la formation de la liaison (1→2)-O-osidique entre deux résidus de ribose.
Il existe plusieurs enzymes susceptibles de reconnaître les chaînes de poly(ADP-ribose), les hydrolyser ou les ramifier, et on a par ailleurs identifié plus de 800 protéines porteuses du motif de liaison au poly(ADP-ribose), motif aux contours par ailleurs mal définis : cela signifie que ce motif, aux contours mal définis, peut également intervenir pour recruter d'autres protéines ou pour régulation de la protéine cible[8].
Rôles physiologiques[modifier | modifier le code]
Apoptose[modifier | modifier le code]
Les poly(ADP-ribose) polymérases sont activées lorsque l'ADN est endommagé ou lors d'un stress cellulaire, ce qui a pour effet d'accroître la quantité de protéines poly(ADP-ribosyl)ées et de réduire la quantité de NAD dans la cellule[9]. On a longtemps cru que la poly(ADP-ribose) polymérase 1 (PARP-1) était la seule poly(ADP-ribose) polymérase des cellules de mammifères, de sorte qu'elle a été la plus étudiée. Les caspases sont des protéases à cystéine qui jouent un rôle déterminant dans les processus d'apoptose. Ces enzymes clivent la PARP-1 en deux fragments, ce qui l'inactive complètement et limite la poly(ADP-ribosylation) des protéines. L'un de ces fragments migre du noyau vers le cytoplasme, où l'on pense qu'il déclenche une réponse auto-immunitaire (en).
Lors du processus de parthanatos (en) — distinct de la nécrose et de l'apoptose — l'activation de poly(ADP-ribose) polymérases ou l'inactivation de la poly(ADP-ribose) glycohydrolase conduit à une accumulation de poly(ADP-ribose) cellulaire. On a pu montrer que le poly(ADP-ribose) induit la translocation du facteur d'induction de l'apoptose (en) (AIF) vers le noyau, où il intervient sur la fragmentation de l'ADN. On a également suggéré qu'un défaut d'activation des caspases sous l'effet d'un stress puisse entraîner une nécrose. L'activation excessive des poly(ADP-ribose) polymérases peut déclencher une nécrose régulée par une protéine de la famille du facteur de nécrose tumorale. Les inhibiteurs des PARP ont un effet sur la nécrose, bien qu'on n'ait pas encore compris le mécanisme de cet effet[10].
Régulation de l'expression génétique[modifier | modifier le code]

L'ADP-ribosylation peut affecter l'expression génétique à peu près à tous les niveaux de régulation, y compris l'organisation de la chromatine, le recrutement et la liaison des facteurs de transcription, et les modifications post-transcriptionnelles de l'ARN messager.
Ainsi, l'organisation des nucléosomes est un élément essentiel de la régulation de l'expression des gènes : leur espacement et leur organisation détermine quelles régions de l'ADN sont accessibles à l'outillage enzymatique de transcription de l'ADN en ARN messager. On a montré que la poly(ADP-ribose) polymérase 1 (PARP-1), une poly(ADP-ribose) polymérase, affecte la structure de la chromatine et déclenche des changements dans l'organisation des nucléosomes à travers la modification d'histones. On a également pu montrer que les poly(ADP-ribose) polymérases agissent sur la structure des facteurs de transcription et provoquent le recrutement de plusieurs d'entre eux pour former des complexes avec l'ADN et permettre la transcription. De même, les mono (ADP-ribosyltransférases) agissent également sur les facteurs de transcription se liant aux promoteurs : c'est par exemple le cas de la PARP-14, une mono(ADP-ribosyltransférase) qui agit sur liaison des protéines STAT aux promoteurs[11].
D'autres ADP-ribosyltransférases peuvent modifier les protéines qui se lient à l'ARN messager, ce qui peut bloquer l'expression du transcript de ce gène (silencing)[12].
Réparation de l'ADN[modifier | modifier le code]
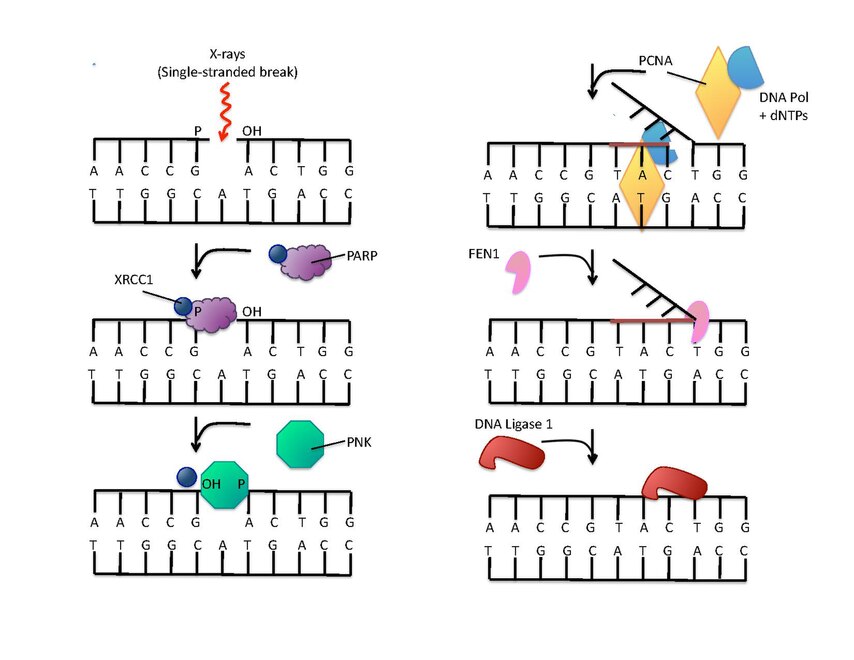
Les poly(ADP-ribose) polymérases peuvent intervenir dans la réparation de l'ADN, qu'il s'agisse de lésions sur un seul brin ou sur les deux brins. Dans le premier cas (réparation par excision de base), les PARP peuvent faciliter l'élimination d'un ose oxydé aussi bien que le clivage du brin. La PARP-1 se lie à une rupture simple brin et rapproche les intermédiaires de la réparation par excision de base, dont les protéines XRCC1 (en) (X-ray Repair Cross Complementing protein 1) et APLF, qui peuvent être recrutées directement ou par le domaine PBZ (poly(ADP-ribose)-binding zinc finger) de la protéine APLF[13]. Ceci conduit à la formation de poly(ADP-ribose). Le domaine PBZ est présent sur de nombreuses protéines intervenant dans la réparation de l'ADN et permet la liaison d'une poly(ADP-ribose) polymérase et par conséquent l'ADP-ribosylation qui a pour effet de recruter les facteurs de réparation interagissant au site de rupture. La PARP-2 est un facteur secondaire dont le rôle est d'assurer une redondance dans le mécanisme de réparation de l'ADN[14].
-
(en) La réparation de l'ADN rompu sur un seul brin est amorcée par la liaison à la PARP-1. La protéine XRCC1 (en) forme un complexe avec une polynucléotide kinase (PNK) qui agit sur les extrémités des brins. La protéine PCNA sert de clamp pour assister l'activité de l'ADN polymérase. La protéine FEN1 (en) élimine ensuite le brin 5’ remplacé, et enfin une ADN ligase assure la suture des brins réparés.
Les poly(ADP-ribose) polymérases ont plusieurs cibles protéiques sur le site où l'ADN est endommagé. On ignore les sites d'ADP-ribosylation des protéines KU (en) et DNA-PKcs (en) (DNA-dependent Protein Kinase, catalytic subunit), qui sont deux composantes de la réparation des lésions double brins d'ADN, tandis qu'on connaît mieux ceux des histones. Ces dernières, qu'il s'agisse des histones de cœur ou des histones de liaison H1, sont toutes ADP-ribosylées à la suite d'un dommage de l'ADN. Le rôle de ces modifications n'est toujours pas connu avec certitude, mais on pense que l'ADP-rybosylation agit sur l'organisation générale de la chromatine afin de faciliter l'accès des facteurs de réparation aux sites endommagés.
Dégradation des protéines[modifier | modifier le code]
Le système ubiquitine-protéasome est l'acteur principal de la dégradation des protéines. Le protéasome 26S est composé d'une sous-unité catalytique 20S et d'une sous-unité régulatrice 19S[15]. Les chaînes de poly-ubiquitine marquent les protéines devant être dégradées pour qu'elles soient reconnues par le protéasome, qui les hydrolyse en petits fragments peptidiques.
Une ADP-ribosyltransférase, en l'occurrence la tankyrase TNKS (en), interagit avec le régulateur PI31 (en) du protéasome. On a pu montrer chez la drosophile et des lignées de cellules humaines que le domaine ankyrine (ANK) de la TNKS facilite l'interaction avec le motif N-terminal de liaison à la TNKS et le domaine C-terminal HbYX de la PI31[16]. Ceci déclenche l'ADP-ribosylation de la PI31 par le domaine PARP de la TNKS. De plus, le traitement de cellules de drosophile avec un inhibiteur de la TNKS réduit l'activité du protéasome 26S, et l'ADP-ribosylation de la PI31 bloque l'inhibition des sous-unités α des particules 20S contrôlée par la PI31. Il est par conséquent possible que l'ADP-ribosylation réalisée par la tankyrase réduise l'activité inhibitrice de la PI31, ce qui a pour effet d'accroître l'activité hydrolytique du protéasome[16].
Importance clinique[modifier | modifier le code]
Cancers[modifier | modifier le code]
Comme vu plus haut, la poly(ADP-ribose) polymérase 1 intervient dans la réparation de l'ADN par excision de base, la réparation des lésions sur un ou deux brins d'ADN et la stabilité de chromosomes. Elle intervient également dans la régulation de la transcription en favorisant les interactions protéine-protéine. Elle utilise le NAD+ pour agir lors de l'apoptose. Lorsqu'une PARP est en suractivité, les taux de NAD+ et d'ATP cellulaires décroissent, ce qui déclenche le processus de nécrose. Ceci est important en carcinogenèse car cela conduit à sélectionner les cellules déficientes (mais pas dépourvues) en PARP-1 en raison de leur meilleur taux de survie lors du développement du cancer[17].
La susceptibilité à la carcinogenèse en cas de déficience en PARP-1 dépend beaucoup du type de dommages à l'ADN. Plusieurs PARP interviennent dans la prévention des cancers : les PARP-1 et PARP-2 interviennent dans la réparation de l'ADN par excision de bases et dans la stabilité des chromosomes, tandis que la PARP-3 intervient dans la régulation du centrosome, et que la tankyrase intervient dans le contrôle de la longueur des télomères[5].
L'inhibition de la PARP-1 a de ce fait été largement étudiée en vue de traitements anticancéreux. Un inhibiteur de la PARP-1 agit en accroissant les dommages infligés par la chimiothérapie sur l'ADN des cellules cancéreuses en bloquant le rôle réparateur de la PARP-1 chez ces dernières.
La PARP-14 est une autre ADP-ribosyltransférase très étudiée dans le cadre de thérapies anticancéreuses. Elle intervient dans la transduction de signal et l'activation de la protéine STAT6, et est associée à l'agressivité des lymphomes B (en)[17].
Toxines bactériennes[modifier | modifier le code]

Les exotoxines bactériennes d'ADP-ribosylation (bARE) catalysent le transfert du groupe ADP-ribose d'une molécule de NAD+ vers les protéines des eucaryotes infectés, libérant le nicotinamide et un proton. Ces exotoxines sont produites par des précurseurs enzymatiques comprenant des domaines A et B : le domaine A est réalise l'ADP-ribosylation tandis que le domaine B assure la translocation de l'enzyme à travers la membrane plasmique[6].
Une fois activées, ces exotoxines bactériennes ADP-ribosylent un nombre plus ou moins grand de protéines des eucaryotes infectés, ce qui déclenche les pathologies associées. Les protéines G sont des cibles reconnues des bARE. Ainsi, la toxine cholérique et l'entérotoxine thermolabile (en) ciblent les sous-unités α des protéines G hétérotrimériques. L'ADP-ribosylation des sous-unités α a pour effet de bloquer ces enzymes dans un état activé, lié au GTP, ce qui produit continuellement de l'AMP cyclique intracellulaire, lequel stimule la libération de fluides et d'électrolytes depuis l'épithélium intestinal. Par ailleurs, la toxine C3 (en) réalise l'ADP-ribosylation des GTPases Rho (en) et Ras, tandis que la toxine de pertussis (en) réalise celle des protéines Gi, Go et Gt, et que la toxine diphtérique réalise celle du facteur d'élongation EF-2 des ribosomes, ce qui réduit la biosynthèse des protéines[6].
Une grande variété de bactéries utilisent de telles exotoxines, par exemple la toxine cholérique chez Vibrio cholerae (choléra), l'entérotoxine thermolabile (en) chez Escherichia coli, l'exotoxine A chez Pseudomonas aeruginosa, la toxine de pertussis (en) chez Bordetella pertussis (coqueluche), la toxine C3 (en) chez Clostridium botulinum (botulisme) et la toxine diphtérique chez Corynebacterium diphtheriae (diphtérie)[19].
Voir aussi[modifier | modifier le code]
Articles connexes[modifier | modifier le code]
Bibliographie[modifier | modifier le code]
- (en) Palmiro Poltronieri, « ADP-Ribosylation Reactions in Animals, Plants, and Bacteria », Challenges, vol. 8, no 1, , p. 14 (ISSN 2078-1547, DOI 10.3390/challe8010014, lire en ligne, consulté le )
Notes et références[modifier | modifier le code]
- (en) Peter Belenky, Katrina L. Bogan et Charles Brenner, « NAD+ metabolism in health and disease », Trends in Biochemical Sciences, vol. 32, no 1, , p. 12-19 (PMID 17161604, DOI 10.1016/j.tibs.2006.11.006, lire en ligne)
- (en) Mathias Ziegler, « New functions of a long-known molecule. Emerging roles of NAD in cellular signaling », The FEBS Journal, vol. 267, no 6, , p. 1550-1564 (PMID 10712584, DOI 10.1046/j.1432-1327.2000.01187.x, lire en ligne)
- (en) « The new life of a centenarian: signalling functions of NAD(P) », Trends in Biochemical Sciences, vol. 29, no 3, , p. 111-118 (PMID 15003268, DOI 10.1016/j.tibs.2004.01.007, lire en ligne)
- (en) Daniela Corda et Maria Di Girolamo, « Functional aspects of protein mono‐ADP‐ribosylation », The EMBO Journal, vol. 22, no 9, , p. 1953-8 (PMID 12727863, DOI 10.1093/emboj/cdg209, lire en ligne)
- (en) Emanuele S. Scarpa, Gaia Fabrizio et Maria Di Girolamo, « A role of intracellular mono-ADP-ribosylation in cancer biology », The FEBS Journal, vol. 280, no 15, , p. 3551-3562 (PMID 23590234, DOI 10.1111/febs.12290, lire en ligne)
- (en) K. M. Krueger et J. T. Barbieri, « The family of bacterial ADP-ribosylating exotoxins », Clinical Microbiology Reviews, vol. 8, no 1, , p. 34-47 (PMID 7704894, PMCID 172848, lire en ligne)
- (en) Sabrina Laing, Mandy Unger, Friedrich Koch-Nolte et Friedrich Haag, « ADP-ribosylation of arginine », Amino Acids, vol. 41, no 2, , p. 257-269 (PMID 20652610, PMCID 3102197, DOI 10.1007/s00726-010-0676-2, lire en ligne)
- (en) Roko Žaja, Andreja Mikoč, Eva Barkauskaite et Ivan Ahel, « Molecular Insights into Poly(ADP-ribose) Recognition and Processing », Biomolecules, vol. 3, no 1, , p. 1-17 (PMID 24970154, PMCID 4030884, DOI 10.3390/biom3010001, lire en ligne)
- (en) A. I. Scovassi, M. Denegri, M. Donzelli, L. Rossi, R. Bernardi, A. Mandarino, I. Frouin et C. Negri, « Poly(ADP-ribose) synthesis in cells undergoing apoptosis: an attempt to face death before PARP degradation », European Journal of Histochemistry, vol. 42, no 4, , p. 251-258 (PMID 10068897)
- (en) Francesca Aredia1, Anna Ivana Scovassi, « Involvement of PARPs in cell death », Frontiers in Bioscience, vol. E6, , p. 308-317 (PMID 24896207, DOI 10.2741/707, lire en ligne)
- (en) Jonathan P. Riley, Aishwarya Kulkarni, Purvi Mehrotra, Byunghee Koh, Narayanan B. Perumal, Mark H. Kaplan et Shreevrat Goenka, « PARP-14 Binds Specific DNA Sequences to Promote Th2 Cell Gene Expression », PLoS One, vol. 8, no 12, , e83127 (PMID 24376650, PMCID 3869773, DOI 10.1371/journal.pone.0083127)
- (en) Keun Woo Ryu, Dae-Seok Kim et W. Lee Kraus, « New Facets in the Regulation of Gene Expression by ADP-Ribosylation and Poly(ADP-ribose) Polymerases », Chemical Reviews, vol. 115, no 6, , p. 2453-2481 (PMID 25575290, PMCID 4378458, DOI 10.1021/cr5004248, lire en ligne)
- (en) « Poly(ADP-ribose) polymerase-2 (PARP-2) is required for efficient base excision DNA repair in association with PARP-1 and XRCC1 », Journal of Biological Chemistry, vol. 277, no 25, , p. 23028-23036 (PMID 11948190, DOI 10.1074/jbc.M202390200, lire en ligne)
- (en) Catherine J. Pears, C. Anne-Marie Couto, Hong-Yu Wang, Christine Borer, Rhian Kiely et Nicholas D. Lakin, « The role of ADP-ribosylation in regulating DNA double-strand break repair », Cell cycle, vol. 11, no 1, , p. 48-56 (PMID 22186780, PMCID 3272231, DOI 10.4161/cc.11.1.18793, lire en ligne)
- (en) Yifan Cheng, « Toward an atomic model of the 26S proteasome », Current Opinion in Structural Biology, vol. 19, no 2, , p. 203-208 (PMID 19286367, PMCID 2743420, DOI 10.1016/j.sbi.2009.02.004, lire en ligne)
- (en) Park F. Cho-Park, Hermann Steller, « Proteasome Regulation by ADP-Ribosylation », Cell, vol. 153, no 3, , p. 614-627 (PMID 23622245, PMCID 3676968, DOI 10.1016/j.cell.2013.03.040, lire en ligne)
- (en) A. Hamid Boulares, Alexander G. Yakovlev et Mark E. Smulson, « Genome Degradation by DNAS1L3 Endonuclease. A Key PARP-1-Regulated Event in Apoptosis », Madame Curie Bioscience Database, , p. 118-131 (DOI 10.1007/0-387-36005-0_11, lire en ligne)
- (en) M. J. Bennett et David Eisenberg, « Refined structure of monomelic diphtheria toxin at 2.3 Å resolution », Protein Science, vol. 3, no 9, , p. 1464-1475 (PMID 7833808, DOI 10.1002/pro.5560030912, lire en ligne)
- (en) Annual Review of Microbiology, « Molecular Mechanisms of the Cytotoxicity of ADP-Ribosylating Toxins », Annual Review of Microbiology, vol. 62, , p. 271-288 (PMID 18785839, DOI 10.1146/annurev.micro.62.081307.162848, lire en ligne)