Régulation de l'expression des gènes
La régulation de l'expression des gènes désigne l'ensemble de mécanismes mis en œuvre pour passer de l'information génétique incluse dans une séquence d'ADN à un produit de gène fonctionnel (ARN ou protéine). Elle a pour effet de moduler, d'augmenter ou de diminuer la quantité des produits de l'expression des gènes (ARN, protéines). Toutes les étapes allant de la séquence d'ADN au produit final peuvent être régulées, que ce soit la transcription, la maturation des ARNm, la traduction des ARNm ou la stabilité des ARNm et protéines. La régulation de l'expression génique est indispensable pour que le métabolisme de la cellule soit en adéquation avec son environnement. Elle est également fondamentale pour la différenciation cellulaire, structurelle et fonctionnelle[1], au cours de l'ontogenèse. Chez les organismes pluricellulaires, elle est responsable du fait que, bien que possédant toutes le même génome, les cellules d'un individu ne l'expriment pas de la même façon.
Chez les procaryotes comme chez les eucaryotes, les gènes sont régulés principalement à l’étape de l’initiation de la transcription, le plus souvent par des protéines capables d’activer ou d’inhiber le fonctionnement du gène. Les activateurs fonctionnent toujours par recrutement de l’ARN polymérase mais aussi, chez les eucaryotes, de complexes protéiques responsables de l’initiation de la transcription.

Chez les procaryotes[modifier | modifier le code]
Régulation transcriptionnelle[modifier | modifier le code]
La transcription est régulée chez les bactéries au niveau de l’initiation de la transcription. Chez les procaryotes (eubactéries et archées), des gènes sont regroupés en opérons, qui sont des unités transcriptionnelles. Un opéron comporte plusieurs gènes dont la transcription génère un seul ARN messager dit polycistronique, aussi appelé polycistron. Sur un ARN polycistronique, les séquences codantes de chaque gène sont séparées par des séquences non codantes de 1 à 40 nucléotides généralement, qui sont l’un des rares types de séquences non codantes connues chez les procaryotes. Il existe des cas de gènes chevauchants : les nucléotides codant les derniers acides aminés du premier gène codent également les premiers acides aminés du second gène. Ces opérons sont généralement impliqués dans la synthèse des acides aminés (comme l’opéron tryptophane (en)) ou dans le métabolisme des nutriments (par exemple l’opéron lactose). L’activité de ces gènes est régulée par des facteurs sigma (en), qui dépendent des conditions extérieures comme la température ou la disponibilité d’une molécule. Par exemple en l’absence d’un substrat, l’expression de gènes codant les enzymes impliquées dans le métabolisme de ce substrat est réprimée, permettant ainsi à la bactérie d’économiser de l’énergie. Mais si ce substrat réapparaît dans le milieu, l’expression des gènes codant ces enzymes est induite. Les gènes d’un même opéron sont régulés ensemble, de façon coordonnée. Des molécules, les effecteurs, peuvent se fixer sur un site allostérique et de cette façon modifier la conformation du domaine de liaison de l’ADN. Un inducteur active la transcription d’un gène ou d’un opéron, un corépresseur l’inhibe.
Opérons inductibles[modifier | modifier le code]
Le cas typique est celui de l’opéron lactose qui contient trois gènes adjacents : lac Z (codant la Bêta galactosidase qui hydrolyse le lactose en glucose + galactose), lac Y (codant la lactose perméase qui transporte le lactose dans la cellule) et lac A (codant la thiogalactoside transacétylase débarrassant la cellule du thiogalactoside importé avec le lactose). Deux protéines régulatrices sont impliquées dans la régulation de l’opéron : le répresseur Lac et l’activateur CAP, ces deux protéines se lient à l’ADN. En présence de lactose, le répresseur est inhibé par le lactose et ne peut se lier à l’opérateur. Le lactose est donc un inducteur et induit la traduction de protéines impliquées dans son métabolisme. En l’absence de glucose, la protéine CAP se lie à l’ADN et active les gènes de l’opéron. Mais en présence de glucose, meilleure source d’énergie que le lactose, l’expression du gène est inhibée même en présence de lactose car CAP ne peut se lier à l’ADN. L’opéron lactose est donc un opéron inductible.

Opérons répressibles[modifier | modifier le code]
L’opéron tryptophane, qui code 5 gènes impliqués dans la biosynthèse du tryptophane, est un opéron répressible. En amont se trouve une séquence codant un répresseur. Le tryptophane, qui est un corépresseur, se fixe au répresseur, ce qui l’active et lui permet de réprimer la transcription des gènes impliqués dans sa biosynthèse. En absence de tryptophane, les gènes sont activés et la cellule synthétise alors son propre tryptophane.
Séquences et protéines régulatrices[modifier | modifier le code]
Il existe chez les procaryotes comme chez les eucaryotes des séquences capables d'activer les promoteurs, les Amplificateur (biologie), ou enhancers. D'autres, les inactivateurs, ou silencers sont capables de les inhiber. Les activateurs sont des protéines capables de se lier à l’ADN recrutant au niveau de l’ARN polymérase, l’enzyme indispensable de la transcription. Les répresseurs sont des protéines qui inhibent l’expression des gènes en se fixant au niveau du promoteur : ils empêchent donc l’ARN polymérase de s’y fixer. Les procaryotes disposent, comme les eucaryotes, de séquences cis-régulatrices. Les promoteurs comprennent des séquences conservées : au point 0 d'initiation de la transcription se trouve le triplet CAT (A étant le point 0). Vers -10, se trouve la séquence TATAAT (semblable à la TATA box des eucaryotes) et vers -35 la séquence TTGACA.
Régulation traductionnelle[modifier | modifier le code]
Certaines protéines régulent leur propre traduction en se fixant sur la séquence d’initiation de la traduction de l’ARN messager. Le ribosome ne peut alors se lier à l’ARN messager, rendant impossible la traduction. Une autre possibilité repose sur l’utilisation d’ARN antisens, s’appariant à l’ARN messager et formant un ARN double brin empêchant la traduction. Enfin, un dernier cas utilise la faculté de formation de structures secondaires par l’ARN messager.
Chez les eucaryotes[modifier | modifier le code]
Régulation transcriptionnelle[modifier | modifier le code]
La transcription peut être régulée par des signaux extracellulaires, des séquences cis-régulatrices et des facteurs trans. Comme chez les bactéries, c’est généralement l’initiation de la transcription qui est régulée.
Régulation par l’intermédiaire de récepteurs cellulaires[modifier | modifier le code]
Les cellules perçoivent les signaux extracellulaires par l'intermédiaire de systèmes récepteurs-effecteurs, qui transmettent l'information au noyau. Les récepteurs peuvent être membranaires ou nucléaires. Les récepteurs membranaires reconnaissent des facteurs hydrosolubles. Les récepteurs nucléaires sont des facteurs de transcription inductibles par des facteurs liposolubles, comme les hormones stéroïdiennes, l'hormone thyroïdienne, l'acide rétinoïque ou encore le calcitriol. Ces récepteurs reconnaissent dans le promoteur des gènes qu’ils régulent des éléments de réponse aux hormones, les HRE (hormone réponse élément). Dans le cas des hormones liposolubles, le site de reconnaissance du récepteur pour l'élément de réponse peut être masqué par une protéine chaperonne, qui se dissocie lors de la fixation de l'hormone. La grande variabilité de réponses entre les cellules d’un même tissu et dans un même environnement a conduit à l'hypothèse d'un mécanisme probabiliste de l'expression des gènes, plutôt que déterministe.
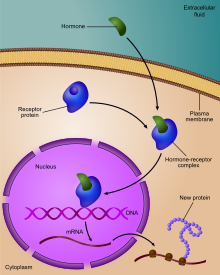
Séquences cis-régulatrices[modifier | modifier le code]
La régulation cis désigne la régulation de l’activité d’un gène par une séquence ADN. Plusieurs types de séquences régulent l’activité des gènes.
Les promoteurs, situés en amont du gène, sont des séquences permettant la fixation de l'ARN polymérase II, responsable de la transcription du gène, sur l`ADN. Un promoteur comporte des séquences conservées. Au point d'initiation de la transcription se trouve généralement une adénine encadrée par deux pyrimidines. En -25 chez les eucaryotes se trouve la TATA box et en -75 se trouve la CAAT box, qui augmente l'efficacité du promoteur. Enfin en -90, on retrouve la CG box, une séquence GGGCGG régulant également la transcription. Suivant les promoteurs, le nombre et la position de ces séquences varient. Il arrive que des promoteurs soient dépourvus de certaines de ces séquences. Mais tous ces facteurs influent sur la performance du promoteur. Des séquences spécifiques, appelées éléments de réponse (response elements ou RE en Anglais), reconnues par un même facteur de transcription inductible, peuvent être présentes dans les promoteurs de plusieurs gènes. Ces éléments permettent une régulation coordonnée d’un grand nombre de gènes impliqués dans un processus physiologique particulier. C’est le cas de gènes activés en réponse à des facteurs externes, tels que la lumière, des hormones comme les glucocorticoïdes, le choc thermique, l’hypoxie ou encore le sérum. Les gènes ribosomiques, les histones, les globulines, ou encore les gènes homéotiques sont souvent régulés de manière coordonnée. Commne chez les procaryotes, des amplificateurs et des inactivateurs sont impliqués dans la régulation de l’activité des promoteurs. Ils peuvent être situées loin en amont ou en aval du promoteur. Un amplificateur active les promoteurs de certains gènes en utilisant des facteurs de transcription. Un inactivateur inhibe la transcription. Ces séquences régulatrices ont souvent une action tissu-spécifique. Des facteurs de transcription viennent se fixer sur ces séquences, et peuvent interagir de manière directe ou indirecte avec les facteurs de transcription du complexe transcriptionnel. Ces contacts peuvent être directs, sous la forme de boucle d'ADN mettant en interaction ces facteurs de transcription, ou indirects, par l’intermédiaire de molécules des co-activatrices.
Facteurs Trans[modifier | modifier le code]
La régulation trans désigne la régulation de l’activité des gènes par des protéines appelées facteurs trans. Les facteurs trans viennent se fixer aux séquences cis et peuvent être des activateurs, capables de recruter l’ARN polymérase et le complexe protéique nécessaires à la transcription, ou bien des enzymes capables de modifier les nucléosomes, ou encore des répresseurs empêchant la fixation, entre autres, de l’ARN polymérase. Les facteurs de transcription sont des protéines possédant le plus souvent une structure modulaire constitué d'un certain nombre de sous-unités et présentant des domaines avec des fonctions spécifiques. Ils sont capables de se fixer à l'ADN d'une part, et d'activer la transcription d'autre part. Plusieurs types de domaines protéiques permettant l’interaction avec l’ADN sont décrits, tels que les motifs hélice-coude-hélice, doigt de zinc, avec un atome de zinc interagissant avec les résidus cystéine et histidine, hélice-boucle-hélice, ou encore leucine zipper. Les facteurs de transcription peuvent être restreints à type de tissu, de cellule ou à un stade particulier du développement embryonnaire. Ils peuvent être régulés par la fixation d'un ligand, comme des hormones stéroïdiennes, par la formation d'un dimère activant ou inactivant le site de fixation à l'ADN, ou encore par des modifications sur des acides aminés spécifiques, comme des phosphorylations. Ainsi, les protéines à homéodomaine sont des facteurs de transcription spécifiques à des tissus particuliers. Elles comportent un homéodomaine (ou homéoboîte) leur permettant de se lier à l’ADN, ainsi qu’un domaine d’activation de la transcription capable de recruter l’ARN polymérase et les complexes protéiques. Ces protéines étant généralement restreintes à un type de tissu particulier, elles n’activent donc l’expression de gènes que dans ces tissus. Par exemple, les gènes Hox jouent un rôle crucial dans le développement embryonnaire des animaux, en particulier dans la détermination de l’identité des cellules.
Régulation post-transcriptionnelle[modifier | modifier le code]
Régulation des modifications post-transcriptionnelles[modifier | modifier le code]
L'expression des gènes peut être régulée par la modulation des modifications post-transcriptionnelles, c'est-à-dire la maturation des pré-ARNm, soit l’ajout d'une coiffe GTP et d’un ou plusieurs plus groupements méthyles en 5', et d'une queue poly A (polyadénylation) en 3', sans quoi les ARNm peuvent être détruits par les RNases. Cette modulation peut donc influer sur la stabilité des ARNm.
Régulation par les petits ARNs[modifier | modifier le code]
L’expression des gènes peut aussi être régulée au niveau post-transcriptionnel par des petits ARNs, comme chez les procaryotes. Les ARN interférents (ARNi) conduisent à la destruction de l’ARN messager. Les micro-ARN (miARN), sont également responsables de l’extinction de gène en s’appariant aux ARN messagers, ce qui conduit à l’inhibition de leur traduction et à leur destruction. Ils sont notamment actifs lors de phases de développement et de prolifération cellulaire. La propriété de ces ARN est notamment utilisée en recherche in vitro afin d’inactiver l’expression d’un gène étudié.
Epissage alternatif[modifier | modifier le code]
L'épissage alternatif est également un mode de régulation post-transcriptionnelle[2]. Il permet, à partir d'un seul gène donnant un seul pré-ARNm d'obtenir plusieurs ARN messagers différents en combinant les exons de manière différenciée. Un ou des exons peuvent être éliminés, et des introns peuvent être conservés, devenant alors des exons. L'épissage alternatif permet d'augmenter le nombre de protéines produites par un organisme sans augmenter le nombre de gènes, comme dans le cas des chaînes lourdes des immunoglobulines. Ce mécanisme permet également de produire des protéines différenciées en fonction du stade de développement ou des spécificités tissulaires. Ainsi, la détermination du sexe chez la drosophile faites intervenir une cascade de régulation impliquant plusieurs facteurs d'épissage. Chez les mammifères, 30 à 50 % des gènes seraient soumis à un épissage alternatif[3]. L`épissage alternatif explique par exemple que le nombre de protéines humaines (environ 90 000), soit plus élevé que le nombre de gènes présent dans le génome humain (environ 25 000).

Régulation traductionnelle[modifier | modifier le code]
Deux voies de régulation de la traduction sont connues : la voie TOR et la voie de réponse au stress. La voie TOR (appelée mTOR pour les mammifères) repose sur la protéine TOR (pour Target of Rapamycine), qui contrôle la croissance cellulaire et le métabolisme. Ces deux voies adaptent l'activité des gènes à l'environnement et aux signaux extracellulaires (par exemple des hormones ou des nutriments). Elles reposent sur des réactions de phosphorylation de protéines (des activateurs ou des inhibiteurs de gènes). Ainsi la disponibilité en nutriments et acides aminés stimulent la voie TOR, qui active des activateurs de transcription, responsables de l'augmentation du métabolisme énergétique et permettant l'expression de gènes impliqués dans la croissance cellulaire et la division. Ce mécanisme est notamment responsable du phénotype particulier de la reine des abeilles par rapport aux ouvrières, et notamment sa grande taille, au sont dus à la gelée royale avec laquelle elle a été nourrie. La gelée royale a la particularité de stimuler la voie TOR. L'inhibition expérimentale de la voie TOR (par la rapamycine ou bien par ARN interférence) induit le développement d'un phénotype d'ouvrière[4]. La voie de réponse au stress exerce l'effet inverse : en présence de rayonnements (UV notamment), de carences alimentaires ou de polluants, d'autres activateurs de transcription sont stimulés et activent des gènes impliqués dans la défense de la cellule. Ces gènes permettent de détruire des protéines anormales produites à cause du stress, de stimuler les systèmes antioxydants, d'éliminer les constituants endommagés par le stress, et de remplacer les protéines anormales. Si le stress se prolonge, la cellule subit une apoptose[5].
Modification des nucléosomes[modifier | modifier le code]
Chez les eucaryotes, la chromatine peut se présenter sous forme d’hétérochromatine (ADN condensé autour d’un octamère d’histones : sous la forme de nucléosomes) et d’euchromatine (ADN décondensé). L’hétérochromatine rend impossible la transcription et seules les séquences de l’euchromatine sont exprimées. Typiquement, les séquences télomériques et centromériques sont sous forme d’hétérochromatine, ces régions contenant peu ou pas de gènes. Des enzymes recrutées par des activateurs sont capables de modifier des histones en ajoutant ou en retirant des groupes chimiques. Ainsi, les histones acétylases ajoutent des groupements acétyles, ce qui active les gènes. A l`inverse, des répresseurs recrutent des modificateurs d’histones, comme les histones désacétylases capables de désacétyler les histones. Elles modulent ainsi l’accessibilité du gène par les enzymes de transcription. Ce mécanisme consomme de l’ATP. Les désacétylations peuvent permettre de maintenir des portions d’ADN à l’état silencieux, et cette condition peut s’étendre à de plus larges segments car les enzymes de modification sont préférentiellement recrutées sur des portions déjà maintenues dans cet état.
D'autres enzymes influent sur l'activité des gènes en méthylant ou phosphorylant les histones, ce qui inactive la chromatine. La Poly ADP ribosylation peut également réguler la structure de la chromatine [6]. La modification d’histones peut être héréditaire. Il existe également un lien avec le métabolisme énergétique. Les réactions responsables de l'état des histones, l'acétylation, la méthylation ou la phosphorylatyion, reposent sur des molécules du métabolisme énergétique[7]. Ainsi, les groupements acétyls sont issus de l'acétyl CoA, le donneur de méthyl est le S-Adénosylméthionine et la polyADP ribosylation repose sur le NAD+, autant de molécules impliquées dans le métabolisme énergétique. L'activité de ce dernier, qui dépend de la disponibilité des nutriments ou de la quantité d'oxygène disponible, influe sur la concentration en ces diverses molécules interagissant avec les histones. Une modification du métabolisme énergétique influe donc sur l'état de la chromatine et donc sur l'expression des gènes. Par exemple, un régime métabolique produisant de l'acétyl CoA en grande quantité permet à une cellule de réagir plus vite aux changements d'environnement en exprimant de nouveaux gènes. Ces modifications étant transmissibles, le phénotype d'un individu dépend en partie de l'environnement de ses ancêtres. Ainsi, des personnes ayant connu la famine durant leur jeunesse ont une descendance dotée d'une espérance de vie supérieure[8].
Méthylation de l’ADN[modifier | modifier le code]
Il est également possible d’inhiber des gènes en faisant intervenir une enzyme, l’ADN méthylase, qui a pour fonction de méthyler l’ADN, généralement au niveau des séquences CpG[1] (Cytosine-phosphate-Guanine). Ces îlots CpG (aussi appelés îlots CG) sont généralement associés aux promoteurs et donc à l'activité des gènes[1]. Le mécanisme est documenté chez les mammifères. À la suite de la réplication, les gènes codant ces méthyltransférases sont activés et les enzymes reconnaissent le brin hémiméthylé et méthylent le nouveau brin. C’est également un phénomène d’extinction génique. En effet la méthylation empêche la liaison des complexes enzymatiques de la transcription ainsi que des activateurs. De plus, dans certains cas elle peut permettre la fixation de répresseurs modifiant les histones. La méthylation concerne généralement les séquences promotrices[9], et les cellules cancéreuses présentent souvent des méthylations aberrantes[10]. Il est à noter que chez les mammifères femelles, seul l’un des deux chromosomes X est actif. L’autre est désactivé par méthylation. Certains gènes ne s’expriment que dans certains tissus car ils sont méthylés dans les autres. La méthylation d’ADN est héréditaire (épigénétique). Par exemple, chez Linaria vulgaris, les fleurs sont généralement zygomorphes, mais il existe des formes asymétriques, bien que le gène CYCLOIDEA, responsable de la symétrie florale, soit intact. Mais son expression est inhibée au niveau des doublets CG, modification épigénétique transmissible selon les lois de Mendel[11]. La méthylation des îlots CG est également documentée chez les insectes sociaux[12], comme les fourmis, les termites ou les abeilles. Ces insectes vivent en communautés où un individu connaîtra au cours de sa vie des fonctions bien distinctes, les castes. Toutes les castes d'insectes (ouvrières, soldats...) ont un génome identique mais les importantes différences morphologiques, comportementales et physiologiques seraient liées à des différences au niveau de la méthylation de certains gènes[13]. Il semblerait que l'origine des épimutations serait liée à un réarrangement chromosomique qui insérerait un site méthylé à proximité d'un gène, ce qui recruterait les méthyltransférases, permettant ainsi le maintien de la méthylation à chaque division[14], mais il existe également des cas de méthylation de novo, créant des sites méthylés maintenus ensuite par les méthyltransférases. Enfin, une inhibition de la méthyltransférase peut être à l'origine de l'apparition d'épiallèles. Il semblerait que l'activité des méthyltransférases soit liée à la nutrition, notamment la disponibilité en acides aminés soufrés, et que celle-ci puisse modifier l'épigénome d'une génération à l'autre. L'activation ou l'inactivation de gènes par la méthylation est souvent liée à l'environnement. Ainsi, chez certaines plantes, la vernalization est due à l'activation du gène FLOWERING LOCUS C par les basses températures, l'activité de ce gène étant modulée par méthylation/déméthylation[15]. Il semblerait également que chez les mammifères, la senescence s'accompagne d'une hypométhylation de certaines séquences répétées et transposons, ainsi que d'une hyperméthylation d'autres gènes. On sait également que chez les souris, la couleur des descendants dépend de l'alimentation de la mère gravide : une souris nourrie à des aliments fournisseurs de méthyle aura proportionnellement plus de descendants dont le pelage sera “wild-type”[16]. C'est ainsi que l'on sait qu'un phénotype adulte peut avoir pour origine un stimulus environnemental survenu au plus jeune âge.

Références[modifier | modifier le code]
- Jaenish R., Bird A., 2003. "Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals", Nature genetics supplement, 33(245:254)
- Kelemen O, Convertini P, Zhang Z, et al, 2013. “Function of alternative splicing”, Gene 514:1–30
- Gavin C Roberts, Christopher W.J Smith, 2002. “Alternative splicing: combinatorial output from the genome”, Current Opinion in Chemical Biology 6(3):375-383
- Patel A, Fondrk MK, Kaftanoglu O, Emore C, Hunt G, et al., 2007. "The Making of a Queen: TOR Pathway Is a Key Player in Diphenic Caste Development". PLoS ONE 2(6): e509
- Olivier Jean-Jean, "La cellule à l'écoute de son environnement", Découverte, revue du Palais de la découverte, janvier-février 2014, p. 44-51.
- Beneke S, 2012. “Regulation of chromatin structure by poly(ADP-ribosyl)ation”, Fron. Genet. 3;3:169
- Wellen K. E., Hatzivassiliou G., Sachdeva U. M., Bui T. V., Cross J. R., Thompson C. B., 2009. “ATP-Citrate Lyase Links Cellular Metabolism to Histone Acetylation”, Science 5930:1076-1080
- Andràs Pàldi, "épigénétique et métabolisme", Dossier Pour la Science, octobre-décembre 2013, p. 104-109
- Bell JT, Pai AA, Pickrell JK, Gaffney DJ, Pique-Regi R, Degner JF, Gilad Y, Pritchard JK (2011). “DNA methylation patterns associate with genetic and gene expression variation in HapMap cell lines”. Genome Biology 12 (1): R10
- Vertino PM, Spillare EA, Harris CC, Baylin SB (April 1993). “Altered chromosomal methylation patterns accompany oncogene-induced transformation of human bronchial epithelial cells”. Cancer Res. 53 (7): 1684–9
- Cubal P., Vincent C., Coen E., 1999. “An epigenetic mutation responsible for natural variation in floral symmetry”, Nature, 401, 157-161
- Kronforst M. R., Giley D.C. Strassman J. E., Queller D., 2008. “DNA methylation is widespread across social Hymenoptera”, Current Biology 19: R287— R288.
- Weiner S.A., Galbraith D. A., Adams D. C., Valenzuela N., Noll F. B., Grozinger C. M., Toth A. L.. 2013 "A survey of DNA methylation across social insect species, life stages, and castes reveals abundant and caste-associated methylation in a primitively social wasp", Naturwissenschaften 100:795:799.
- Frédéric Berger, "La mémoire d'une cellule", Dossier Pour la Science, octobre-décembre 2013, p. 93-97
- Sheldon, C.C. et al., 1999. "The FLF MADS box gene: a repressor of flowering in Arabidopsis regulated by vernalization and methylation", Plant Cell 11, 445-458
- Wolff, G.L., Kodell, R.L., Moore, S.R. & Cooney, C.A., 1998. "Maternal epigenetics and methyl supplements affect agouti gene expression in Avy/a mice", Faseb J. 12, 949-957
Sources[modifier | modifier le code]
- Watson J., Baker T., Bell S., Gann A., Levine M., Losick R., Biologie moléculaire du gène, 6e édition, Pearson Education France, 2008, traduction française par Jacquet M. et Le Gaillard F., 2009, 688 p.
- Harry M., Génétique moléculaire et évolutive, deuxième édition, Maloine, 2008, 465 p.
- Alberts, Bray, Hopkin, Johnson, Lewis, Raff, Roberts, Walter, L’Essentiel de la biologie cellulaire, deuxième édition, Médecine-Sciences Flammarion, 2004, 739 p.


