Banque génomique
Une banque génomique est une collection de l'ADN génomique total d'un seul organisme. L'ADN est stocké dans une population de vecteurs identiques, chacun contenant un insert d'ADN différent. Afin de construire une banque génomique, l'ADN de l'organisme est extrait des cellules puis digéré par une enzyme de restriction pour couper l'ADN en fragments d'une taille spécifique. Les fragments sont ensuite insérés dans le vecteur à l'aide de la ligase[1]. Ensuite, le vecteur d'ADN peut être absorbé par un organisme hôte - généralement une population d'Escherichia coli ou de levure -, chaque cellule ne contenant qu'une seule molécule de vecteur. L'utilisation d'une cellule hôte pour transporter le vecteur permet de faciliter l'amplification et la récupération de clones spécifiques de la banque pour analyse[2]. Il existe plusieurs types de vecteurs avec différentes capacités d'insert. Généralement, les banques faites à partir d'organismes avec des génomes plus grands nécessitent des vecteurs comportant des inserts plus grands, donc moins de molécules vectorielles sont nécessaires pour constituer la banque. Les chercheurs peuvent choisir un vecteur en considérant également la taille idéale de l'insert afin de trouver le nombre souhaité de clones nécessaires pour avoir une couverture complète du génome[3]. Les banques génomiques sont couramment utilisées pour le séquençage de régions génomiques. Elles ont joué un rôle important dans le séquençage du génome entier de plusieurs organismes, y compris du génome humain et de plusieurs organismes modèles[4],[5].
Histoire[modifier | modifier le code]
Le premier génome basé sur l'ADN entièrement séquencé a été réalisé par le double lauréat du prix Nobel, Frederick Sanger en 1977. Sanger et son équipe de scientifiques ont créé une banque du bactériophage phi X 174 pour une utilisation dans le séquençage de l'ADN[6]. L'importance de ce succès a contribué à la demande toujours croissante de séquençage des génomes pour la recherche sur la thérapie génique. Les équipes sont désormais en mesure de cataloguer les polymorphismes dans les génomes et d'étudier les gènes candidats contribuant à des maladies telles que la maladie de Parkinson, la maladie d'Alzheimer, la sclérose en plaques, la polyarthrite rhumatoïde et le diabète de type 1[7]. Celles-ci sont dues à l'avancée des études d'association à l'échelle du génome à partir de la capacité à créer et à séquencer des banques génomiques. Des études préalables, de liaison et de gènes candidats constituaient quelques-unes des seules approches[8].
Construction d'une banque génomique[modifier | modifier le code]
La construction d'une banque génomique implique la création de nombreuses molécules d'ADN recombinant. L'ADN génomique d'un organisme est extrait puis digéré par une enzyme de restriction. Pour les organismes à très petits génomes (environ 10 kb), les fragments digérés peuvent être séparés par électrophorèse sur gel. Les fragments séparés peuvent ensuite être excisés et clonés séparément dans le vecteur. Cependant, lorsqu'un génome de grande taille est digéré par une enzyme de restriction, il y a beaucoup trop de fragments pour les exciser individuellement. L'ensemble complet des fragments doit alors être cloné avec le vecteur, et à ce moment-là on peut séparer les clones. Dans les deux cas, les fragments sont ligaturés dans un vecteur qui a été digéré avec la même enzyme de restriction. Le vecteur contenant les fragments d'ADN génomique insérés peut ensuite être introduit dans un organisme hôte[1]. Voici les étapes de création d'une banque génomique à partir d'un grand génome :
- Extraction et purification de l'ADN ;
- Digestion de l'ADN avec une enzyme de restriction ; cela crée des fragments de taille statistiquement homogène ;
- Insertion des fragments d'ADN dans des vecteurs qui ont été coupés avec la même enzyme de restriction ; on utilise une enzyme, l'ADN ligase, pour lier les extrémités des fragments d'ADN à celles du vecteur, et former des molécules recombinantes circulaires ;
- Absorption de ces molécules recombinantes par une bactérie-hôte par transformation, créant la banque d'ADN proprement dite[9],[10].
Voici un diagramme des étapes décrites ci-dessus.

Détermination du titre de la banque[modifier | modifier le code]
Après qu'une banque génomique ait été construite avec un vecteur viral, tel qu'un phage lambda, le titre de la banque peut être déterminé. Le calcul du titre permet aux chercheurs d'estimer le nombre de particules virales infectieuses qui ont été créées avec succès dans la banque. Pour ce faire, des dilutions de la banque sont utilisées pour transformer des cultures d'E. coli de concentrations connues. Les cultures sont ensuite étalées sur des plaques d'agar et incubées pendant une nuit. Le nombre de plaques virales est compté et peut être utilisé pour calculer le nombre total de particules virales infectieuses dans la banque. La plupart des vecteurs viraux portent également un marqueur qui permet de distinguer les clones contenant un insert de ceux qui n'ont pas d'insert. Cela permet aux chercheurs de déterminer également le pourcentage de particules virales infectieuses portant réellement un fragment de la banque[11]. Une méthode similaire peut être utilisée pour titrer les banques génomiques faites avec des vecteurs non viraux, tels que les plasmides et les BAC. Un test de ligature de la banque peut être utilisé pour transformer E. coli. La transformation est ensuite étalée sur des plaques de gélose et incubée pendant une nuit. Le titre de la transformation est déterminé en comptant le nombre de colonies présentes sur les plaques. Ces vecteurs ont généralement un marqueur sélectionnable permettant la différenciation des clones contenant un insert de ceux qui n'en contiennent pas. En faisant ce test, les chercheurs peuvent également déterminer l'efficacité de la ligature et effectuer les ajustements nécessaires pour s'assurer qu'ils obtiennent le nombre souhaité de clones pour la banque[12].
Criblage d'une banque[modifier | modifier le code]

Afin d'isoler des clones qui contiennent des régions d'intérêt d'une banque, celle-ci doit d'abord être filtrée. Une méthode de criblage est l'hybridation, dans laquelle chaque cellule hôte transformée d'une banque ne contiendra qu'un seul vecteur avec un insert d'ADN. Toute la banque peut être plaquée sur un filtre sur support. Le filtre et les colonies sont préparés pour l'hybridation puis marqués avec une sonde[13]. L'ADN-cible (l'insert d'intérêt) peut être identifié par détection, par exemple en autoradiographie, en raison de l'hybridation avec la sonde comme indiqué ci-dessous. Une autre méthode de criblage est la réaction en chaîne par polymérase (PCR). Certaines banques sont stockées sous forme de « pools » de clones et le criblage par PCR est un moyen efficace d'identifier les pools contenant des clones spécifiques[2].
Types de vecteurs[modifier | modifier le code]
La taille du génome varie selon les différents organismes et le vecteur de clonage doit être sélectionné en conséquence. Pour un grand génome, un vecteur de grande capacité doit être choisi de manière qu'un nombre relativement petit de clones soit suffisant pour couvrir l'ensemble du génome. Cependant, il est souvent plus difficile de caractériser un insert contenu dans un vecteur de plus grande capacité que dans des plus petits[3].
Ci-dessous un tableau de plusieurs types de vecteurs couramment utilisés pour les banques génomiques et la taille de l'insert que chacun contient généralement :
| Type de vecteur | Taille de l'insert (milliers de bases) |
|---|---|
| Plasmide | jusqu'à 10 |
| Phage lambda (λ) | jusqu'à 25 |
| Cosmide | jusqu'à 45 |
| Bactériophage P1 | 70 à 100 |
| Chromosome artificiel P1 (PAC) | 130 à 150 |
| Chromosome artificiel bactériens (BAC) | 120 à 300 |
| Chromosome artificiel de levure (YAC) | 250 à 2 000 |
Plasmides[modifier | modifier le code]
Un plasmide est une molécule d'ADN circulaire double brin couramment utilisée pour le clonage moléculaire. Les plasmides ont généralement une longueur de 2 à 4 kb et sont capables de porter des inserts jusqu'à 15 kb. Les plasmides contiennent une origine de réplication leur permettant de se répliquer à l'intérieur d'une bactérie indépendamment du chromosome hôte. Les plasmides portent généralement un gène de résistance aux antibiotiques qui permet la sélection de cellules bactériennes contenant le plasmide. De nombreux plasmides portent également un gène rapporteur qui permet aux chercheurs de distinguer les clones contenant un insert de ceux qui n'en contiennent pas[3].
Phage lambda (λ)[modifier | modifier le code]
Le phage λ est un virus à ADN double brin qui infecte E. coli. Le chromosome λ mesure 48,5 kb de long et peut porter des inserts jusqu'à 25 kb. Ces inserts remplacent les séquences virales non essentielles du chromosome λ, tandis que les gènes nécessaires à la formation de particules virales et à l'infection restent intacts. L'ADN inséré est répliqué avec l'ADN viral ; ainsi, ensemble, ils sont encapsidés en particules virales. Ces particules sont très efficaces pour l'infection et la multiplication, conduisant ainsi à une production plus élevée de chromosomes λ recombinants[3]. Cependant, en raison de la taille plus petite de l'insert, les banques faites avec le phage λ peuvent nécessiter de nombreux clones pour une couverture complète du génome[14].
Cosmides[modifier | modifier le code]
Les cosmides sont des plasmides qui contiennent une petite région d'ADN du bactériophage λ appelée séquence cos. Cette séquence permet au cosmide d'être conditionné en particules de bactériophage λ. Ces particules, contenant un cosmide linéarisé, sont introduites dans la cellule hôte par transduction. Une fois à l'intérieur de l'hôte, les cosmides circulent à l'aide de l'ADN ligase de l'hôte, puis fonctionnent comme des plasmides. Les cosmides sont capables de transporter des inserts jusqu'à 40 kb[2].
Vecteurs de bactériophage P1[modifier | modifier le code]
Les vecteurs du bactériophage P1 peuvent contenir des inserts de 70 à 100 kb. Ce sont au départ des molécules d'ADN linéaires encapsidées dans des particules de bactériophage P1. Ces particules sont injectées dans une souche d'E. coli exprimant la recombinase CRE. Le vecteur P1 linéaire devient alors circularisé par recombinaison entre deux sites loxP dans le vecteur. Les vecteurs P1 contiennent généralement un gène de résistance aux antibiotiques et un marqueur de sélection positive pour distinguer les clones contenant un insert de ceux qui n'en contiennent pas. Les vecteurs P1 contiennent également un réplicon de plasmide P1, qui garantit qu'une seule copie du vecteur est présente dans une cellule. Cependant, il existe un deuxième réplicon P1 - appelé réplicon lytique P1 - qui est contrôlé par un promoteur inductible. Ce promoteur permet l'amplification de plus d'une copie du vecteur par cellule avant l'extraction de l'ADN[2].
Chromosomes artificiels P1[modifier | modifier le code]
Les chromosomes artificiels P1 (PAC) possèdent des caractéristiques à la fois des vecteurs P1 et des chromosomes artificiels bactériens (BAC). Semblables aux vecteurs P1, ils contiennent un plasmide et un réplicon lytique comme décrit ci-dessus. Mais contrairement aux vecteurs P1, ils n'ont pas besoin d'être encapsidés dans des particules de bactériophage pour la transduction. Au lieu de cela, ils sont introduits dans E. coli sous forme de molécules d'ADN circulaires par électroporation, tout comme les BAC[2]. Également similaires aux BAC, ils sont relativement plus difficiles à préparer en raison du fait qu'ils n'ont qu'une seule origine de réplication[15].
Chromosomes artificiels bactériens[modifier | modifier le code]
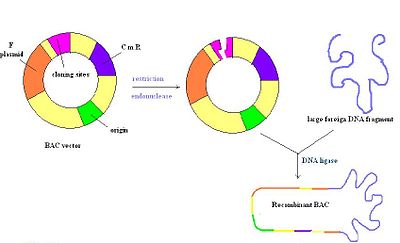
Les chromosomes artificiels bactériens (BAC) sont des molécules d'ADN circulaires, généralement d'environ 7 kb de longueur, qui sont capables de contenir des inserts jusqu'à 300 kb. Les vecteurs BAC contiennent un réplicon dérivé du facteur F dE. coli, ce qui garantit qu'ils sont maintenus à une copie par cellule[4]. Une fois qu'un insert est ligaturé dans un BAC, celui-ci est introduit dans des souches déficientes en recombinaison dE. coli par électroporation. La plupart des vecteurs BAC contiennent un gène de résistance aux antibiotiques et également un marqueur de sélection positive[2]. La figure de droite représente un vecteur BAC coupé avec une enzyme de restriction, suivi de l'insertion d'un ADN étranger qui est réannexé par une ligase. Dans l'ensemble, il s'agit d'un vecteur très stable, mais certains peuvent être difficiles à préparer en raison de la présence d'une seule origine de réplication, tout comme les PAC[15].
Chromosomes artificiels de levure[modifier | modifier le code]
Les chromosomes artificiels de levure (YAC) sont des molécules d'ADN linéaires contenant les caractéristiques nécessaires à un chromosome de levure authentique, y compris des télomères, un centromère et une origine de réplication. De grands inserts d'ADN peuvent être ligaturés au milieu du YAC de sorte qu'il y ait un « bras » du YAC de chaque côté de l'insert. Le YAC recombinant est introduit dans la levure par transformation ; les marqueurs sélectionnables présents dans le YAC permettent d'identifier les transformants réussis. Les YAC peuvent contenir des insertions jusqu'à 2000 kb, mais la plupart des banques YAC contiennent des inserts de 250 à 400 kb. Théoriquement, il n'y a pas de limite supérieure sur la taille de l'insert qu'un YAC peut contenir. C'est la qualité de la préparation de l'ADN utilisé pour les inserts qui détermine la taille limite[2]. L'aspect le plus problématique à l'utilisation de YAC est le fait qu'ils sont potentiellement sujets à des réarrangements[15].
Comment sélectionner un vecteur[modifier | modifier le code]
La sélection des vecteurs est nécessaire pour s'assurer que la banque créée est représentative de l'ensemble du génome. Tout insert du génome dérivé d'une enzyme de restriction devrait avoir une chance égale d'être dans la banque par rapport à tout autre insert. En outre, les molécules recombinantes doivent contenir des inserts suffisamment grands pour garantir que la taille de la banque peut être manipulée de manière pratique[15]. Ceci est particulièrement déterminé par le nombre de clones nécessaires à avoir dans une banque. Le nombre de clones pour obtenir un échantillonnage de tous les gènes est déterminé par la taille du génome de l'organisme ainsi que la taille moyenne de l'insert. Ceci est représenté par la formule suivante (également connue sous le nom de formule Carbon et Clarke[16] :
où
est le nombre nécessaire de recombinants[17]
est la probabilité souhaitée qu'un fragment du génome se produise au moins une fois dans la banque créée
est la proportion fractionnaire du génome dans un seul recombinant
peut en outre être démontré comme étant :
où
est la taille de l'insert
est la taille du génome
Ainsi, l'augmentation de la taille de l'insert (par le choix du vecteur) permettrait de nécessiter moins de clones nécessaires pour représenter un génome. La proportion de la taille de l'insert par rapport à la taille du génome représente la proportion du génome respectif dans un seul clone[15]. Voici l'équation avec toutes les parties considérées :
Exemple de sélection de vecteur[modifier | modifier le code]
La formule ci-dessus peut être utilisée pour déterminer le niveau de confiance à 99% certifiant que toutes les séquences d'un génome sont représentées en utilisant un vecteur avec une taille d'insert de 20 000 paires de bases (comme le vecteur phage lambda). La taille du génome de l'organisme est de 3000 Mb dans cet exemple.
clones
Ainsi, environ 688 060 clones sont nécessaires pour garantir une probabilité de 99% qu'une séquence d'ADN donnée de ce génome de trois milliards de paires de bases soit présente dans une banque utilisant un vecteur avec une taille d'insert de vingt mille paires de bases.
Applications[modifier | modifier le code]
Après la création d'une banque, le génome d'un organisme peut être séquencé pour étudier comment les gènes affectent un organisme ou pour comparer des organismes similaires au niveau du génome. Les études d'association pangénomiques susmentionnées peuvent identifier des gènes candidats issus de nombreux traits fonctionnels. Les gènes peuvent être isolés grâce à des banques génomiques et utilisés sur des lignées cellulaires humaines ou des modèles animaux pour poursuivre les recherches[18]. En outre, la création de clones de haute fidélité avec une représentation précise du génome - et sans problèmes de stabilité - contribuerait effectivement à servir d'intermédiaire pour le séquençage "méthode globale" (ou "shotgun") ou l'étude de gènes complets dans l'analyse fonctionnelle[10].
Séquençage hiérarchique[modifier | modifier le code]

Une utilisation majeure des banques génomiques est le séquençage "shotgun" hiérarchique, également appelé séquençage top-down, basé sur un séquençage par cartographie ou clone par clone. Cette stratégie a été développée dans les années 1980 pour le séquençage de génomes entiers avant que des techniques de séquençage à haut débit ne soient disponibles. Les clones individuels des banques génomiques peuvent être cisaillés en fragments plus petits, généralement de 500 pb à 1000 pb, qui sont plus faciles à gérer pour le séquençage[4]. Une fois qu'un clone d'une banque génomique est séquencé, la séquence peut être utilisée pour cribler la banque pour d'autres clones contenant des inserts qui chevauchent le clone séquencé. Tout nouveau clone se chevauchant peut ensuite être séquencé pour former un contig. Cette technique, appelée arpentage chromosomique, peut être exploitée pour séquencer des chromosomes entiers[2]. Le séquençage "shotgun" du génome entier est une autre méthode de séquençage du génome qui ne nécessite pas de banque de vecteurs de grande capacité. Au lieu de cela, il utilise des algorithmes informatiques pour assembler des lectures de séquences courtes pour couvrir l'ensemble du génome. Les banques génomiques sont souvent utilisées en combinaison avec le séquençage "shotgun" du génome entier pour cette raison. Une cartographie haute résolution peut être créée en séquençant les deux extrémités des inserts de plusieurs clones dans une banque génomique. Cette carte fournit des séquences de distances connues, qui peuvent être utilisées pour faciliter l'assemblage des lectures de séquence acquises par le séquençage "shotgun"[4]. La séquence du génome humain, qui a été déclarée complète en 2003, a été assemblée à l'aide d'une banque BAC et d'un séquençage "shotgun"[19], [20].
Études d'association à l'échelle du génome[modifier | modifier le code]
Les études d'association pangénomiques sont des applications générales pour trouver des cibles génétiques spécifiques et des polymorphismes au sein de l'humanité. En fait, le projet International HapMap a été créé grâce à un partenariat de scientifiques et d'agences de plusieurs pays pour cataloguer et utiliser ces données[21]. Le but de ce projet est de comparer les séquences génétiques de différents individus pour élucider les similitudes et les différences au sein des régions chromosomiques. Les scientifiques de tous les pays participants répertorient ces attributs avec des données provenant de populations d'ascendance africaine, asiatique et européenne. De telles évaluations à l'échelle du génome peuvent conduire à d'autres thérapies diagnostiques et médicamenteuses tout en aidant les futures équipes à se concentrer sur l'organisation des thérapies en tenant compte des caractéristiques génétiques. Ces concepts sont déjà exploités en génie génétique. Par exemple, une équipe de recherche a construit un vecteur navette PAC qui crée une banque représentant une couverture double du génome humain[18]. Cela pourrait servir de ressource inédite pour identifier des gènes ou des ensembles de gènes à l'origine de maladies. De plus, ces études peuvent servir de moyen performant pour étudier la régulation transcriptionnelle comme cela a été vu dans l'étude des baculovirus[22]. Dans l'ensemble, les progrès dans la construction de banques de génomes et le séquençage d'ADN ont permis une découverte efficace de différentes cibles moléculaires[5]. L'assimilation de ces caractéristiques par des méthodes aussi efficaces peut accélérer l'emploi de nouveaux médicaments-candidats.
Voir aussi[modifier | modifier le code]
Notes[modifier | modifier le code]
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Genomic library » (voir la liste des auteurs).
Références[modifier | modifier le code]
- Losick, Richard, Watson, James D., Tania A. Baker, Bell, Stephen, Gann, Alexander et Levine, Michael W., Molecular biology of the gene, San Francisco, Pearson/Benjamin Cummings, , 841 p. (ISBN 978-0-8053-9592-1)
- Russell, David W. et Sambrook, Joseph, Molecular cloning : a laboratory manual, Cold Spring Harbor, N.Y, Cold Spring Harbor Laboratory, (ISBN 978-0-87969-577-4, lire en ligne)
- Hartwell, Leland, Genetics : from genes to genomes, Boston, McGraw-Hill Higher Education, , 887 p. (ISBN 978-0-07-284846-5, lire en ligne)
- Muse, Spencer V. et Gibson, Greg, A primer of genome science, Sunderland, Mass, Sinauer Associates, , 378 p. (ISBN 978-0-87893-232-0, lire en ligne)
- « Application of large-scale sequencing to marker discovery in plants », J. Biosci., vol. 37, no 5, , p. 829–41 (PMID 23107919, DOI 10.1007/s12038-012-9253-z)
- « Nucleotide sequence of bacteriophage phi X174 DNA », Nature, vol. 265, no 5596, , p. 687–95 (PMID 870828, DOI 10.1038/265687a0, Bibcode 1977Natur.265..687S)
- « Shared molecular and functional frameworks among five complex human disorders: a comparative study on interactomes linked to susceptibility genes », PLOS One, vol. 6, no 4, , e18660 (PMID 21533026, PMCID 3080867, DOI 10.1371/journal.pone.0018660, Bibcode 2011PLoSO...618660M)
- « Genome-wide association study identifies genetic variation in neurocan as a susceptibility factor for bipolar disorder », Am. J. Hum. Genet., vol. 88, no 3, , p. 372–81 (PMID 21353194, PMCID 3059436, DOI 10.1016/j.ajhg.2011.01.017)
- « Construction and characterization of a bacterial artificial chromosome library from chili pepper », Mol. Cells, vol. 12, no 1, , p. 117–20 (PMID 11561720)
- « Construction of bacterial artificial chromosome (BAC/PAC) libraries », Curr Protoc Hum Genet, vol. Chapter 5, , p. 5.15.1–5.15.33 (PMID 18428289, DOI 10.1002/0471142905.hg0515s21)
- John R. McCarrey, Williams, Steven J. et Barton E. Slatko, Laboratory investigations in molecular biology, Boston, Jones and Bartlett Publishers, , 235 p. (ISBN 978-0-7637-3329-2, lire en ligne)
- Peterson, Jeffrey Tomkins et David Frisch, « Construction of Plant Bacterial Artificial Chromosome (BAC) Libraries: An Illustrated Guide », Journal of Agricultural Genomics, vol. 5, (lire en ligne)
- « Construction and characterization of a human bacterial artificial chromosome library », Genomics, vol. 34, no 2, , p. 213–8 (PMID 8661051, DOI 10.1006/geno.1996.0268)
- (en) Anthony JF Griffiths, William M. Gelbart, Jeffrey H. Miller et Richard C. Lewontin, « Cloning a Specific Gene », Modern Genetic Analysis, (lire en ligne, consulté le )
- « Cloning Genomic DNA »(Archive.org • Wikiwix • Archive.is • Google • Que faire ?), University College London (consulté le )
- « Archived copy » [archive du ] (consulté le )
- Blaber, « Genomic Libraries » (consulté le )
- « An arrayed human genomic library constructed in the PAC shuttle vector pJCPAC-Mam2 for genome-wide association studies and gene therapy », Gene, vol. 496, no 2, , p. 103–9 (PMID 22285925, PMCID 3488463, DOI 10.1016/j.gene.2012.01.011)
- « Sequencing technologies and genome sequencing », J. Appl. Genet., vol. 52, no 4, , p. 413–35 (PMID 21698376, PMCID 3189340, DOI 10.1007/s13353-011-0057-x)
- Pennisi E, « Human genome. Reaching their goal early, sequencing labs celebrate », Science, vol. 300, no 5618, , p. 409 (PMID 12702850, DOI 10.1126/science.300.5618.409)
- « HapMap Homepage »
- « Construction and application of a baculovirus genomic library », Z. Naturforsch. C, vol. 64, nos 7–8, , p. 574–80 (PMID 19791511, DOI 10.1515/znc-2009-7-817)
Bibliographie[modifier | modifier le code]
Klug, Cummings, Spencer, Palladino, Essentials of Genetics, Pearson, , 355–264 p. (ISBN 978-0-321-61869-6)










![{\displaystyle N={\frac {ln(1-0.99)}{ln[1-{\frac {2.0\times 10^{4}pb}{3.0\times 10^{9}pb}}]}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/f7f90992c0eba712b8d26f6f2c8d7cbf01e03030)

