Génétique humaine
La génétique humaine est une branche de la génétique s'occupant de l'espèce animale Homo sapiens, c’est-à-dire l'être humain.
Nombre de chromosomes
[modifier | modifier le code]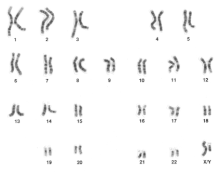
L'être humain possède 46 chromosomes répartis en 23 paires : 22 paires d'autosomes et 1 paire de gonosomes ou chromosomes sexuels, appelés X et Y. Les hommes possèdent un chromosome X et un chromosome Y. Les femmes possèdent 2 chromosomes X.
ADN mitochondrial
[modifier | modifier le code]En plus des chromosomes contenus dans le noyau, les cellules humaines possèdent de l'ADN contenu dans les mitochondries (ADN circulaire)
Cette information génétique est transmise essentiellement par la mère à 99 %, car les mitochondries sont surtout transmises par le cytoplasme de l'ovocyte. Comme cet ADN n'est pas soumis aux lois génétiques de la reproduction sexuée, il n'est pas ou peu soumis aux recombinaisons génétiques. Cependant le taux de mutation reste relativement élevé. Ces raisons font que cet ADN a été privilégié pour l'étude des grandes migrations humaines depuis 200 000 ans.
Nombre de gènes
[modifier | modifier le code]On estime le nombre de gènes codant une protéine chez l'être humain à environ 20 000. Ces estimations ont été rendues possibles depuis le séquençage du génome humain. Cependant, les estimations varient encore, donnant des chiffres compris entre 18 877 et 22 619 en 2009. Au démarrage du projet génome humain on estimait ce nombre à 100 000 en 1990, contre 6 700 000 en 1964.
Notons par ailleurs que la majorité du génome humain est composée de séquences ne codant pas de protéines. Ces séquences correspondent notamment à des régions régulatrices de l'ADN. En effet, ces gènes codants ne représentent qu'environ 1,1% du génome (35 Mb), en ne tenant compte que des régions codantes (exclusion des régions régulatrices).
Ainsi, la taille du génome humain est approximativement de 3,2 milliards de paires de nucléotides. Si l'on déroulait l'ADN contenu dans chaque cellule, on obtiendrait une chaîne d'environ deux mètres de longueur, ce qui correspond à 5,8 milliards nucléotides[1].
Séquençage du génome humain
[modifier | modifier le code]Le séquençage du génome humain a été achevé en 2003, grâce au projet Génome humain initié en 1988. Il s'agit, en fait, d'une compilation de données recueillies sur plusieurs individus.
Le premier séquençage fait sur un seul individu a été publié en [2]. La variation entre deux génomes humains est d'environ une base pour 1 000[3], ce qui est environ un tiers de moins que pour les gorilles[4] et dix fois moins que chez la mouche drosophile[5].
Depuis, plusieurs entreprises américaines proposent aux particuliers une analyse de leur génome par séquençage de quelques centaines de milliers de nucléotides sélectionnés comme étant les plus intéressants, permettant d'établir un profil de risque théorique. Cette attitude, essentiellement commerciale, est contestable car non étayée scientifiquement pour le tout venant et au bénéfice incertain[6].
Dans le génome publié par le projet Génome humain il manquait près de 8 % de l'ADN (soit l'équivalent d'un chromosome entier), notamment au niveau des centromères et des télomères. En 2022, après seulement trois ans de travaux, un consortium de plus de cent chercheurs publie le premier génome complet d'un même individu. Il comprend 3,055 milliards de paires de bases, et 19 969 gènes. Parmi ces gènes, 3604 n'étaient pas encore connus (dont seulement 140 susceptibles d'être exprimés chez un même individu). Le rapport du consortium identifie de plus, par la confrontation de diverses bases de données, deux millions de variants supplémentaires des gènes, concernant notamment 622 gènes dont l'importance médicale est connue[7],[8],[9].
Composition du génome humain
[modifier | modifier le code]L'ascendance des humains se reflète dans leur signature génomique[10]. La génomique comparative montre que sur les 23 000 gènes humains, 37 % ont des homologues chez les procaryotes, 28 % chez les eucaryotes unicellulaires, 16 % chez les animaux, 13 % chez les vertébrés et 6 % chez les primates[11].
Près de 8 % du génome humain est constitué de séquences d'origine rétrovirale[12],[13]. Ces rétrovirus endogènes représentent des vestiges d'intégrations virales ancestrales qui font maintenant partie du génome humain. La plupart de ces séquences ont subi plusieurs mutations dues à l'évolution, rendant le rétrovirus généralement défectif mais certains ont gardé leur pouvoir infectieux et jouent même un rôle dans certains aspects physiologiques associés à la reproduction et au développement humain (comme le HERV-W de l'être humain, qui participe à un des mécanismes assurant la formation du placenta)[12].
La taille et la composition du génome sont le résultat de nombreuses pressions évolutives qui s'exercent sur les populations. La taille du génome n'est pas corrélée, pour autant, à la complexité de l'organisme considéré, et la composition du génome humain réfute la vision anthropocentrée d'une marche linéaire de la vie vers la complexité et le « progrès » (du plus simple, les bactéries et les unicellulaires, vers les différents groupes d'invertébrés, puis vers les vertébrés pour finir aux mammifères et à l'être humain)[14]. Comme le dit le généticien Michael Lynch (en) : « Malgré les aspects uniques de la biologie humaine, notre génome n'est pas particulièrement remarquable quant à sa structure ou l'organisation de ses gènes, au moins du point de vue multicellulaire, et il a la plupart des caractéristiques qui sont la marque de l'évolution des génomes eucaryotes[15] ».
Lien entre gènes et caractères physiques
[modifier | modifier le code]Beaucoup de spécificités individuelles ou familiales comme la couleur de la peau, la taille, dépendent d'un grand nombre de gènes. Seuls quelques caractères ne dépendent que d'un seul gène. C'est le cas de la plupart des maladies génétiques, mais aussi de certains caractères physiques et physiologiques :
| Caractère dominant | Caractère récessif | Références |
|---|---|---|
| « Pic de veuve (en) » | Pas de « pic de veuve » | [16],[17] |
| Fossette (menton, joue) | Pas de fossette | [18],[19] |
| Fossette du menton | pas de fossette du menton | [20] |
| Capacité à détecter le PTC | Incapacité à détecter le PTC | [21] |
| Lobes des oreilles non attachés | Lobes des oreilles attachés | [18],[22],[23] |
| Iris non bleu | Iris bleu | |
| Tache de rousseur | Pas de Tache de rousseur | [18],[24] |
| Cérumen de type humide | Cérumen de type sec | [22],[25] |
Mosaïcisme génétique : Hors des maladies génétiques, un certain nombre de variants de gènes ont été identifiées comme des facteurs de risque de survenue de maladie commune (cancer du sein, maladies cardio-vasculaires...). Malgré l'existence d'un gène TP53 (aussi dénommé «gardien du génome»), dont le rôle est de réparer les dommages causés à l’ADN, plus le corps humain vieillit, plus il se présente en réalité comme une mosaïque complexe de « grappes de cellules » aux génomes légèrement différents. Ceci est du aux mutations issues d'erreurs de duplication de l'ADN, ou accumulée, à la suite notamment de l'exposition aux UV solaires, à des polluants, aliments et produits cancérigènes (fumées...) qui induisent dans certaines cellules des mutations, ensuite transmissibles aux cellules filles. Une étude récente ayant porté sur 29 types de tissus humains différents a confirmé que la mosaïcité des organes est plus élevée pour ceux dont les cellules doivent se reproduire fréquemment (peau, sang, et muqueuse pulmonaire, tube digestif, œsophage notamment)[26],[27],[28].
Des variantes génétiques peuvent aussi modifier la réponse de l'individu à certains médicaments (clopidogrel, antivitamine K...). L'étude de ces derniers cas fait l'objet de la pharmacogénomique.
Notes et références
[modifier | modifier le code]- (en) J. D. Watson et F. H. C. Crick, « Molecular of Nucleic Acids: A structure for Deoxyribose Nucleic Acid », Nature, vol. 171, no 4356, 25 avril 1953, p. 737-738 (PMID 13054692, DOI 10.1038/171737a0, Bibcode 1953Natur.171..737W, lire en ligne [archive])
- Levy S, Sutton G, Pauline C, The diploid genome sequence of an individual human, PLoS Biol 5(10): e254. doi:10.1371/journal.pbio.0050254
- Rotimi CN, Jorde LB, Ancestry and disease in the age of genomic medicine, N Engl J Med, 2010;363:1551-1558
- Yu N, Jensen-Seaman MI, Chemnick L, Ryder O, Li WH, Nucleotide diversity in gorillas, Genetics, 2004;166:1375-1383
- Li W-H, Sadler LA, Low nucleotide diversity in man, Genetics, 1991;129:513-523
- Hunter DJ, Khoury MJ, Drazen JM, Letting the genome out of the bottle — Will we get our wish?, N Eng J Med, 2008;358:105-107
- William Rowe-Pirra, « Le génome humain enfin séquencé dans son intégralité », Pour la science, no 536, , p. 6-7.
- (en) Sergey Nurk, Sergey Koren, Arang Rhie, Mikko Rautiainen, Andrey V. Bzikadze et al., « The complete sequence of a human genome », Science, vol. 376, no 6588, , p. 44-53 (DOI 10.1126/science.abj6987, lire en ligne
 , consulté le ).
, consulté le ).
- (en) Mitchell R. Vollger, Xavi Guitart, Philip C. Dishuck, Ludovica Mercuri, William T. Harvey et al., « Segmental duplications and their variation in a complete human genome », Science, vol. 376, no 6588, (DOI 10.1126/science.abj6965, lire en ligne
 , consulté le ).
, consulté le ).
- L'ascendance humaine se reflète dans la signature génomique
- (en) Margaret McFall-Ngaia et al, « Animals in a bacterial world, a new imperative for the life sciences », Proc. Natl. Acad. Sci. USA, vol. 110, no 9, , p. 3229–3236 (DOI 10.1073/pnas.1218525110).
- Anne-Geneviève Marcelin et Vincent Calvez (Sous la direction de Jean-Marie Huraux), Traité de virologie médicale, Paris, Éditions Estem, , 699 p. (ISBN 2-84371-203-3), « Rétrovirus humains émergents », p. 615
- « "Les humains sont apparentés aux virus" », sur lemonde.fr, (consulté le ).
- Cédric Grimoult, Histoire de l'évolutionnisme contemporain en France, 1945-1995, Librairie Droz, , p. 119.
- (en) Michael Lynch, The Origins of Genome Architecture, Sinauer Associates, , p. 43.
- Neil Campbell, Biology, San Francisco, Benjamin Cummings, , p. 265
- Online Mendelian Inheritance in Man, ID=194000
- Singapore Science Centre: ScienceNet|Life Sciences|Genetics/ Reproduction
- Online Mendelian Inheritance in Man, ID=126100
- Online Mendelian Inheritance in Man, ID=119000
- Natural selection at work in genetic variation to taste
- (en) Cruz-Gonzalez L., Lisker R., « Inheritance of ear wax types, ear lobe attachment and tongue rolling ability. », Acta Anthropogenet., vol. 6, no 4, , p. 247-54 (PMID 7187238)
- Online Mendelian Inheritance in Man, ID=128900
- Xue-Jun Zhang et al. A Gene for Freckles Maps to Chromosome 4q32–q34 Journal of Investigative Dermatology (2004) 122, 286–290.
- Online Mendelian Inheritance in Man, ID=117800
- Ledford H (2019) The human body is a mosaic of different genomes ; Survey finds that ‘normal’ human tissues are riddled with mutations. NEWS 06 juin 2019
- Martincorena, I., Roshan, A., Gerstung, M., Ellis, P., Van Loo, P., McLaren, S., ... & Stebbings, L. (2015). High burden and pervasive positive selection of somatic mutations in normal human skin. Science, 348(6237), 880-886.
- Martincorena, I., Fowler, J. C., Wabik, A., Lawson, A. R., Abascal, F., Hall, M. W., ... & Fitzgerald, R. C. (2018) Somatic mutant clones colonize the human esophagus with age. Science, 362(6417), 911-917.
Annexes
[modifier | modifier le code]Bibliographie
[modifier | modifier le code]- Bertrand Jordan, L'humanité au pluriel : la génétique et la question des races, Paris, Seuil, coll. « Science ouverte », , 227 p. (ISBN 978-2-02-096658-0)
- Laurence Perbal, Une brève histoire de la génétique humaine, Hermann éditeur (collection Histoire des sciences), Paris, 2014 ; 200 p. (ISBN 978-2-70568966-7)
- Evelyne Heyer, L'odyssée des gènes, Flammarion, , 384 p. (ISBN 9782081517967, lire en ligne)
