« Paléontologie moléculaire » : différence entre les versions
création par clonage et traduction depuis l'anglais Balises : Nowiki dans un article Liens d’homonymie |
(Aucune différence)
|
Version du 26 avril 2024 à 01:39
La paléontologie moléculaire fait référence à la récupération et à l'analyse de l'ADN, des protéines, des glucides ou des lipides, ainsi que de leurs produits diagénétiques, à partir d'anciens restes humains, animaux et végétaux.
Description
Le domaine de la paléontologie moléculaire a fourni des informations importantes sur les événements évolutifs, les diasporas d'espèces, la découverte et la caractérisation d'espèces disparues[1],[2]. loo En peu de temps, les progrès dans le domaine de la paléontologie moléculaire ont permis aux scientifiques d'approfondir les questions évolutives au niveau génétique plutôt que de s'appuyer uniquement sur la variation phénotypique. En appliquant des techniques d'analyse moléculaire à l'ADN des restes d'animaux récents, on peut quantifier le niveau de parenté entre deux organismes pour lesquels l'ADN a été récupéré[3]. En utilisant diverses techniques biotechnologiques telles que l’isolement, l’amplification et le séquençage de l’ADN[4], les scientifiques ont pu acquérir de nouvelles connaissances sur la divergence et l’histoire évolutive d’innombrables organismes récemment éteints. En février 2021, des scientifiques ont rapporté, pour la première fois, le séquençage de l'ADN de restes d'animaux, en l'occurrence un mammouth, vieux de plus d'un million d'années, le plus ancien ADN séquencé à ce jour[5],[6].
Dans les temps anciens, les hétérogénéités de composition des restes carbonés d'une diversité d'animaux, allant du Néoprotérozoïque au Récent, ont été liées à des signatures biologiques codées dans des biomolécules modernes via une cascade de réactions de fossilisation oxydative[7],[8],[9],[10]. La composition macromoléculaire des fossiles carbonés, certains d'âge tonien[11], préserve les signatures biologiques reflétant la biominéralisation originale, les types de tissus, le métabolisme et les affinités relationnelles (phylogénie)[9].
Histoire
L'étude de la paléontologie moléculaire aurait commencé avec la découverte par Abelson d'acides aminés vieux de 360 millions d'années conservés dans des coquilles fossiles[12].[12] Cependant, Svante Pääbo est souvent considéré comme le fondateur du domaine de la paléontologie moléculaire[13].
Le domaine de la paléontologie moléculaire a connu plusieurs avancées majeures depuis les années 1950 et constitue un domaine en constante croissance. Vous trouverez ci-dessous une chronologie montrant les contributions notables qui ont été apportées.
Chronologie
milieu des années 1950 : Abelson a découvert des acides aminés préservés dans des coquilles fossiles vieilles d’environ 360 millions d’années. Idée produite de comparer les séquences d'acides aminés fossiles avec un organisme existant afin que l'évolution moléculaire puisse être étudiée.[12].
Années 1970 : Les peptides fossiles sont étudiés par analyse des acides aminés[14].[14] Commencez à utiliser des peptides entiers et des méthodes immunologiques[15].
Fin des années 1970 : les paléobotanistes (peut également être orthographié comme paléobotanistes) ont étudié les molécules de plantes fossiles bien préservées[16].
1984 : Premier séquençage réussi de l'ADN d'une espèce disparue, le quagga, une espèce ressemblant à un zèbre[1].
1991 : Article publié sur l'extraction réussie de protéines à partir de l'os fossile d'un dinosaure, en particulier du séismosaure[17].
2005 : Des scientifiques ressuscitent le virus grippal disparu de 1918[18].
2006 : Les segments de séquences d'ADN nucléaire de Néandertal commencent à être analysés et publiés[19].
2007 : Des scientifiques synthétisent à partir de zéro l'intégralité du rétrovirus endogène humain (HERV-K) éteint[20].
2010 : Une nouvelle espèce d'hominidé primitif, les Dénisoviens, découverte à partir de génomes mitochondriaux et nucléaires récupérés à partir d'os trouvés dans une grotte en Sibérie. L'analyse a montré que le spécimen de Denisovan vivait il y a environ 41 000 ans et partageait un ancêtre commun avec les humains modernes et les Néandertaliens il y a environ 1 million d'années en Afrique[21].
2013 : Le premier génome complet de Néandertal est séquencé avec succès. Plus d'informations peuvent être trouvées sur le projet du génome néandertalien[22].
2013 : Un spécimen vieux de 400 000 ans avec un reste d'ADN mitochondrial séquencé et s'avère être un ancêtre commun des Néandertaliens et des Dénisoviens, Homo heidelbergensis[23].
2013 : Mary Schweitzer et ses collègues proposent le premier mécanisme chimique expliquant la préservation potentielle des cellules et des tissus mous des vertébrés dans les archives fossiles. Le mécanisme propose que les radicaux libres d'oxygène, potentiellement produits par le fer rédox-actif, induisent la réticulation des biomolécules. Ce mécanisme de réticulation est quelque peu analogue à la réticulation qui se produit lors de la fixation des tissus histologiques, comme avec le formaldéhyde. Les auteurs suggèrent également que la source de fer serait l'hémoglobine de l'organisme décédé[24].
2015 : Une dent fossile vieille de 110 000 ans contenant de l'ADN de Dénisoviens a été signalée[25][26].
2018 : Les paléobiologistes moléculaires lient mécaniquement les polymères de composition hétérocyclique N-, O-, S (AGE/ALE, comme mentionné dans la publication citée, Wiemann et al. 2018) dans les restes fossiles carbonés aux biomolécules structurelles des tissus d'origine. Grâce à la réticulation oxydative, un processus similaire à la réaction de Maillard, les résidus d'acides aminés nucléophiles se condensent avec des espèces carbonylées réactives dérivées de lipides et de sucres[8]. Les processus de fossilisation de biomolécules, identifiés via la spectroscopie Raman de tissus modernes et fossiles, la modélisation expérimentale et l'évaluation des données statistiques, comprennent la glycosylation avancée et la lipoxydation avancée[8].
2019 : Un laboratoire indépendant de paléontologues moléculaires confirme la transformation de biomolécules par glycosylation avancée et lipoxydation lors de la fossilisation[10]. Les auteurs utilisent la spectroscopie infrarouge à transformée de Fourier synchrotron.
2020 : Wiemann et ses collègues identifient des signatures biologiques reflétant la biominéralisation originale, les types de tissus, le métabolisme et l'affinité relationnelle (phylogénie) dans les hétérogénéités de composition préservées d'une diversité de fossiles d'animaux carbonés[9]. Il s'agit de la première analyse à grande échelle de fossiles allant du Néoprotérozoïque au Récent, et du premier enregistrement publié de signaux biologiques trouvés dans la matière organique complexe[9]. Les auteurs s’appuient sur des analyses statistiques d’un ensemble de données de spectroscopie Raman d’une taille unique.
2021 : Les géochimistes trouvent des signaux de type tissulaire dans la composition de fossiles carbonés remontant au Tonien[11] et appliquent ces signaux pour identifier les épibiontes. Les auteurs utilisent la spectroscopie Raman.
2022 : Les données de spectroscopie Raman révélant des modèles de fossilisation de biomolécules structurelles ont été répliquées avec la spectroscopie infrarouge à transformée de Fourier et une diversité de différents instruments Raman, filtres et sources d'excitation[27].
2023 : La première description chimique approfondie de la façon dont les cellules et tissus biologiques originaux se fossilisent est publiée. Surtout, l’étude montre que l’hypothèse des radicaux libres d’oxygène (proposée par Mary Schweitzer et ses collègues en 2013) est dans de nombreux cas identique à l’hypothèse de la formation AGE/ALE (proposée par Jasmina Wiemann et ses collègues en 2018). Les hypothèses combinées, ainsi que la maturation thermique et la carbonisation, forment un cadre souple pour la fossilisation des cellules biologiques et des tissus[7].
Le quagga
Le premier séquençage réussi de l'ADN d'une espèce disparue a eu lieu en 1984, à partir d'un spécimen de musée vieux de 150 ans du quagga, une espèce ressemblant à un zèbre[1]. L'ADN mitochondrial (également connu sous le nom d'ADNmt) a été séquencé à partir du muscle desséché du quagga et s'est avéré différer par 12 substitutions de bases de l'ADN mitochondrial d'un zèbre de montagne. Il a été conclu que ces deux espèces avaient un ancêtre commun il y a 3 à 4 millions d'années, ce qui est cohérent avec les preuves fossiles connues de l'espèce[28].
Dénisoviens
Les Dénisoviens d'Eurasie, une espèce d'hominidé apparentée aux Néandertaliens et aux humains, ont été découverts à la suite du séquençage de l'ADN d'un spécimen vieux de 41 000 ans récupéré en 2008. L'analyse de l'ADN mitochondrial d'un os de doigt récupéré a montré que le spécimen était génétiquement distinct des humains et des Néandertaliens. Il a été découvert plus tard que deux dents et un os d'orteil appartenaient à des individus différents de la même population. L'analyse suggère que les Néandertaliens et les Dénisoviens étaient déjà présents dans toute l'Eurasie lorsque les humains modernes sont arrivés[22] In November 2015, scientists reported finding a fossil tooth containing DNA from Denisovans, and estimated its age at 110,000-years-old.[25],[26].[21] En novembre 2015, des scientifiques ont rapporté avoir découvert une dent fossile contenant de l'ADN de Dénisoviens et ont estimé son âge à 110 000 ans.
Analyse de l'ADN mitochondrial
L'ADNmt de l'os du doigt de Denisovan diffère de celui de l'homme moderne par 385 bases (nucléotides) dans le brin d'ADNmt sur environ 16 500, alors que la différence entre l'homme moderne et l'homme de Néandertal est d'environ 202 bases. En revanche, la différence entre les chimpanzés et les humains modernes est d’environ 1 462 paires de bases d’ADNmt[21]. Cela suggère une période de divergence il y a environ un million d'années. L'ADNmt d'une dent présentait une grande similitude avec celui de l'os du doigt, ce qui indique qu'ils appartenaient à la même population[29]. À partir d’une deuxième dent, une séquence d’ADNmt a été récupérée, montrant un nombre étonnamment élevé de différences génétiques par rapport à celle trouvée dans l’autre dent et le doigt, suggérant un degré élevé de diversité d’ADNmt. Ces deux individus de la même grotte présentaient une plus grande diversité que celle observée parmi les Néandertaliens échantillonnés dans toute l'Eurasie et étaient aussi différents que les humains modernes de différents continents[30].
Analyse du génome nucléaire
L'isolement et le séquençage de l'ADN nucléaire ont également été réalisés à partir de l'os du doigt de Denisova. Ce spécimen présentait un degré inhabituel de préservation de l’ADN et un faible niveau de contamination. Ils ont pu réaliser un séquençage génomique presque complet, permettant une comparaison détaillée avec l’Homme de Néandertal et l’homme moderne. De cette analyse, ils ont conclu que, malgré l'apparente divergence de leur séquence mitochondriale, la population de Denisova et celle de Néandertal partageaient une branche commune de la lignée menant aux humains africains modernes. Le temps moyen estimé de divergence entre les séquences de Denisovan et de Néandertal est il y a 640 000 ans, et le temps entre ces deux séquences et les séquences des Africains modernes est il y a 804 000 ans. Ils suggèrent que la divergence de l'ADNmt de Denisova résulte soit de la persistance d'une lignée purgée des autres branches de l'humanité par dérive génétique, soit d'une introgression d'une lignée d'hominidés plus ancienne[29].
Homo heidelbergensis

Homo heidelbergensis a été découvert pour la première fois en 1907 près de Heidelberg, en Allemagne, puis également ailleurs en Europe, en Afrique et en Asie[31],[32].
Cependant, ce n'est qu'en 2013 qu'un spécimen avec un ADN récupérable a été découvert, dans un fémur vieux d'environ 400 000 ans découvert dans la grotte Sima de los Huesos en Espagne. Le fémur s’est avéré contenir à la fois de l’ADNmt et de l’ADN nucléaire. Les améliorations apportées aux techniques d'extraction d'ADN et de préparation de bibliothèques ont permis d'isoler et de séquencer avec succès l'ADNmt. Cependant, l'ADN nucléaire s'est avéré trop dégradé dans le spécimen observé et a également été contaminé par l'ADN d'un ancien ours des cavernes (Ursus deningeri) présent dans la grotte[33]. L’analyse de l’ADNmt a révélé un lien surprenant entre le spécimen et les Dénisoviens, et cette découverte a soulevé de nombreuses questions. Plusieurs scénarios ont été proposés dans un article de janvier 2014 intitulé « Une séquence du génome mitochondrial d'un hominidé de Sima de los Huesos », élucidant le manque de convergence dans la communauté scientifique sur la façon dont Homo heidelbergensis est lié à d'autres groupes d'hominidés connus. Un scénario plausible proposé par les auteurs était que H. heidelbergensis serait un ancêtre à la fois des Dénisoviens et des Néandertaliens[33]. Les génomes nucléaires entièrement séquencés des Dénisoviens et des Néandertaliens suggèrent un ancêtre commun il y a environ 700 000 ans, et l'un des principaux chercheurs dans le domaine, Svante Paabo, suggère que ce nouveau groupe d'hominidés est peut-être ce premier ancêtre[23].
Applications
Découverte et caractérisation de nouvelles espèces
Les techniques de paléontologie moléculaire appliquées aux fossiles ont contribué à la découverte et à la caractérisation de plusieurs nouvelles espèces, dont les Dénisoviens et Homo heidelbergensis. Nous avons pu mieux comprendre le chemin emprunté par les humains pour peupler la terre, et quelles espèces étaient présentes lors de cette diaspora.
Désextinction

Il est désormais possible de faire revivre des espèces disparues grâce aux techniques de paléontologie moléculaire. Cela a été réalisé pour la première fois par clonage en 2003 avec le bouquetin des Pyrénées, un type de chèvre sauvage qui a disparu en 2000. Les noyaux des cellules du bouquetin des Pyrénées ont été injectés dans des œufs de chèvre vidés de leur propre ADN et implantés dans des mères chèvres porteuses[34]. La progéniture n’a vécu que sept minutes après sa naissance, en raison de défauts dans ses poumons. On a observé que d'autres animaux clonés présentaient des anomalies pulmonaires similaires.[35].
De nombreuses espèces ont disparu en raison directe de l’activité humaine. Quelques exemples incluent le dodo, le grand pingouin, le tigre de Tasmanie, le dauphin de rivière chinois et la tourte voyageuse. Une espèce éteinte peut être réanimée en utilisant le remplacement allélique[36] d'une espèce étroitement apparentée qui est encore vivante. En n'ayant à remplacer que quelques gènes au sein d'un organisme, au lieu de devoir reconstruire le génome d'une espèce disparue à partir de zéro, il pourrait être possible de ramener plusieurs espèces de cette manière, même les Néandertaliens.
L'éthique entourant la réintroduction d'espèces disparues est très controversée. Les critiques de la résurrection des espèces disparues affirment que cela détournerait des fonds et des ressources limitées de la protection des problèmes actuels de biodiversité mondiale[37]. Avec des taux d'extinction actuels estimés à environ 100 à 1 000 fois le taux d'extinction de base[38], il est à craindre qu'un programme de désextinction puisse apaiser les inquiétudes du public concernant la crise actuelle d'extinction massive, si l'on croit que ces espèces peuvent simplement être ramenées retour à la vie. Comme le posent les rédacteurs d’un article du Scientific American sur la désextinction : devrions-nous ramener le mammouth laineux uniquement pour laisser les éléphants disparaître entre-temps ?[37] Le principal facteur déterminant de l’extinction de la plupart des espèces à cette époque (après 10 000 BC) est la perte d'habitat, et le fait de ramener temporairement une espèce disparue ne recréera pas l'environnement dans lequel elle habitait autrefois[39].
Les partisans de la désextinction, comme George Church, parlent de nombreux avantages potentiels. La réintroduction d’une espèce clé disparue, comme le mammouth laineux, pourrait contribuer à rééquilibrer les écosystèmes qui en dépendaient autrefois. Certaines espèces disparues pourraient créer de vastes avantages pour les environnements qu’elles habitaient autrefois, si elles étaient réintroduites. Par exemple, les mammouths laineux peuvent être capables de ralentir la fonte de la toundra russe et arctique de plusieurs manières, par exemple en mangeant de l'herbe morte afin que de nouvelles herbes puissent pousser et prendre racine, et en brisant périodiquement la neige, soumettant le sol en dessous aux températures arctiques. air. Ces techniques pourraient également être utilisées pour réintroduire la diversité génétique chez une espèce menacée, ou même introduire de nouveaux gènes et traits pour permettre aux animaux de mieux rivaliser dans un environnement changeant[40].
Recherche et technologie
Lorsqu’un nouveau spécimen potentiel est découvert, les scientifiques effectuent généralement d’abord une analyse de préservation des cellules et des tissus à l’aide de techniques histologiques, puis testent les conditions de survie de l’ADN. Ils tenteront ensuite d'isoler un échantillon d'ADN en utilisant la technique décrite ci-dessous et effectueront une amplification PCR de l'ADN pour augmenter la quantité d'ADN disponible pour les tests. Cet ADN amplifié est ensuite séquencé. On prend soin de vérifier que la séquence correspond aux traits phylogénétiques de l'organisme[1]. Lorsqu’un organisme meurt, une technique appelée datation des acides aminés peut être utilisée pour vieillir l’organisme. Il inspecte le degré de racémisation de l'acide aspartique, de la leucine et de l'alanine dans les tissus. Au fil du temps, le rapport D/L (où « D » et « L » sont des images miroir l'un de l'autre) augmente de 0 à 1[41]. Dans les échantillons où le rapport D/L de l'acide aspartique est supérieur à 0,08, les anciennes séquences d'ADN ne peuvent pas être récupérées (à partir de 1996)[42].
ADN mitochondrial vs ADN nucléaire
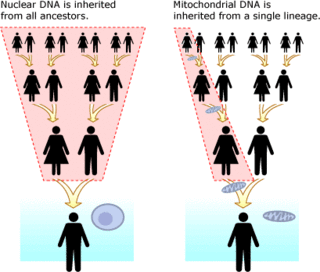
L'ADN mitochondrial (ADNmt) est distinct de l'ADN nucléaire. Il est présent dans des organelles appelées mitochondries dans chaque cellule. Contrairement à l’ADN nucléaire, qui est hérité des deux parents et réorganisé à chaque génération, une copie exacte de l’ADN mitochondrial est transmise de la mère à ses fils et filles. L’avantage d’effectuer une analyse d’ADN avec l’ADN mitochondrial est qu’il présente un taux de mutation bien inférieur à celui de l’ADN nucléaire, ce qui facilite grandement le suivi des lignées à l’échelle de plusieurs dizaines de milliers d’années. Connaissant le taux de mutation des bases de l'ADNmt[43] (chez l'homme, ce taux est également connu sous le nom d'horloge moléculaire mitochondriale humaine), on peut déterminer la durée pendant laquelle deux lignées ont été séparées. Un autre avantage de l'ADNmt est qu'il existe des milliers de copies dans chaque cellule, alors que seulement deux copies de l'ADN nucléaire existent dans chaque cellule[44]. Tous les eucaryotes, un groupe qui comprend toutes les plantes, animaux et champignons, possèdent un ADNmt[45]. Un inconvénient de l’ADNmt est que seule la lignée maternelle est représentée. Par exemple, un enfant héritera de 1/8 de son ADN de chacun de ses huit arrière-grands-parents, mais il héritera d'un clone exact de l'ADNmt de son arrière-grand-mère maternelle. Ceci est analogue à un enfant héritant uniquement du nom de famille de son arrière-grand-père paternel, et non d'un mélange des huit noms de famille.
Isolement
Il y a de nombreux éléments à prendre en compte lors de l’isolement d’une substance. Premièrement, selon de quoi il s'agit et où il se trouve, certains protocoles doivent être mis en œuvre afin d'éviter toute contamination et toute dégradation supplémentaire de l'échantillon.[4] Ensuite, la manipulation des matériaux s'effectue généralement dans une zone de travail physiquement isolée et dans des conditions spécifiques (c'est-à-dire température, humidité, etc.) également pour éviter toute contamination et toute perte supplémentaire d'échantillon[4].
Une fois le matériau obtenu, selon de quoi il s’agit, il existe différentes manières de l’isoler et de le purifier. L'extraction d'ADN à partir de fossiles est l'une des pratiques les plus populaires et différentes étapes peuvent être suivies pour obtenir l'échantillon souhaité[4]. L'ADN extrait de fossiles ensevelis dans l'ambre peut être prélevé à partir de petits échantillons et mélangé avec différentes substances, centrifugé, incubé et centrifugé à nouveau.[46] D'un autre côté, l'extraction de l'ADN des insectes peut être effectuée en broyant l'échantillon, en le mélangeant avec un tampon et en le soumettant à une purification sur des colonnes en fibre de verre[46]. En fin de compte, quelle que soit la manière dont l'échantillon a été isolé pour ces fossiles, l'ADN isolé doit pouvoir subir une amplification[4],[46],[46].
Amplification

Le domaine de la paléontologie moléculaire a grandement bénéficié de l'invention de la réaction en chaîne par polymérase (PCR), qui permet de réaliser des milliards de copies d'un fragment d'ADN à partir d'une seule copie conservée de l'ADN. L’un des plus grands défis jusqu’à présent était l’extrême rareté de l’ADN récupéré en raison de sa dégradation au fil du temps[1].
Séquençage
Le séquençage de l'ADN est effectué pour déterminer l'ordre des nucléotides et des gènes[47]. Il existe de nombreux matériaux différents à partir desquels l’ADN peut être extrait. Chez les animaux, le chromosome mitochondrial peut être utilisé pour des études moléculaires. Les chloroplastes peuvent être étudiés dans les plantes comme source principale de données de séquence[47].

En fin de compte, les séquences générées sont utilisées pour construire des arbres évolutifs[47]. Les méthodes permettant de faire correspondre les ensembles de données comprennent : la probabilité maximale, l'évolution minimale (également connue sous le nom de adhésion de voisin) qui recherche l'arbre avec la longueur totale la plus courte et la méthode de parcimonie maximale qui trouve l'arbre nécessitant le moins de changements d'état de caractère. Les groupes d'espèces définis au sein d'un arbre peuvent également être évalués ultérieurement par des tests statistiques, tels que la méthode bootstrap, pour voir s'ils sont effectivement significatifs[47].
Limites et défis
Il est difficile de trouver des conditions environnementales idéales pour préserver l’ADN là où l’organisme a été desséché et découvert, ainsi que pour maintenir son état jusqu’à l’analyse. L'ADN nucléaire se dégrade normalement rapidement après la mort par des processus hydrolytiques endogènes[42], par le rayonnement UV[1], et d'autres facteurs de stress environnementaux.
En outre, il a été démontré que les interactions avec les produits de dégradation organique du sol environnant aident à préserver les matériaux biomoléculaires[48]. Cependant, ils ont également créé le défi supplémentaire de pouvoir séparer les différents composants afin de pouvoir effectuer l'analyse appropriée sur eux[49]. Il a également été constaté que certaines de ces dégradations interfèrent avec l'action de certaines des enzymes utilisées lors de la PCR[48].
Enfin, l’un des plus grands défis dans l’extraction de l’ADN ancien, en particulier de l’ADN humain ancien, réside dans la contamination lors de la PCR. De petites quantités d’ADN humain peuvent contaminer les réactifs utilisés pour l’extraction et la PCR de l’ADN ancien. Ces problèmes peuvent être surmontés par un soin rigoureux dans la manipulation de toutes les solutions ainsi que de la verrerie et des autres outils utilisés dans le processus. Il peut également être utile qu'une seule personne effectue les extractions, afin de minimiser les différents types d'ADN présents[42].
Voir aussi
Bibliographie
![]() : document utilisé comme source pour la rédaction de cet article.
: document utilisé comme source pour la rédaction de cet article.
Liens externes
Notes et références
Notes
Références
- « Molecular paleontology », Cellular and Molecular Life Sciences, vol. 59, no 1, , p. 97–111 (PMID 11846037, DOI 10.1007/s00018-002-8408-8, S2CID 6377604)
- « Molecular paleontology: some current advances and problems », Annales de Paléontologie, vol. 90, no 2, , p. 81–102 (DOI 10.1016/j.annpal.2004.02.001, lire en ligne, consulté le )
- « A paleogenomic perspective on evolution and gene function: new insights from ancient DNA », Science, vol. 343, no 6169, , p. 1236573 (PMID 24458647, DOI 10.1126/science.1236573, S2CID 1015148)
- « Molecular Palaeontology », Encyclopedia of Life Sciences, Nature Publishing Group, , p. 1–5 (lire en ligne)
- (en) « World's oldest DNA sequenced from a mammoth that lived more than a million years ago », CNN News, (lire en ligne)
- « Million-year-old mammoth genomes shatter record for oldest ancient DNA », Nature, vol. 590, no 7847, , p. 537–538 (PMID 33597786, DOI 10.1038/d41586-021-00436-x, Bibcode 2021Natur.590..537C)
- « A chemical framework for the preservation of fossil vertebrate cells and soft tissues », Earth-Science Reviews, vol. 240, , p. 104367 (DOI 10.1016/j.earscirev.2023.104367, S2CID 257326012)
- « Fossilization transforms vertebrate hard tissue proteins into N-heterocyclic polymers », Nature Communications, vol. 9, no 1, , p. 4741 (PMID 30413693, PMCID 6226439, DOI 10.1038/s41467-018-07013-3, Bibcode 2018NatCo...9.4741W)
- « Phylogenetic and physiological signals in metazoan fossil biomolecules », Science Advances, vol. 6, no 28, , eaba6883 (PMID 32832604, PMCID 7439315, DOI 10.1126/sciadv.aba6883, Bibcode 2020SciA....6.6883W)
- « Mechanisms of soft tissue and protein preservation in Tyrannosaurus rex », Scientific Reports, vol. 9, no 1, , p. 15678 (PMID 31666554, PMCID 6821828, DOI 10.1038/s41598-019-51680-1, Bibcode 2019NatSR...915678B)
- (en) « One-billion-year-old epibionts highlight symbiotic ecological interactions in early eukaryote evolution », Gondwana Research, vol. 97, , p. 22–33 (ISSN 1342-937X, DOI 10.1016/j.gr.2021.05.008, Bibcode 2021GondR..97...22T)
- « Organic constituents of fossils », Carnegie Institute of Washington Yearbook, vol. 53, , p. 97–101
- « 2013 Gruber Genetics Prize Press Release », Gruber Foundation
- « Preservation of antigenic properties of macromolecules over 70 Myr », Nature, vol. 252, no 5478, , p. 63–64 (PMID 4139661, DOI 10.1038/252063a0, Bibcode 1974Natur.252...63D, S2CID 4286786)
- « Fossil macromolecules from cephalopod shells: Characterization, immunological response and diagenesis », Paleobiology, vol. 5, no 2, , p. 151–167 (DOI 10.1017/S0094837300006448, S2CID 88664754)
- (en) Biochemical Aspects of Evolutionary Biology, Chicago, University of Chicago Press, , 29–91 p.
- « Proteins in the fossil bone of the dinosaur, Seismosaurus », Journal of Protein Chemistry, vol. 10, no 1, , p. 75–90 (PMID 2054066, DOI 10.1007/BF01024658, S2CID 9046093)
- « Virology. Resurrected influenza virus yields secrets of deadly 1918 pandemic », Science, vol. 310, no 5745, , p. 28–29 (PMID 16210501, DOI 10.1126/science.310.5745.28, S2CID 26252589)
- « Neanderthal DNA yields to genome foray », Nature, vol. 441, no 7091, , p. 260–261 (PMID 16710377, DOI 10.1038/441260b, Bibcode 2006Natur.441..260D)
- (en) Rockefeller University, « Ancient Retrovirus Is Resurrected », Science Daily, (lire en ligne)
- « The complete mitochondrial DNA genome of an unknown hominin from southern Siberia », Nature, vol. 464, no 7290, , p. 894–897 (PMID 20336068, PMCID 10152974, DOI 10.1038/nature08976, Bibcode 2010Natur.464..894K)
- « The complete genome sequence of a Neanderthal from the Altai Mountains », Nature, vol. 505, no 7481, , p. 43–49 (PMID 24352235, PMCID 4031459, DOI 10.1038/nature12886, Bibcode 2014Natur.505...43P)
- « Hominin DNA baffles experts », Nature, vol. 504, no 7478, , p. 16–17 (PMID 24305130, DOI 10.1038/504016a, Bibcode 2013Natur.504...16C)
- « A role for iron and oxygen chemistry in preserving soft tissues, cells and molecules from deep time », Proceedings of the Royal Society B: Biological Sciences, vol. 281, no 1775, , p. 20132741 (PMID 24285202, PMCID 3866414, DOI 10.1098/rspb.2013.2741)
- (en) « In a Tooth, DNA From Some Very Old Cousins, the Denisovans », New York Times, (lire en ligne)
- « Nuclear and mitochondrial DNA sequences from two Denisovan individuals », Proceedings of the National Academy of Sciences of the United States of America, vol. 112, no 51, , p. 15696–15700 (PMID 26630009, PMCID 4697428, DOI 10.1073/pnas.1519905112, Bibcode 2015PNAS..11215696S)
- « Raman spectroscopy is a powerful tool in molecular paleobiology: An analytical response to Alleon et al. (https://doi.org/10.1002/bies.202000295) », BioEssays, vol. 44, no 2, , e2100070 (PMID 34993976, DOI 10.1002/bies.202100070, S2CID 245824320, lire en ligne)
- « DNA sequences from the quagga, an extinct member of the horse family », Nature, vol. 312, no 5991, , p. 282–284 (PMID 6504142, DOI 10.1038/312282a0, Bibcode 1984Natur.312..282H, S2CID 4313241)
- « Genetic history of an archaic hominin group from Denisova Cave in Siberia », Nature, vol. 468, no 7327, , p. 1053–1060 (PMID 21179161, PMCID 4306417, DOI 10.1038/nature09710, Bibcode 2010Natur.468.1053R)
- « Human evolution. More genomes from Denisova Cave show mixing of early human groups », Science, vol. 340, no 6134, , p. 799 (PMID 23687020, DOI 10.1126/science.340.6134.799, Bibcode 2013Sci...340..799P)
- « Is Homo heidelbergensis a distinct species? New insight on the Mauer mandible », Journal of Human Evolution, vol. 56, no 3, , p. 219–246 (PMID 19249816, DOI 10.1016/j.jhevol.2008.12.006)
- (en) The Human Lineage, John Wiley & Sons, (ISBN 978-0471214915, lire en ligne)
- « A mitochondrial genome sequence of a hominin from Sima de los Huesos », Nature, vol. 505, no 7483, , p. 403–406 (PMID 24305051, DOI 10.1038/nature12788, Bibcode 2014Natur.505..403M, S2CID 4456221, lire en ligne [archive du ])
- « Bringing Extinct Species Back To Life » [archive du ], National Geographic (consulté le )
- (en) « Extinct ibex is resurrected by cloning », Telegraph, (lire en ligne)
- « Multiplex Automated Genomic Engineering (MAGE): A machine that speeds up evolution is revolutionizing genome design » [archive du ], Wyss Institute (consulté le )
- (en) The Editors, « Why Efforts to Bring Extinct Species Back from the Dead Miss the Point », Scientific American, (lire en ligne)
- « The future of biodiversity », Science, vol. 269, no 5222, , p. 347–350 (PMID 17841251, DOI 10.1126/science.269.5222.347, Bibcode 1995Sci...269..347P, S2CID 35154695)
- « Causes of Extinction », The Encyclopedia of Earth (consulté le )
- (en) « De-Extinction Is a Good Idea », Scientific American, (lire en ligne)
- « Method: Principles » [archive du ], Amino Acid Geochronology Laboratory, Northern Arizona University (consulté le )
- « Amino acid racemization and the preservation of ancient DNA », Science, vol. 272, no 5263, , p. 864–866 (PMID 8629020, DOI 10.1126/science.272.5263.864, Bibcode 1996Sci...272..864P, hdl 2060/19980202342, S2CID 20626782)
- « Characterizing the time dependency of human mitochondrial DNA mutation rate estimates », Molecular Biology and Evolution, vol. 26, no 1, , p. 217–230 (PMID 18984905, DOI 10.1093/molbev/msn244)
- « Mitochondrial DNA » [archive du ], DNA Analyst Training, NFSTC Science Serving Justice (consulté le )
- « Mitochondrial genome evolution and the origin of eukaryotes », Annual Review of Genetics, vol. 33, , p. 351–397 (PMID 10690412, DOI 10.1146/annurev.genet.33.1.351)
- « Rapid isolation of DNA from fossil and museum specimens suitable for PCR », BioTechniques, vol. 15, no 3, , p. 432–4, 436 (PMID 8217155)
- (en) Molecular Paleontology, Encyclopedia of Paleontology, 752–754 p. (lire en ligne)
- « The biochemistry of ancient DNA in bone », Experientia, vol. 50, no 6, , p. 530–535 (PMID 7517371, DOI 10.1007/bf01921721, S2CID 27896535)
- (en) Ancient proteins in fossil bones, vol. 224, coll. « Methods in Enzymology », , 121–129 p. (ISBN 9780121821258, PMID 8264383, DOI 10.1016/0076-6879(93)24010-r), « [9] Ancient proteins in fossil bones »

