Intron
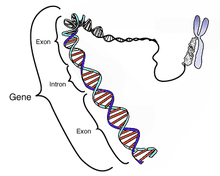
Un intron est une portion d'un gène qui est transcrite en ARN, au sein d'un ARN précurseur, et qui est ensuite éliminée par un processus d'excision programmé et qu'on ne retrouve donc pas dans l'ARN mature. On trouve principalement des introns dans les gènes codant des protéines, où ils sont présents dans l'ARN pré-messager et excisés dans l'ARNm mature[1]. Les introns sont donc des régions non codantes. On trouve aussi des introns dans des gènes codant des ARN non codants comme les ARN ribosomiques ou les ARN de transfert.
Un gène pourvu d'introns est appelé gène discontinu, gène fragmenté ou gène mosaïque.
Le processus d'excision des introns s'appelle l'épissage. Les segments d'ARN qui sont conservés après épissage des introns sont appelés des exons.
On trouve principalement des introns dans les gènes codant des protéines des organismes eucaryotes. Les gènes eucaryotes sont constitués d'une alternance d'exons et d'introns, commençant et terminant par un exon.
- Exon 1
- Intron 1
- Exon 2
- Intron 2
- …
- Intron n-1
- Exon n.
Après la transcription, l'ARN précurseur synthétisé va subir un certain nombre de modifications, dont l'épissage, au cours duquel les introns vont être excisés de l'ARN. Les exons vont quant à eux être suturés pour donner l'ARN mature par ce mécanisme d'épissage. On obtiendra donc un ARN de type Exon 1 - Exon 2 - Exon 3 - ... - Exon n.

Les introns ne jouent donc aucun rôle dans la fonction de l'ARN mature (traduction en protéine pour l'ARNm, incorporation dans le ribosome pour les ARNr...), si bien que leur fonction éventuelle reste à ce jour difficile à cerner. Leur rôle le plus important est de permettre une combinatoire lors de l'épissage. Cela permet à certains gènes de coder plusieurs protéines ou variants d'une même protéine, par épissage alternatif d'un même ARN pré-messager. Ceci permet par exemple à certains rétrovirus de produire plusieurs ARNm et donc plusieurs protéines virales à partir d'un seul promoteur de transcription et donc d'un seul pré-ARNm.
La taille des introns est très variable, allant de quelques dizaines de paires de bases, jusqu'à plusieurs dizaines de milliers[2]. La taille moyenne varie suivant les espèces et a tendance à augmenter avec la taille du génome[2].
De manière plus rare, on retrouve également des introns dans certains gènes chez les archéobactéries et chez les procaryotes[3]. Il s'agit d'introns d'un type particulier, dits introns autoépissables.
Historique[modifier | modifier le code]
La découverte des introns est due à Phillip Allen Sharp et Richard Roberts[4],[5], ce qui leur a valu le prix Nobel de physiologie ou médecine en 1993. Ils les ont observés pour la première fois dans les ARN messagers de l'adénovirus en 1976, peu avant que l'équipe de Pierre Chambon en trouve également dans des ARN messagers cellulaires comme celui de l'ovalbumine. C'est Walter Gilbert qui a inventé en 1978 la dénomination intron pour ces séquences non codantes intercalées entre les régions codantes[6] : Intron est la contraction de INTragenic RegiON.
Épissage des introns[modifier | modifier le code]
Il existe trois principaux mécanismes d'épissage des introns qui dépendent de la nature de l'intron considéré : l'épissage des introns par le splicéosome, machinerie ribonucléoprotéique agissant en trans, les introns autocatalytiques qui sont des ribozymes capables de s'exciser de manière autonome en cis, et l'épissage par des nucléases spécifiques.
Épissage des introns par le splicéosome[modifier | modifier le code]
Le mode le plus fréquent d'épissage des introns des ARN pré-messagers chez les eucaryotes est l'épissage par le splicéosome. Le splicéosome est constitué de cinq particules appelées les petites ribonucléoprotéines nucléaires (en anglais small nuclear ribonucleoprotein, ou snRNP). Chaque snRNP est constitué d'un ARN spécifique (ARNsn ou petit ARN nucléaire) et de plusieurs protéines associés en complexes. Ces différents ARNsn sont appelés U1, U2, U4, U5 et U6. L'ensemble forme un large complexe qui s'assemble sur l'intron pour réaliser l'épissage.
Pour que ces particules puissent repérer précisément les introns et leurs jonctions avec les exons à l'intérieur d'un gène, la séquence nucléotidique comporte des motifs consensus à la jonction intron-exon permettant de les identifier. Chaque intron possède au niveau de ses extrémités (5' et 3') une séquence qui sera alors reconnue par les ARNsn et ensuite clivée : en 5', on retrouve une séquence "GURAGU" (consensus) et en 3' une séquence "CAG". Ces deux séquences sont les sites de clivage des introns. De plus, les introns qui sont excisés par des splicéosomes contiennent une séquence à l'intérieur de l'intron appelé la « boîte de branchement », nécessaire à l'épissage. La séquence en 5' est également appelée la séquence donneuse.

L'excision de l'intron par un splicéosome est assez particulière : le complexe va cliver l'intron par une transestérification de liaison phosphodiester à son extrémité 5', pour le rattacher au niveau de la boîte de branchement par formation d'une liaison inhabituelle 5'-2' phosphodiester et former une boucle d'ARN de la forme d'un lasso. La séquence de clivage en 3' est ensuite reconnue et subit une seconde réaction de transestérification. L'extrémité 3'-OH de l'exon amont, libérée lors de la première étape, attaque la liaison phosphodiester à la jonction de l'intron et de l'exon aval. Ceci aboutit à la formation d'une liaison phosphodiester 5'-3' entre les deux exons et à la libération de l'intron excisé sous forme de lasso. L'intron subit enfin une réaction de débranchement spécifique qui ouvre la structure lasso au niveau de la liaison 5'-2' phosphodiester, avant d'être dégradé par des nucléases.
Lorsque tous les introns sont excisés, le pré-ARNm devient un ARNm (ARN messager mature) prêt à la traduction, après export dans le cytoplasme.
Introns autoépissables[modifier | modifier le code]
Les introns autoépissables sont des introns particuliers capables de s'épisser de manière autonome, sans l'action de molécules en trans comme le splicéosome. Ils sont hautement structurés et sont doués d'une activité catalytique de type ribozyme qui assure l'épissage. On distingue deux principales classes d'introns autoépissables, en fonction de leur organisation structurale et de leur mécanisme d'action. On les appelle introns de groupe I et introns de groupe II. Les deux types d'introns partagent le principe d'un mécanisme en deux étapes, avec deux transestérifications successives, d'abord du côté 5' de l'intron, ce qui libère le 3'-OH de l'exon amont qui agit du comme nucléophile pour faire la seconde transestérification avec l'exon aval.
- Les introns de groupe I utilisent l'hydroxyle (-OH) d'une guanosine comme premier nucléophile. Cette guanosine le plus souvent un nucléotide guanylique libre (GMP, GTP...) et parfois interne à l'intron. Cette attaque par un cofacteur nucléotidique externe a pour conséquence le fait que l'intron ne se cyclise pas sous forme de lasso, contrairement au cas de l'épissage par le splicéosome.
- Les introns de groupe II utilisent le 2'-OH d'une adénosine interne de l'intron comme premier nucléophile. L'intron est excisé sous forme de lasso, comme pour le splicéosome.
La proximité structurale et fonctionnelle entre le splicéosome et les introns de groupe II a conduit à formuler l'hypothèse que le splicéosome actuel aurait évolué à partir d'un intron de groupe II qui se serait autonomisé, en même temps qu'il a acquis la capacité d'agir en trans[7].
On trouve des introns autoépissables chez certaines bactéries et eucaryotes, en particulier dans le génome d'organites cellulaires (mitochondrie).
Nucléases spécifiques des introns[modifier | modifier le code]
Chez les eucaryotes, on trouve des introns particuliers dans les ARN de transfert, localisés dans la boucle de l'anticodon, qui sont épissés par l'action de protéines nucléaires spécifiques[8]. L'intron, dont la longueur varie entre 14 et 60 nucléotides, est toujours localisé immédiatement en 3' de l'anticodon. Il est d'abord excisé par une ARNt-nucléase spécifique, les deux extrémités de l'ARNt sont ensuite suturées par une ARNt-ligase. Enfin, le produit de cette réaction comporte un 2'-phosphate supplémentaire qui est enlevé par une phosphotransferase.
On trouve aussi parfois des introns dans les ARNt bactériens, mais ce sont des introns autoépissables de groupe II.
Origine et évolution des introns[modifier | modifier le code]
L'origine des introns et leur scénario d'apparition dans l'Évolution est un sujet de discussion depuis leur découverte[9]. Deux hypothèses principales pour expliquer que les introns excisés par le splicéosome ne sont présents que chez les eucaryotes ont été proposées :
- l'hypothèse des introns précoces, dans laquelle les gènes codant des protéines étaient interrompus par de nombreux introns dès le tout début de l'Évolution. Dans ce modèle, les introns auraient joué un rôle majeur dans l'évolution des protéines en facilitant la recombinaison et le mélange de petits modules protéiques codés par les exons (exon shuffling). Ils auraient ensuite été progressivement éliminés dans les espèces qui ne possèdent peu ou pas d'introns dans leurs gènes ;
- l'hypothèse des introns tardifs propose au contraire que les introns sont apparus uniquement chez les eucaryotes et se sont accumulés progressivement par envahissement des génomes.
L'hypothèse précoce prédit que la position des introns dans les gènes a été fixée tôt dans l'Évolution et doit être conservée entre espèces. L'analyse génomique qui se développe depuis les années 2000 suggère que le scénario probable est un mélange des deux hypothèses, où les introns actuels des eucaryotes seraient apparus très tôt, à partir des introns autoépissables de groupe II dont serait dérivé le splicéosome[10].
Notes et références[modifier | modifier le code]
- (en) Bruce Alberts, Molecular Biology of the cell : Sixth International Student Edition, Garland Science, , 1465 p. (ISBN 978-0-8153-4464-3, lire en ligne), Chapter 6 : How cells read the genome : From DNA to protein, "RNA splicing is performed by the Spliceosome", p.319
- (en) Xin Hong, Douglas G. Scofield et Michael Lynch, « Intron size, abundance, and distribution within untranslated regions of genes », Molecular Biology and Evolution, vol. 23, no 12, , p. 2392–2404 (ISSN 0737-4038, PMID 16980575, DOI 10.1093/molbev/msl111, lire en ligne, consulté le )
- F. Martínez-Abarca et N. Toro, « Group II introns in the bacterial world », Molecular Microbiology, vol. 38, no 5, , p. 917–926 (ISSN 0950-382X, PMID 11123668, lire en ligne, consulté le )
- (en) Berget S. M., Moore C., Sharp PA., « Spliced segments at the 5' terminus of adenovirus 2 late mRNA. », Proc. Natl. Acad. Sci. USA, vol. 74, , p. 3171-3175 (PMID 269380)
- (en) Chow L. T., Gelinas R. E., Broker T. R., Roberts R. J., « An amazing sequence arrangement at the 5' ends of adenovirus 2 messenger RNA. », Cell, vol. 12, , p. 1-8 (PMID 902310)
- (en) Tonegawa S., Maxam A. M., Tizard R., Bernard O., Gilbert W., « Sequence of a mouse germ-line gene for a variable region of an immunoglobulin light chain. », Proc. Natl. Acad. Sci. USA, vol. 75, , p. 1485-1489 (PMID 418414)
- Steven Zimmerly et Cameron Semper, « Evolution of group II introns », Mobile DNA, vol. 6, , p. 7 (ISSN 1759-8753, PMID 25960782, PMCID PMC4424553, DOI 10.1186/s13100-015-0037-5, lire en ligne, consulté le )
- J. Abelson, C. R. Trotta et H. Li, « tRNA splicing », The Journal of Biological Chemistry, vol. 273, no 21, , p. 12685–12688 (ISSN 0021-9258, PMID 9582290, lire en ligne, consulté le )
- Walter Gilbert, « Why genes in pieces? », Nature, vol. 271, no 5645, , p. 501 (ISSN 0028-0836, PMID 622185, lire en ligne, consulté le )
- Igor B. Rogozin, Liran Carmel, Miklos Csuros et Eugene V. Koonin, « Origin and evolution of spliceosomal introns », Biology Direct, vol. 7, , p. 11 (ISSN 1745-6150, PMID 22507701, PMCID 3488318, DOI 10.1186/1745-6150-7-11, lire en ligne, consulté le )
