ARN ribosomique 5S

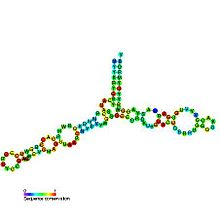

L'ARN ribosomique 5S (ARNr 5S) est l'un des petits ARN ribosomiques constituant aussi bien la grande sous-unité 50S des ribosomes de procaryotes que la grande sous-unité 60S des ribosomes cytosoliques des eucaryotes. Il est en revanche absent des ribosomes mitochondriaux des mycètes et des animaux[3]. Chez l'homme, il est constitué de 121 nucléotides[4].
Structure
Avec une masse moléculaire d'environ 40 kDa, l'ARNr 5S est un ARN relativement petit. Sa structure secondaire a pu être établie et consiste en cinq hélices, quatre boucles et une charnière[5] formant une structure en Y. Dans le génome des eucaryotes, les gène ADNr 5S s'assemblent par répétitions en tandem[6]. Le nombre de ces gènes varie d'une espèce à l'autre[7].
Les cinq hélices sont numérotées de I à V tandis que la charnière et les quatre boucles sont identifiées de A à E : les boucles C et D sont en tige-boucle tandis que les boucles B et E sont internes et la boucle A forme la charnière[7].
L'hélice III a deux résidus d'adénine hautement conservés[8]. Les études phylogénétiques montrent que les hélices I et III sont probablement très anciennes[9].
Localisation dans le ribosome
L'utilisation de plusieurs techniques d'investigation moléculaire, notamment les techniques de coloration immunohistochimique, de réticulation intermoléculaire et de cristallographie aux rayons X, ont permis de déterminer avec une grande précision la localisation de l'ARNr 5S au sein de la grande sous-unité ribosomique. Cette sous-unité est elle-même constituée de deux ARN ribosomiques chez les procaryotes — l'ARNr 5S et l'ARNr 23S — et de trois chez les eucaryotes — l'ARNr 5S, l'ARNr 5,8S et l'ARNr 28S — auxquels s'associent diverses protéines.
Chez les procaryotes, la structure tridimensionnelle de la grande sous-unité ribosomique montre une surface relativement lisse et, à l'opposé, une surface comprenant trois projections, appelées protubérance L1, protubérance centrale et tige L7/L12. La protubérance L1 et la tige L7/L12 sont disposées latéralement et entourent la protubérance centrale, dont la formation et la structure reposent sur l'ARNr 5S. La grande sous-unité ribosomique des procaryotes contient par ailleurs l'ARNr 23S ainsi que plusieurs protéines ribosomiques, dont les protéines L5, L18, L25 et L27[10].
Fonction
Le rôle exact de l'ARN ribosomique 5S n'est pas encore éclairci. Un déficit en ARNr 5S a un effet délétère plus accentué sur les cellules d'E. coli que la délétion d'autres gènes tels que ceux de l'ARNr 16S de la petite sous-unité ou de l'ARNr 23S de la grande sous-unité[11]. Les cellules dépourvues d'ARNr 5S ont une moindre capacité à produire des protéines, tandis que des études cristallographiques de ribosomes fonctionnels montrent que les protéines qui se lient à l'ARNr 5S ainsi qu'à la protubérance centrale de la grande sous-unité du ribosome pourraient jouer un rôle dans la liaison de l'ARN de transfert au ribosome[10].
Par ailleurs, la proximité topographique entre l'ARNr 23S et l'ARNr 5S, qui constituent deux centres catalytiques actifs sur le ribosome à l'activité peptidyltransférase et GTPase, a conduit à émettre l'hypothèse que l'ARNr 5S puisse agir comme médiateur coordonnant les différents centres fonctionnels du ribosome[10]. Les études tridimensionnelles par cristallographie ont par ailleurs montré que le complexe entre les protéines ribosomiques et l'ARNr 5S ainsi que d'autres composants de la protubérance centrale de la grande sous-unité du ribosome pourraient également jouer un rôle dans la formation de liaisons entre les sous-unités ainsi qu'avec les sites de liaison à l'ARN de transfert.
L'ARNr 5S des eucaryotes est produit par l'ARN polymérase III alors que la plupart des autres ARN ribosomiques d'eucaryotes proviennent du clivage d'un précurseur 45S produit par l'ARN polymérase I. On a pu montrer que, dans les ovocytes de xénopes (dactylèthre du Cap), les doigts 4 à 7 des neuf doigts de zinc du facteur de transcription TFIIIA se lient à la région centrale de l'ARNr 5S[12],[13] : cette interaction permet à la fois réprimer la transcription excessive des gènes ADNr 5S et de stabiliser leur produit de transcription jusqu'à leur utilisation pour l'assemblage des ribosomes[14].
Assemblage
L'assemblage des ribosomes des eucaryotes est un processus complexe qui implique quatre ARN ribosomiques différents et plus de 80 protéines[15]. Alors que la biogenèse des autres ARN ribosomiques constituant les sous-unités ribosomiques 60S et 40S débute dans les nucléoles par une transcription sous l'action d'une ARN polymérase I, l'ARNr 5S est unique en ce qu'il est transcrit par une ARN polymérase III à partir de gènes indépendants situés sur un locus différent[7]. Les exonucléases Rex1p, Rex2p et Rex3p traitent l'extrémité 3' des ARNr 5S. L'assemblage des constituants des ribosomes issus de la transcription des gènes correspondants a lieu dans les nucléoles, puis les ribosomes terminent leur maturation dans le cytoplasme, où a lieu notamment l'assemblage des sous-unités 60S et 40S pour former le ribosome 80S fonctionnel. Les recherches sont encore en cours concernant la détermination du moment où l'ARNr 5S est intégré au ribosome, les résultats à ce sujet étant contradictoires : une étude récente indique en effet que cette intégration aurait lieu à un stade très précoce à 90S de la maturation des ribosomes tandis que les travaux préliminaires correspondants suggéraient que cette intégration intervenait bien plus tardivement. Les plus récentes de ces données, obtenues à partir de levures, suggèrent en effet que l'ARNr 5S est incorporé dans les particules 90S sous forme d'un petit complexe ribonucléoprotéique ; en particulier, l'ARNr 5S se trouve associé à la protéine ribosomique Rp15p dans une petite ribonucléoprotéine, et ce aussi bien dans les cellules de levures que dans celles de mammifères, ce qui tend au moins à démontrer qu'il forme des particules pré-ribosomiques sous forme d'un complexe d'ARNr 5S et de Rp15p[15].
Protéines ribosomiques
Plusieurs protéines importantes interagissant avec l'ARNr 5S sont listées ci-dessous.
Protéine La
La protéine La empêche la dégradation de l'ARN par les exonucléases de la cellule[16]. La protéine La est présente chez tous les eucaryotes et se trouve dans le noyau de leurs cellules, où elle s'associe à plusieurs protéines transcrites par l'ARN polymérase III. L'interaction entre cette protéine et ces ARN fait intervenir des résidus d'uridine 3'-terminaux qui stabilisent le repliement des ARN[7],[17].
Protéine L5
La protéine ribosomique L5 interagit également avec l'ARNr 5S. Ce dernier a ceci de particulier parmi les ARN ribosomiques qu'il est présent en dehors des ribosomes au sein de particules ribonucléoprotéiques (RNP) avec la protéine L5. De tels complexes sont importés dans les nucléoles pour l'assemblage en ribosomes, de sorte que la protéine L5 est présente à la fois dans le cytoplasme et dans le noyau des cellules d'eucaryotes. Un déficit en protéine L5 fonctionnelle empêche le transport de l'ARNr 5S dans le noyau et limite l'assemblage des ribosomes[7].
Autres protéines ribosomiques
Chez les procaryotes, l'ARNr 5S se lie aux protéines L5, L18 et L25 tandis que, chez les eucaryotes, l'ARNr 5S ne se lie qu'à la protéine ribosomique L5[18]. Chez Trypanosoma brucei, l'agent infectieux à l'origine des trypanosomiases (maladie du sommeil et maladie de Chagas), l'ARNr 5S interagit avec deux protéines étroitement apparentées, P34 et P37, dont la carence conduit à une diminution du taux d'ARNr 5S fonctionnel[7].
Notes et références
- (en) Florian Mueller, Ingolf Sommer, Pavel Baranov, Rishi Matadeen, Matthias Stoldt, Jens Wöhnert, Matthias Görlach, Marin van Heel et Richard Brimacombe, « The 3D arrangement of the 23 S and 5 S rRNA in the Escherichia coli 50 S ribosomal subunit based on a cryo-electron microscopic reconstruction at 7.5 Å resolution », Journal of Molecular Biology, vol. 298, no 1, , p. 35-59 (PMID 10756104, DOI 10.1006/jmbi.2000.3635, lire en ligne)
- (en) Gregor Blaha, Güliz Gürel, Susan J. Schroeder, Peter B. Moore et Thomas A. Steitz, « Mutations Outside the Anisomycin-Binding Site Can Make Ribosomes Drug-Resistant », Journal of Molecular Biology, vol. 379, no 3, , p. 505-519 (PMID 18455733, PMCID 2442718, DOI 10.1016/j.jmb.2008.03.075, lire en ligne)
- (en) Maciej Szymanski, Miroslawa Z. Barciszewska, Volker A. Erdmann et Jan Barciszewski, « 5S Ribosomal RNA Database », Nucleic Acid Research, vol. 30, no 1, , p. 176-178 (PMID 11752286, PMCID 99124, DOI 10.1093/nar/30.1.176, lire en ligne)
- (en) Homo sapiens RNA, 5S ribosomal 17 (RNA5S17), ribosomal RNA, sur le site du NCBI.
- (en) Brian M. Lee, Jing Xu, Bryan K. Clarkson, Maria A. Martinez-Yamout, H. Jane Dyson, David A. Case, Joel M. Gottesfeld et Peter E. Wright, « Induced Fit and “Lock and Key” Recognition of 5 S RNA by Zinc Fingers of Transcription Factor IIIA », Journal of Molecular Biology, vol. 357, no 1, , p. 275-291 (PMID 16405997, DOI 10.1016/j.jmb.2005.12.010, lire en ligne)
- (en) J. Douet et S. Tourmente, « Transcription of the 5S rRNA heterochromatic genes is epigenetically controlled in Arabidopsis thaliana and Xenopus laevis », Heredity, vol. 99, no 1, , p. 5-13 (PMID 17487217, DOI 10.1038/sj.hdy.6800964, lire en ligne)
- (en) Martin Ciganda et Noreen Williams, « Eukaryotic 5S rRNA biogenesis », Wiley Interdisciplinary Reviews: RNA, vol. 2, no 4, , p. 523-533 (PMID 21957041, PMCID 3278907, DOI 10.1002/wrna.74, lire en ligne)
- (en) Jonathan P. DiNitto et Paul W. Huber, « A Role for Aromatic Amino Acids in the Binding of Xenopus Ribosomal Protein L5 to 5S rRNA », Biochemistry, vol. 40, no 42, , p. 12645-12653 (PMID 11601989, DOI 10.1021/bi011439m, lire en ligne)
- (en) Feng-Jie Sun et Gustavo Caetano-Anollés, « The Evolutionary History of the Structure of 5S Ribosomal RNA », Journal of Molecular Evolution, vol. 69, no 5, , p. 430-443 (PMID 19639237, DOI 10.1007/s00239-009-9264-z, lire en ligne)
- (en) G. M. Gongadze, « 5S rRNA and ribosome », Biochemistry (Moscow), vol. 76, no 13, , p. 1450-1464 (PMID 22339598, DOI 10.1134/S0006297911130062, lire en ligne)
- (en) David Ammons, Joanne Rampersad et George E. Fox, « 5S rRNA gene deletions cause an unexpectedly high fitness loss in Escherichia coli », Nucleic Acids Research, vol. 27, no 2, , p. 637-642 (PMID 9862991, DOI 10.1093/nar/27.2.637, lire en ligne)
- (en) Steven J. McBryant, Nik Veldhoen, Ben Gedulin, Anne Leresche, Mark P. Foster, Peter E. Wright, Paul J. Romaniuk et Joel M. Gottesfeld, « Interaction of the RNA binding Fingers of Xenopus Transcription Factor IIIA with Specific Regions of 5 S Ribosomal RNA », Journal of Molecular Biology, vol. 248, no 1, , p. 44-57 (PMID 7731045, DOI 10.1006/jmbi.1995.0201, lire en ligne)
- (en) M. A. Searles, D. Lu et A Klug, « The role of the central zinc fingers of transcription factor IIIA in binding to 5 S RNA », Journal of Molecular Biology, vol. 301, no 1, , p. 47-60 (PMID 10926492, DOI 10.1006/jmbi.2000.3946, lire en ligne)
- (en) H. R. Pelham et D. D. Brown, « A specific transcription factor that can bind either the 5S RNA gene or 5S RNA », Proceedings of the National Academy of Sciences of the United States of America, vol. 77, no 7, , p. 4170-4174 (PMID 7001457, PMCID 349792, DOI 10.1073/pnas.77.7.4170, lire en ligne)
- (en) A. K. Henras, J. Soudet, M. Gérus, S. Lebaron, M. Caizergues-Ferrer, A. Mougin et Y. Henry, « The post-transcriptional steps of eukaryotic ribosome biogenesis », Cellular and Molecular Life Sciences, vol. 65, no 15, , p. 2334-2359 (PMID 18408888, DOI 10.1007/s00018-008-8027-0, lire en ligne)
- (en) Sandra L. Wolin et Tommy Cedervall, « The La protein », Annual Review of Biochemistry, vol. 71, , p. 375-403 (PMID 12045101, DOI 10.1146/annurev.biochem.71.090501.150003, lire en ligne)
- (en) R. J. Maraia et R. V Intine, « La Protein and its Associated Small Nuclear and Nucleolar Precursor RNAs », Gene Expression, vol. 10, nos 1-2, , p. 41-57 (PMID 11868987, DOI 10.0000/096020197390068, lire en ligne)
- (en) Peter B. Moore, « The Ribosome at Atomic Resolution », Biochemistry, vol. 40, no 11, , p. 3243-3250 (PMID 11258942, DOI 10.1021/bi0029402, lire en ligne)
