ADN fossile

Le terme ADN fossile fait référence à l'ADN provenant d'un échantillon très ancien, comme les fossiles. L'étude de l'ADN fossile est utilisée en paléogénétique et en génétique des populations. En 2016, les restes d'ADN humains les plus anciens qui ont pu être récupérés et analysés appartiennent aux prénéandertaliens de la Sima de los Huesos, un aven de la sierra d'Atapuerca en Espagne[1],[2], et sont âgés de 430 000 ans[3]. L'ADN d'un fossile d'un cheval de 700 000 ans a également pu être analysé[4]. Bien que dans les années 1990, certains scientifiques aient pu croire avoir réussi à construire des séquences d'ADN d'échantillons vieux de plusieurs millions d'années (notamment celui d'un coléoptère[5], voire, en 1994, celui d'un dinosaure[6], qui se révéla en fait être de l'ADN humain[7]) grâce à la technique PCR, on s'accorde aujourd'hui à dire qu'ils avaient en fait été contaminés par l'intervention humaine, et que l'ADN ne parvient pas à résister à une telle échelle de temps [8],[9].
De la technique PCR au next generation sequencing (NGS) et portée des découvertes
[modifier | modifier le code]La possibilité d'extraire les restes d'ADN de cellules issues fossiles, et de les amplifier par PCR (Réaction en chaîne par polymérase) afin d'obtenir une séquence d'ADN suffisamment grande, est récente. Dès la fin des années 1980, cette technique a été utilisée: les premiers restes d'ADN, respectivement de quagga[10] (une sorte de zèbre éteint au XIXe siècle) et d'une momie égyptienne[11], ont été analysés en 1984[5]. Cependant, on s'est rendu compte que l'utilisation de l'analyse PCR posait des problèmes de contamination, l'ADN fossile étant très souvent contaminé par l'intervention humaine[12]. Des annonces spectaculaires, et attirant de façon substantielle l'attention du public (notamment après la parution de Jurassic Park), faites au cours des années 1990, ont ainsi été rétractées par la suite[12].
Désormais, plutôt que la PCR, on utilise des techniques dites de next generation sequencing (NGS), lesquelles permettent d'extraire de courtes séquences d'ADN, moins susceptibles de contamination[12]. Emergeant vers 2010[13], cette technique a été utilisée sur des fossiles de Néandertal et de mammouth. Elles ont permis notamment de mettre en évidence une petite portion d'ADN commun entre homo sapiens et Néandertal, accréditant la thèse d'existence de relations sexuelles entre ces deux espèces (ce qui fut d'abord montré par l'équipe de Svante Pääbo, de l'Institut Max-Planck d'anthropologie évolutionniste, avant d'être confirmé par d'autres études)[13]. En 2010, toujours grâce à cette technique, l'équipe de Pääbo identifia une nouvelle espèce, l'Hominidé de Denisova, déclarant qu'elle s'était mêlée à Homo sapiens[13].
Quelques années après, il y eut aussi une multitude de tels exemples chez les plantes[14] et même bactéries[15]. Ainsi, Golenberg et son équipe ont obtenu une séquence partielle d'ADN de chloroplaste appartenant à un fossile de Magnolia[16]. Selon le Web of Science, le nombre d'articles ayant « ADN fossile » dans leur titre est ainsi passé de 30 en 1995 à 275 en 2014[12]. Quatorze articles en particulier, datant en moyenne de 2013 et portant sur des fossiles humains, sont cités de façon récurrente[12].
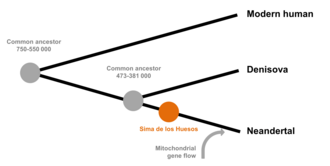
Cependant, la controverse sur la fiabilité des procédures utilisées a persisté[17]. L'ADN fossile permettrait d'établir des relations phylogenétiques entre divers taxons, et en outre de faciliter une vision globale des diverses branches évolutives. De plus, il facilite l'estimation du taux de mutation existant entre taxons liés[16],[18]. Ainsi en les équipes de Svante Pääbo ont pu proposer une hypothèse de reconstruction de l'arbre phylogénétique probable entre les lignées humaines récentes dont l'ADN est déjà connu : homme de Néandertal, l'hominidé de Denisova et l'homme moderne[1],[2].
Méthodes
[modifier | modifier le code]Les méthodes proposés sont:

- Extraction d'ambre: Cette suggestion, selon un principe non-viable et fictif, a été nourrie dans la fantaisie populaire à travers le roman de fiction (et a posteriori film) Jurassic Park. Dans ce livre, il a été suggéré que des insectes suceurs (tels que le moustique) piégés dans de l'ambre pouvaient parfaitement préserver l'ADN d'autres animaux, comme des dinosaures. Actuellement, en dépit d'affirmations antérieures[19], la possibilité d'extraire l'ADN d'insectes conservés dans de l'ambre est sujet à débat[20],[21].
- Extraction d'os partiellement cristallisés: Il fut noté que certains os fossilisés présentent parfois des structures contenant des agrégats cristallins. Des scientifiques ont démontré que l'ADN contenu dans ces agrégats de cristaux se maintenait dans un relativement bon état et dont un traitement à l'hypochlorite de sodium (NaClO) a permis à ces chercheurs d'obtenir des fragments d'ADN plus grands et mieux préservés[22].
Rubriques connexes
[modifier | modifier le code]Notes et références
[modifier | modifier le code]- Meyer et al. 2016.
- Meyer et al. 2013.
- Arsuaga et al. 2014.
- Orland et al. 2013.
- "L'ADN ancien", dans La science au présent, 1997, Encyclopaedia Universalis éditeur, sur le site du CNRS.
- Woodward et al. 1994.
- Zischler et al. 1995.
- Hoelzel 2015, review de Shapiro 2015
- Willerslev 2003.
- Higuchi et al. 1984.
- Pääbo 1984.
- Culotta 2015.
- Gibbons 2015.
- Soltis 1993.
- Coolen 1998.
- Golenberg 1990.
- Hebsgaard et al. 2005.
- Xue et al. 2009.
- DeSalle et al. 1992.
- Austin et al. 1997.
- Penney et al. 2013.
- Salamon et al. 2005.
Bibliographie
[modifier | modifier le code]Articles scientifiques
[modifier | modifier le code]- (en) Juan Luis Arsuaga, Martínez, L. J. Arnold, A. Aranburu, A. Gracia-Téllez, W. D. Sharp, R. M. Quam, C. Falguères, A. Pantoja-Pérez, J. Bischoff, E. Poza-Rey, J. M. Parés, J. M. Carretero, M. Demuro, C. Lorenzo, N. Sala, M. Martinón-Torres, N. García, A. Alcázar de Velasco, G. Cuenca-Bescós, A. Gómez-Olivencia, D. Moreno, A. Pablos, C.-C. Shen, L. Rodríguez, A. I. Ortega, R. García, A. Bonmatí, J. M. Bermúdez de Castro et Eudald Carbonell, « Neandertal roots: Cranial and chronological evidence from Sima de los Huesos », Science, 6190e série, vol. 344, , p. 1358-1363 (DOI doi:10.1126/science.1253958)
- (en) J. J. Austin, A. J. Ross, A. B. SMITH, R. A. Fortey et R. H. Thomas, « Problems of reproducibility - does geologically ancient DNA survive in amber-preserved insects? », The Royal Society, vol. 264, no 1381, , p. 467–474 (ISSN 0962-8452, DOI 10.1098/rspb.1997.0067)
- (en) MJ Coolen et J Overmann, « Analysis of Subfossil Molecular Remains of Purple Sulfur Bacteria in a Lake Sediment », Applied and Environmental Microbiology, vol. 64, no 11, , p. 4513–4521 (PMID 9797316)
- (en) Elizabeth Culotta, « New life for old bones », American Association for the Advancement of Science (AAAS), vol. 349, no 6246, , p. 358–361 (ISSN 0036-8075, DOI 10.1126/science.349.6246.358)
- (en) Rob DeSalle, John Gatesy, Ward Wheeler et David Grimaldi, « DNA sequences from a fossil termite in Oligo-Miocene amber and their phylogenetic implications », American Association for the Advancement of Science (AAAS), vol. 257, no 5078, , p. 1933–1936 (DOI 10.1126/science.1411508)
- (en) Edward M. Golenberg, David E. Giannasi, Michael T. Clegg, Charles J. Smiley, Mary Durbin, David Henderson et Gerard Zurawski, « Chloroplast DNA sequence from a Miocene Magnolia species », Springer Nature, vol. 344, no 6267, , p. 656–658 (DOI 10.1038/344656a0)
- (en) Anne Gibbons, « Revolution in human evolution », American Association for the Advancement of Science (AAAS), vol. 349, no 6246, , p. 362–366 (DOI 10.1126/science.349.6246.362)
- (en) Martin B. Hebsgaard, Matthew J. Phillips et Eske Willerslev, « Geologically ancient DNA: fact or artefact? », Elsevier BV, vol. 13, no 5, , p. 212–220 (DOI 10.1016/j.tim.2005.03.010)
- (en) Russell Higuchi, Barbara Bowman, Mary Freiberger, Oliver A. Ryder et Allan C. Wilson, « DNA sequences from the quagga, an extinct member of the horse family », Springer Nature, vol. 312, no 5991, , p. 282–284 (ISSN 0028-0836, DOI 10.1038/312282a0)
- (en) A. R. Hoelzel, « Raising the dead », American Association for the Advancement of Science (AAAS), vol. 349, no 6246, , p. 388–388 (ISSN 0036-8075, DOI 10.1126/science.aaa9849)
- (en) Matthias Meyer, Qiaomei Fu, Ayinuer Aximu-Petri, Isabelle Glocke, Birgit Nickel, Juan-Luis Arsuaga, Ignacio Martínez, Ana Gracia, José María Bermúdez de Castro, Eudald Carbonell et Svante Pääbo, « A mitochondrial genome sequence of a hominin from Sima de los Huesos », Nature, vol. 505, , p. 403–406 (DOI doi:10.1038/nature12788, lire en ligne)
- (en) Matthias Meyer, Juan Luis Arsuaga, Cesare de Filippo, Sarah Nagel, Ayinuer Aximu-Petri, Birgit Nickel, Ignacio Martínez Ana Gracia, José María Bermúdez de Castro, Eudald Carbonell, Bence Viola, Janet Kels, Kay Prüfer et Svante Pääbo, « Nuclear DNA sequences from the Middle Pleistocene Sima de los Huesos hominins », Nature, 7595e série, vol. 531, , p. 504-507 (DOI 10.1038/nature17405)
- (en) Ludovic Orlando, Aurélien Ginolhac, Guojie Zhang, Duane Froese, Anders Albrechtsen, Mathias Stiller, Mikkel Schubert, Enrico Cappellini, Bent Petersen, Ida Moltke, Philip L. F. Johnson, Matteo Fumagalli, Julia T. Vilstrup, Maanasa Raghavan, Thorfinn Korneliussen, Anna-Sapfo Malaspinas, Josef Vogt, Damian Szklarczyk, Christian D. Kelstrup, Jakob Vinther, Andrei Dolocan, Jesper Stenderup, Amhed M. V. Velazquez, James Cahill, Morten Rasmussen, Xiaoli Wang, Jiumeng Min, Grant D. Zazula, Andaine Seguin-Orlando, Cecilie Mortensen, Kim Magnussen, John F. Thompson, Jacobo Weinstock, Kristian Gregersen, Knut H. Røed, Véra Eisenmann, Carl J. Rubin, Donald C. Miller, Douglas F. Antczak, Mads F. Bertelsen, Søren Brunak, Khaled A. S. Al-Rasheid, Oliver Ryder, Leif Andersson, John Mundy, Anders Krogh, M. Thomas P. Gilbert, Kurt Kjær, Thomas Sicheritz-Ponten, Lars Juhl Jensen, Jesper V. Olsen, Michael Hofreiter, Rasmus Nielsen, Beth Shapiro, Jun Wang et Eske Willerslev, « Recalibrating Equus evolution using the genome sequence of an early Middle Pleistocene horse », Springer Nature, vol. 499, no 7456, , p. 74–78 (ISSN 0028-0836, DOI 10.1038/nature12323)
- (en) David Penney, Caroline Wadsworth, Graeme Fox, Sandra L. Kennedy, Richard F. Preziosi, Terence A. Brown et Ludovic Orlando (dir.), « Absence of Ancient DNA in Sub-Fossil Insect Inclusions Preserved in ‘Anthropocene’ Colombian Copal », Public Library of Science (PLoS), vol. 8, no 9, , e73150 (ISSN 1932-6203, DOI 10.1371/journal.pone.0073150)
- (en) M. Salamon, N. Tuross, B. Arensburg et S. Weiner, « Relatively well preserved DNA is present in the crystal aggregates of fossil bones », Proceedings of the National Academy of Sciences, vol. 102, no 39, , p. 13783–13788 (ISSN 0027-8424, DOI 10.1073/pnas.0503718102)
- (en) Pamela S. Soltis et Douglas E. Soltis, « Ancient DNA: Prospects and limitations », Informa UK Limite, vol. 31, no 3, , p. 203–209 (DOI 10.1080/0028825x.1993.10419497)
- (de) Svante Pääbo, « Über den Nachweis von DNA in altägyptischen Mumien », Das Altertum, vol. 30, , p. 2013-218 (lire en ligne, consulté le ).
- (en) Svante Pääbo, « Preservation of DNA in ancient Egyptian mummies », Journal of Archaeological Science, vol. 12, no 6, , p. 411-417 (DOI 10.1016/0305-4403(85)90002-0)
- (en) Svante Pääbo, « Molecular cloning of ancient Egyptian mummy DNA », Nature, vol. 314, no 6012, , p. 644-645 (DOI 10.1038/314644a0)
- (en) E. Willerslev, « Diverse Plant and Animal Genetic Records from Holocene and Pleistocene Sediments », American Association for the Advancement of Science (AAAS), vol. 300, no 5620, , p. 791–795 (ISSN 0036-8075, DOI 10.1126/science.1084114)
- (en) E. Willerslev et A. Cooper, « Review Paper. Ancient DNA », The Royal Society, vol. 272, no 1558, , p. 3–16 (DOI 10.1098/rspb.2004.2813)
- (en) Woodward, NJ Weyand et M Bunnell, « DNA sequence from Cretaceous period bone fragments », Science, vol. 266, no 5188, , p. 1229–1232 (ISSN 0036-8075, PMID 7973705, DOI 10.1126/science.7973705)
- (en) Yali Xue, Qiuju Wang, Quan Long, Bee Ling Ng, Harold Swerdlow, John Burton, Carl Skuce, Ruth Taylor, Zahra Abdellah, Yali Zhao, Daniel G. MacArthur, Michael A. Quail, Nigel P. Carter, Huanming Yang et Chris Tyler-Smith, « Human Y Chromosome Base-Substitution Mutation Rate Measured by Direct Sequencing in a Deep-Rooting Pedigree », Elsevier BV, vol. 19, no 17, , p. 1453–1457 (ISSN 0960-9822, DOI 10.1016/j.cub.2009.07.032)
- (en) H Zischler, M Hoss, O Handt, A von Haeseler, A. van der Kuyl et J Goudsmit, « Detecting dinosaur DNA », American Association for the Advancement of Science (AAAS), vol. 268, no 5214, , p. 1192–1193 (ISSN 0036-8075, DOI 10.1126/science.7605504)
Ouvrages de vulgarisation
[modifier | modifier le code]- Svante Pääbo (trad. de l'anglais), Néandertal : À la recherche des génomes perdus, Paris, Les liens qui libèrent, , 394 p. (ISBN 979-10-209-0321-1)
- (en) David Reich, Who We Are And How We Got Here : Ancient DNA and the New Science of the Human Past, Pantheon Books, , 368 p. (ISBN 978-1-101-87032-7)
- (en) Beth Shapiro, How to clone a mammoth : the science of de-extinction, Princeton, Princeton University Press, , 220 p. (ISBN 978-0-691-15705-4, OCLC 891123548)
