Domaine bZIP
| Pfam | PF00170 |
|---|---|
| InterPro | IPR011616 |
| PROSITE | PDOC00036 |
| SCOP | 1ysa |
| SUPERFAMILY | 1ysa |
| CDD | cd14686 |
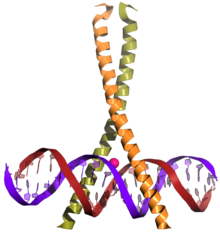
Un domaine bZIP — de l'anglais basic leucine zipper — est un type de domaine de liaison à l'ADN présent dans nombreuses protéines se liant à l'ADN chez les eucaryotes. La région N-terminale de ce domaine contient une région, riche en résidus d'acides aminés basiques tels que la lysine et l'arginine, permettant la liaison de la molécule à une séquence nucléotidique spécifique sur l'ADN, tandis que la région C-terminale en hélice α contient la glissière à leucine permettant d'assurer la dimérisation de l'ensemble. Les domaines bZIP se trouvent en premier lieu sur les facteurs de transcription[2],[3].
Les facteurs de transcription à domaine bZIP sont présents chez tous les êtres vivants. Une étude de 2008 indique que quatre gènes de domaines bZIP pourraient avoir été portés par le dernier ancêtre commun à toutes les plantes[4]. Les interactions entre facteurs de transcription à domaine bZIP jouent un rôle important dans le développement de certains cancers des épithéliums[5], dans la biosynthèse des hormones stéroïdiennes par les cellules des glandes endocrines[6], dans les fonctions reproductives[7] et dans plusieurs autres phénomènes physiologiques.
On trouve des domaines bZIP notamment dans les protéines suivantes :
- le facteur de transcription AP-1 constitué de l'hétérodimère c-Fos/c-Jun (en) ;
- le facteur de transcription jun-B (en) ;
- la protéine CREB, qui agit comme un facteur de transcription liant l'AMP cyclique ;
- le facteur de transcription opaque-2 des gènes de la zéine 22-kD qui encodent une classe de protéines de stockage de l'albumen de maïs ;
- le facteur de transcription NFE2L2 (pour Nuclear Factor (Erythroid-derived 2)-Like 2), ou Nrf2 ;
- les facteurs de transcription Maf.
Notes et références
[modifier | modifier le code]- (en) Maria A. Schumacher, Richard H. Goodman et Richard G. Brennan, « The Structure of a CREB bZIP·Somatostatin CRE Complex Reveals the Basis for Selective Dimerization and Divalent Cation-enhanced DNA Binding », Journal of Biological Chemistry, vol. 274, no 45, , p. 35242-35247 (PMID 10952992, DOI 10.1074/jbc.M007293200, lire en ligne)
- (en) Tom Ellenberger, « Getting a grip on DNA recognition: structures of the basic region leucine zipper, and the basic region helix-loop-helix DNA-binding domains », Current Opinion in Structural Biology, vol. 4, no 1, , p. 12-21 (DOI 10.1016/S0959-440X(94)90054-X, lire en ligne)
- (en) H. C. Hurst, « Transcription factors 1: bZIP proteins », Protein Profile, vol. 2, no 2, , p. 101-168 (PMID 7780801)
- (en) Luiz Gustavo Guedes Corrêa, Diego Mauricio Riaño-Pachón, Carlos Guerra Schrago, Renato Vicentini dos Santos, Bernd Mueller-Roeber et Michel Vincentz, « The Role of bZIP Transcription Factors in Green Plant Evolution: Adaptive Features Emerging from Four Founder Genes », PLoS One, vol. 3, no 8, , e2944 (PMID 18698409, PMCID 2492810, DOI 10.1371/journal.pone.0002944, lire en ligne)
- (en) Spiros A. Vlahopoulos, Stella Logotheti, Dimitris Mikas, Athina Giarika, Vassilis Gorgoulis et Vassilis Zoumpourlis, « The role of ATF-2 in oncogenesis », BioEssays, vol. 30, no 4, , p. 314-327 (PMID 18348191, DOI 10.1002/bies.20734, lire en ligne)
- (en) Pulak R. Manna, Matthew T. Dyson, Darrell W. Eubank, Barbara J. Clark, Enzo Lalli, Paolo Sassone-Corsi, Anthony J. Zeleznik et Douglas M. Stocco, « Regulation of Steroidogenesis and the Steroidogenic Acute Regulatory Protein by a Member of the cAMP Response-Element Binding Protein Family », Molecular Endocrinology, vol. 16, no 1, , p. 184-199 (DOI 10.1210/me.16.1.184, lire en ligne)
- (en) Sarasija Hoare, John A. Copland, Thomas G. Wood, Yow-Jiun Jeng, Michael G. Izban et Melvyn S. Soloff, « Identification of a GABPα/β Binding Site Involved in the Induction of Oxytocin Receptor Gene Expression in Human Breast Cells. Potentiation by c-Fos/c-Jun », Endocrinology, vol. 140, no 5, , p. 2268-2279 (PMID 10218980, DOI 10.1210/endo.140.5.6710, lire en ligne)