« Connectomique » : différence entre les versions
[bot] Mise à jour du modèle {Une pomme est un fruit} (v2.7.7) |
traduction depuis l'anglais |
||
| Ligne 1 : | Ligne 1 : | ||
{{Une pomme est un fruit|année création=2012|année présentation bistro=2021}} |
{{Une pomme est un fruit|année création=2012|année présentation bistro=2021}} |
||
[[Image:Budapest Reference Connectome.png|vignette|Image du [[:en:Budapest Reference Connectome|Budapest Reference Connectome]]]] |
|||
| ⚫ | |||
La connectomique est la production et l'étude des connectomes : des cartes complètes des connexions au sein du [[système nerveux]] d'un organisme. Plus généralement, on peut considérer qu'il s'agit de l'étude des schémas de câblage neuronaux, en mettant l'accent sur la façon dont la connectivité structurelle, les [[synapse]]s individuelles, la morphologie et l'ultrastructure cellulaires contribuent à la constitution d'un réseau. Le système nerveux est un réseau constitué de milliards de connexions et ces connexions sont responsables de nos pensées, émotions, actions, souvenirs, fonctions et dysfonctionnements. Par conséquent, l'étude de la connectomique vise à faire progresser notre compréhension de la santé mentale et de la [[cognition]] en comprenant comment les cellules du système nerveux sont connectées et communiquent. Ces structures étant extrêmement complexes, les méthodes de ce domaine utilisent une application de [[criblage à haut débit]] d'imagerie neuronale fonctionnelle et structurelle, le plus souvent l'[[imagerie par résonance magnétique]] (IRM), la [[microscopie électronique]] et les techniques [[histologie|histologiques]], afin d'augmenter la vitesse, l'efficacité et la résolution de ces cartes du système nerveux. À ce jour, des dizaines d'ensembles de données à grande échelle ont été collectés, couvrant le système nerveux, y compris les différentes zones du [[cortex]], le [[cervelet]]<ref>{{Cite journal|nom1=Quartarone|prénom1=Angelo|nom2=Cacciola|prénom2=Alberto|nom3=Milardi|prénom3=Demetrio|nom4=Ghilardi|prénom4=Maria Felice|nom5=Calamuneri|prénom5=Alessandro|nom6=Chillemi|prénom6=Gaetana|nom7=Anastasi|prénom7=Giuseppe|nom8=Rothwell|prénom8=John|date=01/02/2020|langue=en|titre=New insights into cortico-basal-cerebellar connectome: clinical and physiological considerations|journal=Brain|volume=143|issue=2|pages=396–406|doi=10.1093/brain/awz310|pmid=31628799|issn=0006-8950|doi-access=free}}</ref>{{,}}<ref>{{Cite journal|nom1=Nguyen|prénom1=Tri M.|nom2=Thomas|prénom2=Logan A.|nom3=Rhoades|prénom3=Jeff L.|nom4=Ricchi|prénom4=Ilaria|nom5=Yuan|prénom5=Xintong Cindy|nom6=Sheridan|prénom6=Arlo|nom7=Hildebrand|prénom7=David G. C.|nom8=Funke|prénom8=Jan|nom9=Regehr|prénom9=Wade G.|nom10=Lee|prénom10=Wei-Chung Allen|date=30/11/2021|langue=en|titre=Structured connectivity in the cerebellum enables noise-resilient pattern separation|périodique=bioRxiv-10.1101/2021.11.29.470455v1|pages=2021.11.29.470455}}.</ref>, la [[rétine]]<ref>{{Cite journal|nom1=Helmstaedter|prénom1=Moritz|nom2=Briggman|prénom2=Kevin L.|prénom3=Turaga|nom3=Srinivas C.|prénom4=Jain|nom4=Viren|prénom5=Seung|nom5=H. Sebastian|prénom6=Denk|nom6=Winfried|date=07/08/2013|titre=Connectomic reconstruction of the inner plexiform layer in the mouse retina|périodique=Nature|volume=500|issue=7461|pages=168–174|doi=10.1038/nature12346|pmid=23925239|bibcode=2013Natur.500..168H |s2cid=3119909|issn=0028-0836}}.</ref>, le [[système nerveux périphérique]]<ref>{{Cite journal|nom1=Phelps|prénom1=Jasper S.|nom2=Hildebrand|prénom2=David Grant Colburn|nom3=Graham|prénom3=Brett J.|nomt4=Kuan|prénom4=Aaron T.|nom5=Thomas|prénom5=Logan A.|nom6=Nguyen|prénom6=Tri M.|nom7=Buhmann|prénom7=Julia|nom8=Azevedo|prénom8=Anthony W.|nom9=Sustar|prénom9=Anne|nom10=Agrawal|prénom10=Sweta|nom11=Liu|prénom11=Mingguan|date=04/02/2021|titre=Reconstruction of motor control circuits in adult Drosophila using automated transmission electron microscopy|journal=Cell|langue=en|volume=184|issue=3|pages=759–774.e18|doi=10.1016/j.cell.2020.12.013|pmid=33400916|pmc=8312698|issn=0092-8674}}.</ref> et les [[jonction neuromusculaire|jonctions neuromusculaires]]<ref name="pmid26634293">{{cite journal|nom1=Boonstra|prénom1=Tjeerd W.|prénom2=Danna-Dos-Santos|nom2=Alessander|prénom3=Hong-Bo|nom3=Xie.|prénom4=Roerdink|nom4=Melvyn|prénom5=Stins|nom5=John F.|=Michael Breakspear|nom6=Breakspear|prénom6=Michael|titre=Muscle networks: Connectivity analysis of EMG activity during postural control|périodique=Scientific Reports|volume=5|pages=17830|year=2015|pmid=26634293|doi=10.1038/srep17830|pmc=4669476|bibcode=2015NatSR...517830B }}.</ref>. |
|||
{{Ébauche|neurosciences}} |
|||
D'une manière générale, il existe deux types de connectomes : les connectomes à macro-échelle et les connectomes à micro-échelle. La connectomique à macro-échelle consiste à utiliser les données de l'IRM [[Imagerie par résonance magnétique fonctionnelle|fonctionnelle]] et [[IRM de diffusion|structurelle]] pour cartographier les grands trajets de fibres et les zones de matière grise fonctionnelle dans le cerveau en termes de flux sanguin (fonctionnel) et de diffusivité de l'eau (structurel). La connectomique à micro-échelle est la cartographie du connectome complet des petits organismes à l'aide de la microscopie et de l'histologie ; on considère donc là toutes les connexions qui existent dans leur système nerveux central. |
|||
| ⚫ | |||
== Méthodes == |
|||
[[Image:Connectomics.png|vignette|326x326px|L'imagerie par résonance magnétique est utilisée pour évaluer la connectomique à macro-échelle dans le cerveau humain. Les séries d'images d'[[IRM de diffusion|IRMd]] sont utilisées pour cartographier les trajets de la matière blanche, et les séries d'[[Imagerie par résonance magnétique fonctionnelle|IRMf]] sont utilisées pour évaluer la corrélation entre le flux sanguin et les zones de matière grise connectées..]] |
|||
=== Connectomique à macro-échelle === |
|||
Les connectomes à macro-échelle sont généralement collectés à l'aide de l'[[IRM de diffusion|imagerie par résonance magnétique de diffusion]] (IRMd) et de l'[[Imagerie par résonance magnétique fonctionnelle|imagerie par résonance magnétique fonctionnelle]] (IRMf). Les ensembles de données d'IRMd peuvent couvrir l'ensemble du cerveau, en imagerie de la substance blanche entre le [[cortex]] et le sous-cortex. En revanche, les ensembles de données d'IRMf mesurent le débit sanguin cérébral, en tant que marqueur de l'activation neuronale. L'un des avantages de l'IRM est qu'elle fournit des informations ''in vivo'' sur la connectivité entre les différentes zones du cerveau. La connectomique à macro-échelle a permis de mieux comprendre divers réseaux cérébraux, notamment les réseaux visuels<ref>{{cite journal|nom1=Kammen|prénom1=Alexandra|nom2=Law|prénom2=Meng|nom3=Tjan|prénom3=Bosco S.|nom4=Toga|prénom4=Arthur W.|nom5=Shi|prénom5=Yonggang|date=Janvier 2016|langue=en|titre=Automated retinofugal visual pathway reconstruction with multi-shell HARDI and FOD-based analysis|périodique=NeuroImage|volume=125|pages=767–779|doi=10.1016/j.neuroimage.2015.11.005|pmid=26551261|pmc=4691391|issn=1053-8119}}.</ref>{{,}}<ref>{{Cite journal|nom1=Yogarajah|prénom1=M.|nom2=Focke|prénom2=N. K.|nom3=Bonelli|prénom3=S.|nom4=Cercignani|prénom4=M.|nom5=Acheson|prénom5=J.|nom6=Parker|prénom6=G. J. M.|nom7=Alexander|prénom7=D. C.|nom8=McEvoy|prénom8=A. W.|nom9=Symms|prénom9=M. R.|nom10=Koepp|prénom10=M. J.|nom11=Duncan|prénom11=J. S.|date=21/05/2009|langue=en|titre=Defining Meyer's loop-temporal lobe resections, visual field deficits and diffusion tensor tractography|périodique=Brain|volume=132|issue=6|pages=1656–1668|doi=10.1093/brain/awp114|pmid=19460796|issn=0006-8950|pmc=2685925}}.</ref>, du [[tronc cérébral]]<ref>{{Ouvrage |nom1=Nieuwenhuys|prénom1=Rudolf|nom2=Voogd|prénom2=Jan|nom3=van Huijzen|prénom3=Christiaan|date=2008|langue=en|titre=The Human Central Nervous System|doi=10.1007/978-3-540-34686-9|isbn=978-3-540-34684-5}}</ref>{{,}}<ref>{{Chapitre|nom1=Paxinos|prénom1=George|langue=en|titre chapitre=Organization of Brainstem Nuclei|date=2012|url=http://dx.doi.org/10.1016/b978-0-12-374236-0.10008-2|titre ouvrage=The Human Nervous System|pages=260–327|éditeur=Elsevier|consulté le=07/12/2021|nom2=Xu-Feng|prénom2=Huang|nom3=Sengul|prénom3=Gulgun|nom4=Watson|prénom4=Charles|doi=10.1016/b978-0-12-374236-0.10008-2|isbn=9780123742360}}.</ref>, et du langage<ref>{{Cite journal|nom1=Glasser|prénom1=Matthew F.|nom2=Rilling|prénom2=James K.|date=14/02/2008|langue=en|titre=DTI Tractography of the Human Brain's Language Pathways|périodique=Cerebral Cortex|volume=18|issue=11|pages=2471–2482|doi=10.1093/cercor/bhn011|pmid=18281301|issn=1460-2199}}.</ref>{{,}}<ref>{{Cite journal|nom1=Catani|prénom1=Marco|nom2=Jones|prénom2=Derek K.|nom3=ffytche|prénom3=Dominic H.|date=2004|langue=en|titre=Perisylvian language networks of the human brain|périodique=Annals of Neurology|volume=57|issue=1|pages=8–16|doi=10.1002/ana.20319|pmid=15597383|s2cid=17743067|issn=0364-5134|doi-access=free}}.</ref>. |
|||
=== Connectomique à micro-échelle === |
|||
D'autre part, les connectomes à micro-échelle se concentrent sur une zone beaucoup plus petite du système nerveux avec une résolution beaucoup plus élevée. Ces ensembles de données sont généralement collectés à l'aide de l'imagerie par microscopie électronique et offrent une résolution synaptique unique de circuits locaux entiers. Parmi les jalons de la connectomique à micro-échelle, citons le système nerveux entier du nématode ''[[Caenorhabditis elegans]]''<ref>{{Cite journal|date=12/11/1986|langue=en|titre=The structure of the nervous system of the nematode Caenorhabditis elegans|périodique=Philosophical Transactions of the Royal Society of London. B, Biological Sciences|volume=314|issue=1165|pages=1–340|doi=10.1098/rstb.1986.0056|pmid=22462104|issn=0080-4622|nom1=White|prénom1=J. G.|nom2=Southgate|prénom2=E.|nom3=Thomson|prénom3=J. N.|nom4=Brenner|prénom4=S.|bibcode=1986RSPTB.314....1W }}</ref>, un cerveau entier de mouche<ref>{{Cite journal|nom1=Scheffer|prénom1=Louis K|nom2=Xu|prénom2=C Shan|nom3=Januszewski|prénom3=Michal|nom4=Lu|prénom4=Zhiyuan|nom5=Takemura|prénom5=Shin-ya|nom6=Hayworth|prénom6=Kenneth J|nom7=Huang|prénom7=Gary B|nom8=Shinomiya|prénom8=Kazunori|nom9=Maitlin-Shepard|prénom9=Jeremy|nom10=Berg|prénom10=Stuart|nom11=Clements|prénom11=Jody|date=03/09/2020| nom12=Marder|prénom12=Eve|directeur12=oui|nom13=Eisen|prénom13=Michael B|directeur13=oui|nom14=Pipkin|prénom14=Jason|directeur14=oui|nom15=Doe|prénom15=Chris Q|directeur15=oui|langue=en|titre=A connectome and analysis of the adult Drosophila central brain|périodique=eLife|volume=9|pages=e57443|doi=10.7554/eLife.57443|issn=2050-084X|pmid=32880371|pmc=7546738}}.</ref> et, plus récemment, un cube millimétrique des cortex de la souris<ref>{{Cite journal|collaboration=MICrONS Consortium |nom1=Bae|prénom1=J. Alexander|nom2=Baptiste|prénom2=Mahaly|nom3=Bodor|prénom3=Agnes L.|nom4=Brittain|prénom4=Derrick|nom5=Buchanan|prénom5=JoAnn |nom6=Bumbarger|prénom6=Daniel J.|nom7=Castro|prénom7=Manuel A.|nom8=Celii|prénom8=Brendan|nom9=Cobos|prénom9=Erick|nom10=Collman|prénom10=Forrest |date=29/07/2021|langue=en|titre=Functional connectomics spanning multiple areas of mouse visual cortex|périodique=biorxiv=10.1101/2021.07.28.454025|language=en}}.</ref> et de l'homme<ref>{{Cite journal|nom1=Shapson-Coe|ptrénom1=Alexander|nom2=Januszewski|prénom2=Michał|nom3=Berger|prénom3=Daniel R.|nom4=Pope|prénom4=Art|nom5=Wu|prénom5=Yuelong|nom6=Blakely|prénom6=Tim|nom7=Schalek|prénom7=Richard L.|nom8=Li|prénom8=Peter|nom9=Wang|prénom9=Shuohong |nom10=Maitin-Shepard|prénom10=Jeremy|nom11=Karlupia|prénom11=Neha|date=30/05/2021|titre=A connectomic study of a petascale fragment of human cerebral cortex |périodique=bioRxiv-10.1101/2021.05.29.446289|langue=en}}.</ref>. |
|||
== Outils == |
|||
L'IRM est l'un des principaux outils utilisés pour la recherche en connectomique à l'échelle macroscopique<ref name=pmid18495497>{{cite journal|nom1=Wedeen|prénom1=V.J.|nom2=Wang|prénom2=R.P.|nom3=Schmahmann|prénom3=J.D.|nomt4=Benner|prénom4=T.|nom5=Tseng|prénom5=W.Y.I.|nom6=Dai|prénom6=G.|nom7=Pandya|prénom7=D.N.|nom8=Hagmann|prénom8=P.|nom9=D'arceuil|prénom9=H.|nom10=De Crespigny|prénom10=A.J.|langue=en|titre=Diffusion spectrum magnetic resonance imaging (DSI) tractography of crossing fibers|périodique=NeuroImage|volume=41|issue=4|pages=1267–77|année=2008|pmid=18495497|doi=10.1016/j.neuroimage.2008.03.036|s2cid=2660208|display-authors=8 }}.</ref>. Lorsqu'elles sont utilisées ensemble, un ensemble de données d'IRMf et d'IRMd à l'état de repos fournit une vue complète de la manière dont les régions du cerveau sont structurellement connectées et de l'intensité de leur communication<ref>{{Cite journal|nom1=Damoiseaux|prénom1=Jessica S.|nom2=Greicius|prénom2=Michael D.|date=01/10/2009|langue=en|titre=Greater than the sum of its parts: a review of studies combining structural connectivity and resting-state functional connectivity|périodique=Brain Structure and Function|volume=213|issue=6|pages=525–533|doi=10.1007/s00429-009-0208-6|pmid=19565262|s2cid=16792748|issn=1863-2661}}.</ref>{{,}}<ref>{{Cite journal|nom1=Honey|prénom1=C. J.|nom2=Sporns|prénom2=O.|nom3=Cammoun|prénom3=L.|nom4=Gigandet|prénom4=X.|nom5=Thiran|prénom5=J. P.|nom6=Meuli|prénom6=R.|nom7=Hagmann|prénom7=P.|date=10/02/2009|titre=Predicting human resting-state functional connectivity from structural connectivity|périodique=Proceedings of the National Academy of Sciences|langue=en|volume=106|issue=6|pages=2035–2040|doi=10.1073/pnas.0811168106|issn=0027-8424|pmid=19188601|pmc=2634800|bibcode=2009PNAS..106.2035H |doi-access=free}}.</ref>. Le principal outil de recherche en connectomique à l'échelle microscopique est la préservation chimique du cerveau, suivie de la [[microscopie électronique]] en 3D<ref>{{cite journal|nom1=Anderson|prénom1=J.R.|nom2=Jones|prénom2=B.W.|nom3=Watt|prénom3=C.B.|nom4=Shaw|prénom4=M.V.|nom5=Yang|prénom5=J.H.|nom6=Demill|prénom6=D.|nom7=Lauritzen|prénom7=J.S.|nom8=Lin|prénom8=Y.|nom9=Rapp|prénom9=K.D.|nom10=Mastronarde|prénom10=D.|nom11=Koshevoy|prénom11=P.|nom12=Grimm|prénom12=B.|nom13=Tasdizen|prénom13=T.|nom14=Whitaker|prénom14=R.|nom15=Marc|prénom15=R. E.|langue=en|titre=Exploring the retinal connectome|périodique=Molecular Vision|volume=17|pages=355–79|année=2011|pmid=21311605|pmc=3036568|display-authors=8 }}.</ref>, utilisée pour la {{lien|langue=en|trad=Neural circuit reconstruction|fr=reconstruction des circuits neuronaux}}. La {{lien|langue=en|tradf=Correlative light-electron microscopy|fr=microscopie corrélative}}, qui combine la [[fluorescence]] avec la microscopie électronique 3D, permet d'obtenir des données plus interprétables, car elle est capable de détecter automatiquement des types de neurones spécifiques et de les tracer dans leur intégralité à l'aide de marqueurs fluorescents<ref>{{lien web|url=http://request.delmic.com/neuroscience|langue=en|titre=Neuroscience: Synaptic connectivity in the songbird brain - Application Note {{!}} DELMIC|nom=BV|prénom=DELMIC|site=request.delmic.com|consulté le=16/02/2017}}.</ref>. |
|||
Pour voir l'un des premiers micro-connectomes à pleine résolution, visitez le site [https://neurodata.io/project/ocp/ Open Connectome Project], qui héberge plusieurs ensembles de données sur le connectome, dont l'ensemble de données de 12 To de Bock et al. (2011). |
|||
== Systèmes modèles == |
|||
Outre le cerveau humain, certains des systèmes modèles utilisés pour la recherche en connectomique sont la souris<ref>{{cite journal|nom1=Bock|prénom1=Davi D.|nom2=Lee|prénom2=Wei-Chung Allen|nom3=Kerlin|prénom3=Aaron M.|nom4=Andermann|prénom4=Mark L.|nom5=Hood|prénom5=Greg|nom6=Wetzel|prénom6=Arthur W.|nom7=Yurgenson|prénom7=Sergey|nom8=Soucy|prénom8=Edward R.|nom9=Kim|prénom9=Hyon Suk|nom10=Reid|prénom10=R. Clay|titre=Network anatomy and in vivo physiology of visual cortical neurons|langue=en|périodique=Nature|volume=471|issue=7337|pages=177–82|année=2011|pmid=21390124|doi=10.1038/nature09802|display-authors=8|pmc=3095821|bibcode=2011Natur.471..177B }}.</ref>, la [[drosophile]]<ref name=pmid20833533>{{cite journal|nom1=Chklovskii|prénom1=Dmitri B.|nom2=Vitaladevuni|prénom2=Shiv|nom3=Scheffer|prénom3=Louis K.|langue=en|titre=Semi-automated reconstruction of neural circuits using electron microscopy|périodique=Current Opinion in Neurobiology|volume=20|issue=5|pages=667–75|année=2010|pmid=20833533|doi=10.1016/j.conb.2010.08.002|s2cid=206950616}}</ref>{{,}}<ref>{{cite journal|nom1=Zheng|prénom1=Z.|langue=en||titre=A Complete Electron Microscopy Volume of the Brain of Adult Drosophila melanogaster|périodique=Cell|date=2018|volume=174|issue=3|pages=730–743|doi=10.1016/j.cell.2018.06.019|pmid=30033368|pmc=6063995}}.</ref>, le nématode ''[[Caenorhabditis elegans|C. elegans]]''<ref name=pmid16537428>{{cite journal|nom1=Chen|prénom1=B. L.|nom2=Hall|prénom2=D. H.|nom3=Chklovskii|prénom3=D. B.|langue=en|titre=Wiring optimization can relate neuronal structure and function|périodique=Proceedings of the National Academy of Sciences|volume=103|issue=12|pages=4723–8|année=2006|doi= 10.1073/pnas.0506806103|pmid=16537428|pmc=1550972|bibcode=2006PNAS..103.4723C |doi-access=free}}.</ref>{{,}}<ref name=pmid19918070>{{cite journal|nom1=Perez-Escudero|prénom1=A.|nom2=Rivera-Alba|prénom2=M.|nom3=De Polavieja|prénom3=G. G.|langue=en|titre=Structure of deviations from optimality in biological systems|périodique=Proceedings of the National Academy of Sciences|volume=106|issue=48|pages=20544–9|année=2009| doi=10.1073/pnas.0905336106|pmid=19918070|pmc=2777958|bibcode=2009PNAS..10620544P |doi-access=free}}.</ref>, et la chouette effraie<ref name=pmid21131711>{{cite journal|nom1=Pena|prénom1=J.L.|nom2=Debello|prénom2=W.M.|langue=en|titre=Auditory processing, plasticity, and learning in the barn owl|périodique=ILAR Journal|volume=51|issue=4|pages=338–52|année=2010|pmid=21131711|pmc=3102523|doi=10.1093/ilar.51.4.338}}.</ref>. |
|||
== Applications == |
|||
[[Image:Connectivity matrix.jpg|vignette|326x326px|Matrice de connectivité évaluant la connectivité fonctionnelle entre chaque région du cerveau dans le réseau de mode par défaut (DMN). Ici, les nuances de rouge indiquent un couplage plus fort entre les changements de flux sanguin de deux régions, et les nuances de bleu indiquent une anti-corrélation entre deux régions.]] |
|||
En comparant des connectomes malades et sains, on peut mieux comprendre certaines psychopathologies, telle la [[douleur neuropathique]], et les thérapies potentielles pour celles-ci. D'une manière générale, le domaine des neurosciences bénéficierait de la normalisation et des données brutes. Les réseaux neuronaux actuels reposent principalement sur des représentations probabilistes des schémas de connectivité<ref name=pmid19662159>{{cite journal|nom1=Nordlie|prénom1=Eilen|nom2=Gewaltig|prénom2=Marc-Oliver|nom3=Plesser|prénom3=Hans Ekkehard|nom=4=Friston|prénom=4=Karl J.[|Directeur4=oui|langue=en|titre=Towards Reproducible Descriptions of Neuronal Network Models|périodique=PLOS Computational Biology|volume=5|issue=8|pages=e1000456|année=2009|pmid=19662159|pmc=2713426|doi=10.1371/journal.pcbi.1000456|bibcode=2009PLSCB...5E0456N }}.</ref>. Les matrices de connectivité (diagrammes en damier de la connectomique) ont été utilisées dans la récupération d'un [[accident vasculaire cérébral]] pour évaluer la réponse au traitement par [[stimulation magnétique transcrânienne]]<ref>{{Cite journal|nom1=Yeung|prénom1=Jacky T.|nom2=Young|prénom2=Isabella M.|nom3=Doyen|prénom3=Stephane|nom4=Teo|prénom4=Charles|nom5=Sughrue|prénom5=Michael E.|date=Octobre 2021|langue)en|titre=Changes in the Brain Connectome Following Repetitive Transcranial Magnetic Stimulation for Stroke Rehabilitation|périodique=Cureus|volume=13|issue=10|pages=e19105|doi=10.7759/cureus.19105|issn=2168-8184|pmc=8614179|pmid=34858752}}.</ref>. De même, les [[connectogramme]]s (diagrammes circulaires de la connectomique) ont été utilisés dans des cas de lésions cérébrales traumatiques pour documenter l'étendue des dommages causés aux réseaux neuronaux<ref name="Van Horn">{{cite journal|nom=Van Horn|prénom=John D.|nom2=Irimia|prénom2=Andrei|nom3=Torgerson|prénom3=C.M.|nom4=Chambers|prénom4=M.C.|nom5=Kikinis|prénom5=R.|nom6=Toga|prénom6=A.W.|langue=en|titre=Mapping connectivity damage in the case of Phineas Gage|périodique=PLOS ONE|année=2012|volume=7|issue=5|doi=10.1371/journal.pone.0037454|pmid=22616011|pmc=3353935|pages=e37454|bibcode=2012PLoSO...737454V|doi-access=free}}.</ref>{{,}}<ref name=Irimia>{{cite journal|nom1=Irimia|prénom1=Andrei|nom2=Chambers|prénom2=M.C.|nom3=Torgerson|prénom3=C.M.|nom4=Filippou|prénom4=M.|nom5=Hovda|prénom5=D.A.|nom6=Alger |prénom6= J.R.|nom7=Gerig|prénom7=G.|nom8=Toga|prénom8=A.W.|nom9=Vespa|prénom9=P.M.|nom10=Kikinis|prénom10=R.|nom11=Van Horn|prénom11=John D.|titre=Patient-tailored connectomics visualization for the assessment of white matter atrophy in traumatic brain injury|périodique=Frontiers in Neurology|date=06/02/2012|doi=10.3389/fneur.2012.00010|pmid=22363313|pmc=3275792|volume=3|pages=10|doi-access=free}}.</ref> |
|||
Le connectome humain peut être considéré comme un [[Graphe (mathématiques discrètes)|graphe]], et les riches outils, définitions et algorithmes de la [[théorie des graphes]] peuvent être appliqués à ces graphes. En comparant les connectomes (ou ''braingraphes'') de femmes et d'hommes en bonne santé, Szalkai ''et al''.<ref name=":47">{{Cite journal|langue=en|titre = Graph Theoretical Analysis Reveals: Women's Brains Are Better Connected than Men's|nom1 = Szalkai|prénom = Balazs|nom2 = Varga|prénom2 = Balint|nom3 = Grolmusz|prénom3 = Vince|date = 2015|périodique= PLOS ONE|doi = 10.1371/journal.pone.0130045|volume=10|number=7|pages=e0130045|pmid=26132764|pmc=4488527|arxiv=1501.00727|bibcode=2015PLoSO..1030045S|doi-access = free}}.</ref>{{,}}<ref name="Szalkai2017">{{cite journal|nom1=Szalkai|prénom1=Balázs|nom2=Varga|prénom2=Bálint|nom3=Grolmusz|prénom3=Vince|langue=en|titre=Brain size bias compensated graph-theoretical parameters are also better in women's structural connectomes|journal=Brain Imaging and Behavior|volume=12|issue=3|pages=663–673|year=2017|issn=1931-7565|doi=10.1007/s11682-017-9720-0|pmid=28447246|s2cid=4028467}}.</ref> ont montré que pour plusieurs paramètres profonds de la théorie des graphes, le connectome structurel des femmes est significativement mieux connecté que celui des hommes. Par exemple, le connectome des femmes a plus d'arêtes, une largeur minimale de bipartition plus élevée, un ''{{lien|langue=en|eigengap}}'' plus grand, une {{lien|langue=en|trad=Vertex cover|texte=couverture}} minimale de sommets plus grande que celui des hommes. La largeur minimale de bipartition (ou, en d'autres termes, la coupe minimale équilibrée) est une mesure bien connue de la qualité des {{lien|langue=en|trad=Multistage interconnection networks|texte=réseaux informatiques d'interconnexion à plusieurs étages}}, elle décrit les éventuels goulets d'étranglement dans la communication du réseau : Plus cette valeur est élevée, meilleur est le réseau. Le plus grand ''eigengap'' montre que le connectome des femmes est un meilleur {{lien|langue=en|trad=Taux d'expansion (théorie des graphes) |
|||
|texte=graphe expansif}} que le connectome des hommes. La meilleure propriété d'expansion, la plus grande largeur de bipartition minimale et la plus grande couverture de sommet minimale montrent des avantages profonds dans la connectivité du réseau dans le cas du connectome féminin. |
|||
Des mesures locales de différence entre les populations de ces graphes ont également été introduites, par exemple pour comparer les groupes de cas par rapport aux groupes de contrôle<ref name="Crimi2019">{{cite journal|nom1=Crimi|prénom1=Alessandro|nom2=Giancardo|prénom2=Luca|nom3=Sambataro|prénom3=Fabio|nom4=Diego|prénom4=Sona|langue=en|titre=MultiLink analysis: brain network comparison via sparse connectivity analysis|périodique= Scientific Reports|année=2019|volume=9|issue=1|pages=1–13|doi=10.1038/s41598-018-37300-4|pmid=30635604|pmc=6329758|bibcode=2019NatSR...9...65C|doi-access=free}}.</ref>[34]. Celles-ci peuvent être trouvées en utilisant soit un [[test de Student]] ajusté<ref name="Zalesky2010">{{cite journal|nom1=Zalesky|prénom1=Andrew|nom2=Fornito|prénom2=Alex|nom3=Bullmore|prénom3=Edward|langue=en|titre=Network-based statistic: identifying differences in brain networks.|périodique= NeuroImage|année=2010|volume=53|issue=4|pages=1197–1207|doi=10.1016/j.neuroimage.2010.06.041|pmid=20600983|s2cid=17760084}}.</ref>, soit un modèle de sparsité<ref name="Crimi2019"/>, dans le but de trouver des connexions statistiquement significatives qui sont différentes parmi ces groupes. |
|||
Les connectomes humains ont une variabilité individuelle, qui peut être mesurée à l'aide de la [[Fonction de répartition|fonction de distribution cumulative]], comme cela a été montré dans la référence<ref name="Kerepesi2018">{{cite journal|nom1=Kerepesi|prénom1=Csaba|nom2=Szalkai|prénom2=Balázs|nom3=Varga|prénom3=Bálint|nom4=Grolmusz|prénom4=Vince|langue=en|titre=Comparative connectomics: Mapping the inter-individual variability of connections within the regions of the human brain|périodique= Neuroscience Letters|année=2018|volume=662|issue=1|pages=17–21|doi=10.1016/j.neulet.2017.10.003|pmid=28988973|arxiv=1507.00327|s2cid=378080}}.</ref>. En analysant la variabilité individuelle des connectomes humains dans des zones cérébrales distinctes, on a constaté que les lobes frontal et limbique sont plus conservateurs, et que les bords des lobes temporal et occipital sont plus diversifiés. Une distribution « hybride » conservatrice/diversifiée a été détectée dans le [[lobule paracentral]] et le [[gyrus fusiforme]]. Des zones corticales plus petites ont également été évaluées : les [[gyrus précentral|gyrus précentraux]] se sont révélés plus conservateurs, tandis que les [[gyrus postcentral|gyrus postcentraux]] et [[Gyrus temporal supérieur|temporaux supérieurs]] étaient très diversifiés. |
|||
== Comparaison avec la génomique == |
|||
Le [[Projet Génome humain|Projet ''Génome humain'']] a initialement fait l'objet de plusieurs des critiques susmentionnées, mais il a néanmoins été achevé plus tôt que prévu et a permis de nombreuses avancées en génétique. Certains ont fait valoir que des analogies peuvent être faites entre la génomique et la connectomique, et que nous devrions donc être au moins légèrement plus optimistes quant aux perspectives de la connectomique<ref name=pmid18801435>{{cite journal|nom1=Lichtman|prénom1=J|nom2=Sanes|prénom2=J.|langue=en|titre=Ome sweet ome: what can the genome tell us about the connectome?|périodique=Current Opinion in Neurobiology|volume=18|issue=3|pages=346–53|année=2008|pmid=18801435|pmc=2735215|doi=10.1016/j.conb.2008.08.010}}.</ref>. D'autres ont critiqué les tentatives d'élaboration d'un connectome à micro-échelle, arguant que nous n'avons pas suffisamment de connaissances sur les endroits où chercher des informations, ou qu'il ne peut être réalisé dans un délai réaliste<ref name="Seeking the Connectome, a Mental Map, Slice by Slice">{{lien web|langue=en-US|url=https://www.nytimes.com/2010/12/28/science/28brain.html|site=The New York Times|prénom=Ashlee|nom=Vance|titre=Seeking the Connectome, a Mental Map, Slice by Slice|date=27/12/2010|consulté le=03/03/2023}}.</ref>. |
|||
== Le jeu ''EyeWire'' == |
|||
{{article détaillé|EyeWire}} |
|||
''Eyewire'' est un jeu en ligne développé par le scientifique américain {{lien|langue=en|Sebastian Seung}} de l'[[Université de Princeton]]. Il utilise l'{{lien|langue=en|trad=Social computing|fr=informatique sociale}} pour aider à cartographier le connectome du cerveau. Il a attiré plus de 130 000 joueurs de plus de 100 pays. |
|||
== Données publiques == |
|||
Sites web permettant d'explorer les ensembles de données connectomiques accessibles au public : |
|||
Connectomique à macro-échelle (données sur de jeunes adultes en bonne santé) : |
|||
* Human Connectome Project [https://www.humanconnectome.org/study/hcp-young-adult Young Adult] |
|||
* Amsterdam [https://nilab-uva.github.io/AOMIC.github.io/ Open MRI Collection] |
|||
* Harvard Brain Genomic [https://dataverse.harvard.edu/dataverse/GSP Superstruct Project] |
|||
Pour une liste plus complète de jeux de données ouverts à grande échelle, consultez [https://www.sciencedirect.com/science/article/pii/S1053811921008521 cet article]. |
|||
Connectomique à micro-échelle : |
|||
* Whole C. elegans [https://wormwiring.org/ connectome] |
|||
* NeuPRINT [https://neuprint.janelia.org/ Fly Hemibrain] |
|||
* [https://flywire.ai/ Flywire] (whole fly brain) |
|||
* [https://www.microns-explorer.org/ MICrONS Explorer] (mouse cortical data) |
|||
* [https://h01-release.storage.googleapis.com/landing.html H01 Browser] Release (human cortical data) |
|||
== Voir aussi == |
== Voir aussi == |
||
{{Autres projets|wiktionary=connectomique}} |
{{Autres projets|wiktionary=connectomique}} |
||
* {{en}} [[:en:Dynamic functional connectivity|Dynamic functional connectivity]] |
|||
* {{en}} [[:en:List of functional connectivity software|List of functional connectivity software]] |
|||
* {{en}} [[:en:Human Connectome Project|Human Connectome Project]] |
|||
* {{en}} [[:en:Budapest Reference Connectome|Budapest Reference Connectome]] |
|||
== Notes et références == |
== Notes et références == |
||
{{Traduction/Référence|en| Connectomics |1136410978}} |
|||
{{Références}} |
{{Références}} |
||
Version du 3 mars 2023 à 16:35

La connectomique est l'établissement et l'étude du connectome, c'est-à-dire de l'ensemble des connexions neuronales du cerveau.
La connectomique est la production et l'étude des connectomes : des cartes complètes des connexions au sein du système nerveux d'un organisme. Plus généralement, on peut considérer qu'il s'agit de l'étude des schémas de câblage neuronaux, en mettant l'accent sur la façon dont la connectivité structurelle, les synapses individuelles, la morphologie et l'ultrastructure cellulaires contribuent à la constitution d'un réseau. Le système nerveux est un réseau constitué de milliards de connexions et ces connexions sont responsables de nos pensées, émotions, actions, souvenirs, fonctions et dysfonctionnements. Par conséquent, l'étude de la connectomique vise à faire progresser notre compréhension de la santé mentale et de la cognition en comprenant comment les cellules du système nerveux sont connectées et communiquent. Ces structures étant extrêmement complexes, les méthodes de ce domaine utilisent une application de criblage à haut débit d'imagerie neuronale fonctionnelle et structurelle, le plus souvent l'imagerie par résonance magnétique (IRM), la microscopie électronique et les techniques histologiques, afin d'augmenter la vitesse, l'efficacité et la résolution de ces cartes du système nerveux. À ce jour, des dizaines d'ensembles de données à grande échelle ont été collectés, couvrant le système nerveux, y compris les différentes zones du cortex, le cervelet[1],[2], la rétine[3], le système nerveux périphérique[4] et les jonctions neuromusculaires[5].
D'une manière générale, il existe deux types de connectomes : les connectomes à macro-échelle et les connectomes à micro-échelle. La connectomique à macro-échelle consiste à utiliser les données de l'IRM fonctionnelle et structurelle pour cartographier les grands trajets de fibres et les zones de matière grise fonctionnelle dans le cerveau en termes de flux sanguin (fonctionnel) et de diffusivité de l'eau (structurel). La connectomique à micro-échelle est la cartographie du connectome complet des petits organismes à l'aide de la microscopie et de l'histologie ; on considère donc là toutes les connexions qui existent dans leur système nerveux central.
Méthodes
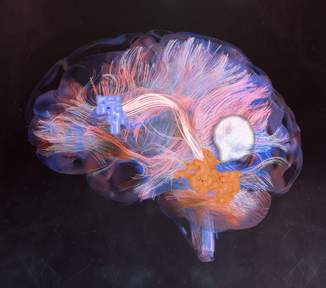
Connectomique à macro-échelle
Les connectomes à macro-échelle sont généralement collectés à l'aide de l'imagerie par résonance magnétique de diffusion (IRMd) et de l'imagerie par résonance magnétique fonctionnelle (IRMf). Les ensembles de données d'IRMd peuvent couvrir l'ensemble du cerveau, en imagerie de la substance blanche entre le cortex et le sous-cortex. En revanche, les ensembles de données d'IRMf mesurent le débit sanguin cérébral, en tant que marqueur de l'activation neuronale. L'un des avantages de l'IRM est qu'elle fournit des informations in vivo sur la connectivité entre les différentes zones du cerveau. La connectomique à macro-échelle a permis de mieux comprendre divers réseaux cérébraux, notamment les réseaux visuels[6],[7], du tronc cérébral[8],[9], et du langage[10],[11].
Connectomique à micro-échelle
D'autre part, les connectomes à micro-échelle se concentrent sur une zone beaucoup plus petite du système nerveux avec une résolution beaucoup plus élevée. Ces ensembles de données sont généralement collectés à l'aide de l'imagerie par microscopie électronique et offrent une résolution synaptique unique de circuits locaux entiers. Parmi les jalons de la connectomique à micro-échelle, citons le système nerveux entier du nématode Caenorhabditis elegans[12], un cerveau entier de mouche[13] et, plus récemment, un cube millimétrique des cortex de la souris[14] et de l'homme[15].
Outils
L'IRM est l'un des principaux outils utilisés pour la recherche en connectomique à l'échelle macroscopique[16]. Lorsqu'elles sont utilisées ensemble, un ensemble de données d'IRMf et d'IRMd à l'état de repos fournit une vue complète de la manière dont les régions du cerveau sont structurellement connectées et de l'intensité de leur communication[17],[18]. Le principal outil de recherche en connectomique à l'échelle microscopique est la préservation chimique du cerveau, suivie de la microscopie électronique en 3D[19], utilisée pour la reconstruction des circuits neuronaux (en). La microscopie corrélative (en), qui combine la fluorescence avec la microscopie électronique 3D, permet d'obtenir des données plus interprétables, car elle est capable de détecter automatiquement des types de neurones spécifiques et de les tracer dans leur intégralité à l'aide de marqueurs fluorescents[20].
Pour voir l'un des premiers micro-connectomes à pleine résolution, visitez le site Open Connectome Project, qui héberge plusieurs ensembles de données sur le connectome, dont l'ensemble de données de 12 To de Bock et al. (2011).
Systèmes modèles
Outre le cerveau humain, certains des systèmes modèles utilisés pour la recherche en connectomique sont la souris[21], la drosophile[22],[23], le nématode C. elegans[24],[25], et la chouette effraie[26].
Applications

En comparant des connectomes malades et sains, on peut mieux comprendre certaines psychopathologies, telle la douleur neuropathique, et les thérapies potentielles pour celles-ci. D'une manière générale, le domaine des neurosciences bénéficierait de la normalisation et des données brutes. Les réseaux neuronaux actuels reposent principalement sur des représentations probabilistes des schémas de connectivité[27]. Les matrices de connectivité (diagrammes en damier de la connectomique) ont été utilisées dans la récupération d'un accident vasculaire cérébral pour évaluer la réponse au traitement par stimulation magnétique transcrânienne[28]. De même, les connectogrammes (diagrammes circulaires de la connectomique) ont été utilisés dans des cas de lésions cérébrales traumatiques pour documenter l'étendue des dommages causés aux réseaux neuronaux[29],[30]
Le connectome humain peut être considéré comme un graphe, et les riches outils, définitions et algorithmes de la théorie des graphes peuvent être appliqués à ces graphes. En comparant les connectomes (ou braingraphes) de femmes et d'hommes en bonne santé, Szalkai et al.[31],[32] ont montré que pour plusieurs paramètres profonds de la théorie des graphes, le connectome structurel des femmes est significativement mieux connecté que celui des hommes. Par exemple, le connectome des femmes a plus d'arêtes, une largeur minimale de bipartition plus élevée, un eigengap (en) plus grand, une couverture minimale de sommets plus grande que celui des hommes. La largeur minimale de bipartition (ou, en d'autres termes, la coupe minimale équilibrée) est une mesure bien connue de la qualité des réseaux informatiques d'interconnexion à plusieurs étages (en), elle décrit les éventuels goulets d'étranglement dans la communication du réseau : Plus cette valeur est élevée, meilleur est le réseau. Le plus grand eigengap montre que le connectome des femmes est un meilleur graphe expansif que le connectome des hommes. La meilleure propriété d'expansion, la plus grande largeur de bipartition minimale et la plus grande couverture de sommet minimale montrent des avantages profonds dans la connectivité du réseau dans le cas du connectome féminin.
Des mesures locales de différence entre les populations de ces graphes ont également été introduites, par exemple pour comparer les groupes de cas par rapport aux groupes de contrôle[33][34]. Celles-ci peuvent être trouvées en utilisant soit un test de Student ajusté[34], soit un modèle de sparsité[33], dans le but de trouver des connexions statistiquement significatives qui sont différentes parmi ces groupes.
Les connectomes humains ont une variabilité individuelle, qui peut être mesurée à l'aide de la fonction de distribution cumulative, comme cela a été montré dans la référence[35]. En analysant la variabilité individuelle des connectomes humains dans des zones cérébrales distinctes, on a constaté que les lobes frontal et limbique sont plus conservateurs, et que les bords des lobes temporal et occipital sont plus diversifiés. Une distribution « hybride » conservatrice/diversifiée a été détectée dans le lobule paracentral et le gyrus fusiforme. Des zones corticales plus petites ont également été évaluées : les gyrus précentraux se sont révélés plus conservateurs, tandis que les gyrus postcentraux et temporaux supérieurs étaient très diversifiés.
Comparaison avec la génomique
Le Projet Génome humain a initialement fait l'objet de plusieurs des critiques susmentionnées, mais il a néanmoins été achevé plus tôt que prévu et a permis de nombreuses avancées en génétique. Certains ont fait valoir que des analogies peuvent être faites entre la génomique et la connectomique, et que nous devrions donc être au moins légèrement plus optimistes quant aux perspectives de la connectomique[36]. D'autres ont critiqué les tentatives d'élaboration d'un connectome à micro-échelle, arguant que nous n'avons pas suffisamment de connaissances sur les endroits où chercher des informations, ou qu'il ne peut être réalisé dans un délai réaliste[37].
Le jeu EyeWire
Eyewire est un jeu en ligne développé par le scientifique américain Sebastian Seung (en) de l'Université de Princeton. Il utilise l'informatique sociale (en) pour aider à cartographier le connectome du cerveau. Il a attiré plus de 130 000 joueurs de plus de 100 pays.
Données publiques
Sites web permettant d'explorer les ensembles de données connectomiques accessibles au public :
Connectomique à macro-échelle (données sur de jeunes adultes en bonne santé) :
- Human Connectome Project Young Adult
- Amsterdam Open MRI Collection
- Harvard Brain Genomic Superstruct Project
Pour une liste plus complète de jeux de données ouverts à grande échelle, consultez cet article.
Connectomique à micro-échelle :
- Whole C. elegans connectome
- NeuPRINT Fly Hemibrain
- Flywire (whole fly brain)
- MICrONS Explorer (mouse cortical data)
- H01 Browser Release (human cortical data)
Voir aussi
- (en) Dynamic functional connectivity
- (en) List of functional connectivity software
- (en) Human Connectome Project
- (en) Budapest Reference Connectome
Notes et références
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Connectomics » (voir la liste des auteurs).
- (en) Angelo Quartarone, Alberto Cacciola, Demetrio Milardi, Maria Felice Ghilardi, Alessandro Calamuneri, Gaetana Chillemi, Giuseppe Anastasi et John Rothwell, « New insights into cortico-basal-cerebellar connectome: clinical and physiological considerations », Brain, vol. 143, no 2, , p. 396–406 (ISSN 0006-8950, PMID 31628799, DOI 10.1093/brain/awz310)
- (en) Tri M. Nguyen, Logan A. Thomas, Jeff L. Rhoades, Ilaria Ricchi, Xintong Cindy Yuan, Arlo Sheridan, David G. C. Hildebrand, Jan Funke, Wade G. Regehr et Wei-Chung Allen Lee, « Structured connectivity in the cerebellum enables noise-resilient pattern separation », bioRxiv-10.1101/2021.11.29.470455v1, , p. 2021.11.29.470455.
- Moritz Helmstaedter, Kevin L. Briggman, Turaga Srinivas C., Jain Viren, Seung H. Sebastian et Denk Winfried, « Connectomic reconstruction of the inner plexiform layer in the mouse retina », Nature, vol. 500, no 7461, , p. 168–174 (ISSN 0028-0836, PMID 23925239, DOI 10.1038/nature12346, Bibcode 2013Natur.500..168H, S2CID 3119909).
- (en) Jasper S. Phelps, David Grant Colburn Hildebrand et Brett J. Graham, « Reconstruction of motor control circuits in adult Drosophila using automated transmission electron microscopy », Cell, vol. 184, no 3, , p. 759–774.e18 (ISSN 0092-8674, PMID 33400916, PMCID 8312698, DOI 10.1016/j.cell.2020.12.013).
- Tjeerd W. Boonstra, Danna-Dos-Santos Alessander, Hong-Bo Xie., Roerdink Melvyn, Stins John F. et Michael Breakspear, « Muscle networks: Connectivity analysis of EMG activity during postural control », Scientific Reports, vol. 5, , p. 17830 (PMID 26634293, PMCID 4669476, DOI 10.1038/srep17830, Bibcode 2015NatSR...517830B).
- (en) Alexandra Kammen, Meng Law, Bosco S. Tjan, Arthur W. Toga et Yonggang Shi, « Automated retinofugal visual pathway reconstruction with multi-shell HARDI and FOD-based analysis », NeuroImage, vol. 125, , p. 767–779 (ISSN 1053-8119, PMID 26551261, PMCID 4691391, DOI 10.1016/j.neuroimage.2015.11.005).
- (en) M. Yogarajah, N. K. Focke, S. Bonelli, M. Cercignani, J. Acheson, G. J. M. Parker, D. C. Alexander, A. W. McEvoy, M. R. Symms, M. J. Koepp et J. S. Duncan, « Defining Meyer's loop-temporal lobe resections, visual field deficits and diffusion tensor tractography », Brain, vol. 132, no 6, , p. 1656–1668 (ISSN 0006-8950, PMID 19460796, PMCID 2685925, DOI 10.1093/brain/awp114).
- (en) Rudolf Nieuwenhuys, Jan Voogd et Christiaan van Huijzen, The Human Central Nervous System, (ISBN 978-3-540-34684-5, DOI 10.1007/978-3-540-34686-9)
- (en) George Paxinos, Huang Xu-Feng, Gulgun Sengul et Charles Watson, « Organization of Brainstem Nuclei », dans The Human Nervous System, Elsevier, , 260–327 p. (ISBN 9780123742360, DOI 10.1016/b978-0-12-374236-0.10008-2, lire en ligne).
- (en) Matthew F. Glasser et James K. Rilling, « DTI Tractography of the Human Brain's Language Pathways », Cerebral Cortex, vol. 18, no 11, , p. 2471–2482 (ISSN 1460-2199, PMID 18281301, DOI 10.1093/cercor/bhn011).
- (en) Marco Catani, Derek K. Jones et Dominic H. ffytche, « Perisylvian language networks of the human brain », Annals of Neurology, vol. 57, no 1, , p. 8–16 (ISSN 0364-5134, PMID 15597383, DOI 10.1002/ana.20319, S2CID 17743067).
- (en) J. G. White, E. Southgate, J. N. Thomson et S. Brenner, « The structure of the nervous system of the nematode Caenorhabditis elegans », Philosophical Transactions of the Royal Society of London. B, Biological Sciences, vol. 314, no 1165, , p. 1–340 (ISSN 0080-4622, PMID 22462104, DOI 10.1098/rstb.1986.0056, Bibcode 1986RSPTB.314....1W)
- (en) Louis K Scheffer, C Shan Xu, Michal Januszewski, Zhiyuan Lu, Shin-ya Takemura, Kenneth J Hayworth, Gary B Huang, Kazunori Shinomiya, Jeremy Maitlin-Shepard, Stuart Berg, Jody Clements, Eve Marder (dir.), Michael B Eisen (dir.), Jason Pipkin (dir.) et Chris Q Doe (dir.), « A connectome and analysis of the adult Drosophila central brain », eLife, vol. 9, , e57443 (ISSN 2050-084X, PMID 32880371, PMCID 7546738, DOI 10.7554/eLife.57443).
- (en) J. Alexander Bae, Mahaly Baptiste, Agnes L. Bodor, Derrick Brittain, JoAnn Buchanan, Daniel J. Bumbarger, Manuel A. Castro, Brendan Celii, Erick Cobos et Forrest Collman, « Functional connectomics spanning multiple areas of mouse visual cortex », biorxiv=10.1101/2021.07.28.454025, .
- (en) Shapson-Coe, Michał Januszewski, Daniel R. Berger, Art Pope, Yuelong Wu, Tim Blakely, Richard L. Schalek, Peter Li, Shuohong Wang, Jeremy Maitin-Shepard et Neha Karlupia, « A connectomic study of a petascale fragment of human cerebral cortex », bioRxiv-10.1101/2021.05.29.446289, .
- (en) V.J. Wedeen, R.P. Wang et J.D. Schmahmann, « Diffusion spectrum magnetic resonance imaging (DSI) tractography of crossing fibers », NeuroImage, vol. 41, no 4, , p. 1267–77 (PMID 18495497, DOI 10.1016/j.neuroimage.2008.03.036, S2CID 2660208).
- (en) Jessica S. Damoiseaux et Michael D. Greicius, « Greater than the sum of its parts: a review of studies combining structural connectivity and resting-state functional connectivity », Brain Structure and Function, vol. 213, no 6, , p. 525–533 (ISSN 1863-2661, PMID 19565262, DOI 10.1007/s00429-009-0208-6, S2CID 16792748).
- (en) C. J. Honey, O. Sporns, L. Cammoun, X. Gigandet, J. P. Thiran, R. Meuli et P. Hagmann, « Predicting human resting-state functional connectivity from structural connectivity », Proceedings of the National Academy of Sciences, vol. 106, no 6, , p. 2035–2040 (ISSN 0027-8424, PMID 19188601, PMCID 2634800, DOI 10.1073/pnas.0811168106, Bibcode 2009PNAS..106.2035H).
- (en) J.R. Anderson, B.W. Jones, C.B. Watt, M.V. Shaw, J.H. Yang, D. Demill, J.S. Lauritzen, Y. Lin, K.D. Rapp, D. Mastronarde, P. Koshevoy, B. Grimm, T. Tasdizen, R. Whitaker et R. E. Marc, « Exploring the retinal connectome », Molecular Vision, vol. 17, , p. 355–79 (PMID 21311605, PMCID 3036568).
- (en) DELMIC BV, « Neuroscience: Synaptic connectivity in the songbird brain - Application Note | DELMIC », sur request.delmic.com (consulté le ).
- (en) Davi D. Bock, Wei-Chung Allen Lee, Aaron M. Kerlin, Mark L. Andermann, Greg Hood, Arthur W. Wetzel, Sergey Yurgenson, Edward R. Soucy, Hyon Suk Kim et R. Clay Reid, « Network anatomy and in vivo physiology of visual cortical neurons », Nature, vol. 471, no 7337, , p. 177–82 (PMID 21390124, PMCID 3095821, DOI 10.1038/nature09802, Bibcode 2011Natur.471..177B).
- (en) Dmitri B. Chklovskii, Shiv Vitaladevuni et Louis K. Scheffer, « Semi-automated reconstruction of neural circuits using electron microscopy », Current Opinion in Neurobiology, vol. 20, no 5, , p. 667–75 (PMID 20833533, DOI 10.1016/j.conb.2010.08.002, S2CID 206950616)
- (en) Z. Zheng, « A Complete Electron Microscopy Volume of the Brain of Adult Drosophila melanogaster », Cell, vol. 174, no 3, , p. 730–743 (PMID 30033368, PMCID 6063995, DOI 10.1016/j.cell.2018.06.019).
- (en) B. L. Chen, D. H. Hall et D. B. Chklovskii, « Wiring optimization can relate neuronal structure and function », Proceedings of the National Academy of Sciences, vol. 103, no 12, , p. 4723–8 (PMID 16537428, PMCID 1550972, DOI 10.1073/pnas.0506806103, Bibcode 2006PNAS..103.4723C).
- (en) A. Perez-Escudero, M. Rivera-Alba et G. G. De Polavieja, « Structure of deviations from optimality in biological systems », Proceedings of the National Academy of Sciences, vol. 106, no 48, , p. 20544–9 (PMID 19918070, PMCID 2777958, DOI 10.1073/pnas.0905336106, Bibcode 2009PNAS..10620544P).
- (en) J.L. Pena et W.M. Debello, « Auditory processing, plasticity, and learning in the barn owl », ILAR Journal, vol. 51, no 4, , p. 338–52 (PMID 21131711, PMCID 3102523, DOI 10.1093/ilar.51.4.338).
- (en) Eilen Nordlie, Marc-Oliver Gewaltig et Hans Ekkehard Plesser, « Towards Reproducible Descriptions of Neuronal Network Models », PLOS Computational Biology, vol. 5, no 8, , e1000456 (PMID 19662159, PMCID 2713426, DOI 10.1371/journal.pcbi.1000456, Bibcode 2009PLSCB...5E0456N).
- Jacky T. Yeung, Isabella M. Young, Stephane Doyen, Charles Teo et Michael E. Sughrue, « Changes in the Brain Connectome Following Repetitive Transcranial Magnetic Stimulation for Stroke Rehabilitation », Cureus, vol. 13, no 10, , e19105 (ISSN 2168-8184, PMID 34858752, PMCID 8614179, DOI 10.7759/cureus.19105).
- (en) John D. Van Horn, Andrei Irimia, C.M. Torgerson, M.C. Chambers, R. Kikinis et A.W. Toga, « Mapping connectivity damage in the case of Phineas Gage », PLOS ONE, vol. 7, no 5, , e37454 (PMID 22616011, PMCID 3353935, DOI 10.1371/journal.pone.0037454, Bibcode 2012PLoSO...737454V).
- Andrei Irimia, M.C. Chambers, C.M. Torgerson, M. Filippou, D.A. Hovda, J.R. Alger, G. Gerig, A.W. Toga, P.M. Vespa, R. Kikinis et John D. Van Horn, « Patient-tailored connectomics visualization for the assessment of white matter atrophy in traumatic brain injury », Frontiers in Neurology, vol. 3, , p. 10 (PMID 22363313, PMCID 3275792, DOI 10.3389/fneur.2012.00010).
- (en) Balazs Szalkai, Balint Varga et Vince Grolmusz, « Graph Theoretical Analysis Reveals: Women's Brains Are Better Connected than Men's », PLOS ONE, vol. 10, , e0130045 (PMID 26132764, PMCID 4488527, DOI 10.1371/journal.pone.0130045, Bibcode 2015PLoSO..1030045S, arXiv 1501.00727).
- (en) Balázs Szalkai, Bálint Varga et Vince Grolmusz, « Brain size bias compensated graph-theoretical parameters are also better in women's structural connectomes », Brain Imaging and Behavior, vol. 12, no 3, , p. 663–673 (ISSN 1931-7565, PMID 28447246, DOI 10.1007/s11682-017-9720-0, S2CID 4028467).
- (en) Alessandro Crimi, Luca Giancardo, Fabio Sambataro et Sona Diego, « MultiLink analysis: brain network comparison via sparse connectivity analysis », Scientific Reports, vol. 9, no 1, , p. 1–13 (PMID 30635604, PMCID 6329758, DOI 10.1038/s41598-018-37300-4, Bibcode 2019NatSR...9...65C).
- (en) Andrew Zalesky, Alex Fornito et Edward Bullmore, « Network-based statistic: identifying differences in brain networks. », NeuroImage, vol. 53, no 4, , p. 1197–1207 (PMID 20600983, DOI 10.1016/j.neuroimage.2010.06.041, S2CID 17760084).
- (en) Csaba Kerepesi, Balázs Szalkai, Bálint Varga et Vince Grolmusz, « Comparative connectomics: Mapping the inter-individual variability of connections within the regions of the human brain », Neuroscience Letters, vol. 662, no 1, , p. 17–21 (PMID 28988973, DOI 10.1016/j.neulet.2017.10.003, arXiv 1507.00327, S2CID 378080).
- (en) J Lichtman et J. Sanes, « Ome sweet ome: what can the genome tell us about the connectome? », Current Opinion in Neurobiology, vol. 18, no 3, , p. 346–53 (PMID 18801435, PMCID 2735215, DOI 10.1016/j.conb.2008.08.010).
- (en-US) Ashlee Vance, « Seeking the Connectome, a Mental Map, Slice by Slice », sur The New York Times, (consulté le ).
