Polyploïdie
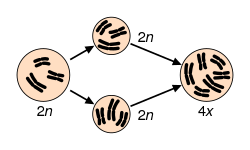
La polyploïdie est le fait, chez un être vivant, de posséder un patrimoine chromosomique au moins égal à 3 lots complets de chromosomes (3 n) voire plus.
L'autopolyploïdie résulte d'une multiplication d'un même génome, alors que l'allopolyploïdie représente un montage réunissant deux ou plusieurs génomes différents.
Occurrence dans la nature[modifier | modifier le code]
Le phénomène est assez rare dans le règne animal, mais on peut citer le cas des grenouilles africaines ou du rat-viscache, tétraploïde potentiel. Le Crapaud Batura, du nord du Pakistan, est une espèce triploïde, et qui se reproduit par la reproduction sexuée[4].
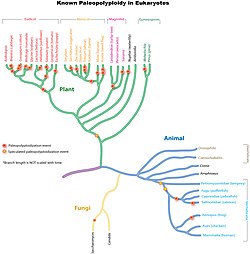
Les végétaux supportent beaucoup mieux la polyploïdie, et nombre des espèces cultivées par l'homme en sont (George Ledyard Stebbins qui attribue la forte proportion de polyploïdes dans un groupe de plantes à la fréquence de contacts secondaires entre populations diploïdes isolées, inventorie ces polyploïdes dans son ouvrage Variation and Evolution in Plants, 1950). 70 % des Angiospermes ont connu au moins un évènement de polyploïdisation[5]. Certains mutants d'Arabidopsis thaliana faisant plus ou moins d’endoréduplication ont été décrits dans des articles scientifiques. Les valeurs les plus élevées découvertes sont de 8 192× dans le suspenseur de Phaseolus coccineus et de 24 576× dans l'haustorium de l'endosperme d’Arum maculatum[6].
La récurrence des évènements de polyploïdie (selon leur ancienneté, on distingue les paléopolyploïdes, les mésopolyploïdes et les néopolyploïdes) explique que la polyploïdisation est un facteur de diversification[7] chez les animaux et les plantes[8].
La redondance des gènes créée par la duplication suivant la polyploïdisation est effacée par une élimination massive bien qu'incomplète des gènes dupliqués, appelée fractionnement des génomes. Ce fractionnement conduit à un retour à l'état diploïde par un processus dit de diploïdisation. La recherche actuelle s'efforce de déterminer si cette perte de gènes est aléatoire ou biaisée[9].
Effets[modifier | modifier le code]
Les effets de la polyploïdie sur le phénotype peuvent être : « effet de gigantisme » cellulaire et morphologique (« morphologie gigas » quasi systématique lors des polyploïdisations artificielles alors que chez les polyploïdes naturels, le gigantisme cellulaire peut être compensé par une baisse du nombre de cellules par organe)[10], croissance plus faible (par la réduction du nombre de mitoses). Les effets génétiques et épigénétiques peuvent être : modification de la séquence génomique (mutations, recombinaisons...), masquage des allèles délétères, modification de l'expression des gènes[11]. Généralement, les polyploïdes ont une barrière post-zygotique empêchant l'hybridation avec leurs parents ou les hybrides sont incapables de se développer normalement. Cet isolement sexuel immédiat est à l'origine d'une spéciation sympatrique instantanée responsable de la genèse de près de la moitié des espèces actuelles de plantes à fleurs et d'un petit nombre d'espèces animales[12].
Chez l'humain[modifier | modifier le code]

Affectant 1 à 3 % des fécondations humaines, la polyploïdie est l'anomalie de la fécondation la plus fréquente. Elle provoque généralement une fausse couche spontanée : on la retrouve dans 26 % des fausses couches dues à des erreurs chromosomiques[13].
Quand la grossesse se prolonge au delà du premier trimestre, le syndrome triploïde (d) ![]() et le syndrome tétraploïde (d)
et le syndrome tétraploïde (d) ![]() conduisent généralement à une mort in utero[14]. Exceptionnellement, l'enfant peut survivre quelques semaines après la naissance[13].
conduisent généralement à une mort in utero[14]. Exceptionnellement, l'enfant peut survivre quelques semaines après la naissance[13].
La triploïdie en mosaïque ou mosaïque (2N/3N) est moins sévère et peut être viable[13].
Notes et références[modifier | modifier le code]
- (en) Kenneth H. Wolfe, « Yesterday's polyploids and the mystery of diploidization », Nature Reviews Genetics, vol. 2, no 5, , p. 333–341 (ISSN 1471-0056, DOI 10.1038/35072009, résumé).
- (en) Keith L. Adams et Jonathan F. Wendel, « Polyploidy and genome evolution in plants », Current Opinion in Plant Biology, vol. 8, no 2, , p. 135–141 (ISSN 1369-5266, DOI 10.1016/j.pbi.2005.01.001, résumé).
- (en) Liying Cui, P. Kerr Wall, James H. Leebens-Mack, Bruce G. Lindsay, Douglas E. Soltis, Jeff J. Doyle, Pamela S. Soltis, John E. Carlson, Kathiravetpilla Arumuganathan, Abdelali Barakat, Victor A. Albert, Hong Ma et Claude W. dePamphilis, « Widespread genome duplications throughout the history of flowering plants », Genome Research, vol. 16, no 6, , p. 738–749 (ISSN 1088-9051, DOI 10.1101/gr.4825606, lire en ligne).
- Le crapaud qui divise 3 par 2
- (en) G. Moore, « Meiosis in allopolyploids - the importance of "teflon" chromosomes », Trends in genetics, vol. 18, no 9, , p. 456-463
- Wilhelm Nultsch & R. Miesch, Botanique générale, De Boeck Université, , p. 119
- Duplication des génomes chez les plantes et les animaux, tiré de Van de Peer 2009, Nature Review genetics
- (en) Y. Van de Peer et al, « The evolutionary significance of ancient genome duplications », Nat Rev Genet., vol. 10, no 10, , p. 725-732 (DOI 10.1038/nrg2600).
- (en) Richard J Langham, Justine Walsh, Molly Dunn, Cynthia Ko, Stephen A Goff & Michael Freeling, « Genomic duplication, fractionation and the origin of regulatory novelty », Genetics, vol. 166, no 2, , p. 935-945.
- (en) A. Müntzing, « The evolutionary significance of autopolyploidy », Hereditas, vol. 21, , p. 263-378
- (en) T. C. Osborn et coll, « Understanding mechanisms of novel gene expression in polyploids », Trends in Genetics, vol. 19, , p. 141-147
- Edward Osborne Wilson, La diversité de la vie, Odile Jacob, , p. 98.
- Polyploïdies, Dr Aly Abbara
- Fiche orphanet du syndrome triploïde
Voir aussi[modifier | modifier le code]
Articles connexes[modifier | modifier le code]
- Paléopolyploïdie
- Aneuploïdie
- Ploïdie
- Trisomie
- Colchicine
- Triangle d'U
- Syndrome de Klinefelter
- Syndrome 49,XXXYY
