« Protéines alternatives » : différence entre les versions
Aucun résumé des modifications |
|||
| Ligne 6 : | Ligne 6 : | ||
Les '''protéines''' '''alternatives''' sont des [[Protéine|protéines]] traduites à partir de [[Séquence codante|séquences codantes]] alternatives localisées dans les [[Acide ribonucléique messager|ARNm matures]].<br /> |
Les '''protéines''' '''alternatives''' sont des [[Protéine|protéines]] traduites à partir de [[Séquence codante|séquences codantes]] alternatives localisées dans les [[Acide ribonucléique messager|ARNm matures]].<br /> |
||
Les protéines sont des molécules essentielles à la vie. La [[synthèse des protéines]] s'effectue à partir de [[gènes]] codant pour des protéines. La synthèse des protéines se fait en deux étapes, la [[Transcription (biologie)|transcription]] et la [[traduction]]. La transcription permet la synthèse d'[[ARN pré-messager|ARN pré-messagers]] qui subissent un certain nombre de modifications pour donner des ARNm matures. Dans les ARNm, la région codante ou [[séquence codante]] (aussi appelée CDS pour Coding DNA Sequence), code pour une protéine spécifique. Chez les [[Eukaryote|eukaryotes]], le dogme actuel de la biologie moléculaire associe une seule séquence codante à chaque ARNm mature, et donc une seule protéine. Cette séquence codante est appelée séquence codante de référence et la protéine correspondante est appelée protéine de référence. Les protéines de référence sont répertoriées dans des bases de données telle que [[Uniprot]]. Or, il existe des séquences codantes alternatives différentes des séquences codantes de référence<ref>Vanderperre |
Les protéines sont des molécules essentielles à la vie. La [[synthèse des protéines]] s'effectue à partir de [[gènes]] codant pour des protéines. La synthèse des protéines se fait en deux étapes, la [[Transcription (biologie)|transcription]] et la [[traduction]]. La transcription permet la synthèse d'[[ARN pré-messager|ARN pré-messagers]] qui subissent un certain nombre de modifications pour donner des ARNm matures. Dans les ARNm, la région codante ou [[séquence codante]] (aussi appelée CDS pour Coding DNA Sequence), code pour une protéine spécifique. Chez les [[Eukaryote|eukaryotes]], le dogme actuel de la biologie moléculaire associe une seule séquence codante à chaque ARNm mature, et donc une seule protéine. Cette séquence codante est appelée séquence codante de référence et la protéine correspondante est appelée protéine de référence. Les protéines de référence sont répertoriées dans des bases de données telle que [[Uniprot]]. Or, il existe des séquences codantes alternatives différentes des séquences codantes de référence<ref>{{article|auteur1=B. Vanderperre |auteur2=J.F. Lucier |auteur3=C. Bissonnette |auteur4=J. Motard |auteur5=G. Tremblay |auteur6=S. Vanderperre |auteur7=M. Wisztorski |auteur8=M. Salzet |auteur9=F.M. Boisvert |auteur10=X. Roucou |titre=Direct detection of alternative open reading frames translation products in human significantly expands the proteome |périodique=PLoS One |date=12 août 2013 |volume=8 |numéro=8 |doi=10.1371/journal.pone.0070698.|lire en ligne=http://www.ncbi.nlm.nih.gov/pubmed/23950983 |PMID=23950983}}</ref>. Ces séquences codantes alternatives codent pour des protéines alternatives. Ainsi, un seul ARNm mature pourrait coder pour plusieurs protéines, et non pas une seule. Le [[protéome]], c'est à dire l'ensemble des protéines exprimées dans une cellule, serait beaucoup plus complexe que le nombre d'ARNm matures.<br /> |
||
[[File:Séquences codantes alternatives.jpg|thumb|Un ARNm mature unique peut produire plusieurs protéines par l'utilisation de séquences codantes (CDS) alternatives.]] |
[[File:Séquences codantes alternatives.jpg|thumb|Un ARNm mature unique peut produire plusieurs protéines par l'utilisation de séquences codantes (CDS) alternatives.]] |
||
Version du 18 décembre 2013 à 23:59
Les protéines alternatives
Les protéines alternatives sont des protéines traduites à partir de séquences codantes alternatives localisées dans les ARNm matures.
Les protéines sont des molécules essentielles à la vie. La synthèse des protéines s'effectue à partir de gènes codant pour des protéines. La synthèse des protéines se fait en deux étapes, la transcription et la traduction. La transcription permet la synthèse d'ARN pré-messagers qui subissent un certain nombre de modifications pour donner des ARNm matures. Dans les ARNm, la région codante ou séquence codante (aussi appelée CDS pour Coding DNA Sequence), code pour une protéine spécifique. Chez les eukaryotes, le dogme actuel de la biologie moléculaire associe une seule séquence codante à chaque ARNm mature, et donc une seule protéine. Cette séquence codante est appelée séquence codante de référence et la protéine correspondante est appelée protéine de référence. Les protéines de référence sont répertoriées dans des bases de données telle que Uniprot. Or, il existe des séquences codantes alternatives différentes des séquences codantes de référence[1]. Ces séquences codantes alternatives codent pour des protéines alternatives. Ainsi, un seul ARNm mature pourrait coder pour plusieurs protéines, et non pas une seule. Le protéome, c'est à dire l'ensemble des protéines exprimées dans une cellule, serait beaucoup plus complexe que le nombre d'ARNm matures.
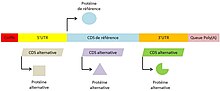
Les séquences codantes alternatives peuvent être localisées dans différentes régions de l'ARNm. Elle peuvent être présentes dans les trois cadres de lecture[2] des régions non traduites ou 5'UTR (de l'anglais 5' UnTranslated Region) et 3'UTR (de l'anglais 3' UnTranslated Region), ou bien chevaucher la séquence codante de référence dans les cadres de lecture +2 et +3. En effet, par définition, la séquence codante de référence est située sur le cadre de lecture +1.
Protéines alternatives et diversité protéique
Chez les eukaryotes, l'épissage alternatif permet la production de plusieurs ARNm matures à partir d'un seul ARN pré-messager, et les séquences codantes peuvent varier entre les différents ARNm matures. C'est ainsi qu'un gène peut coder pour plusieurs protéines. Cependant, même si les protéines produites à partir d'un gène peuvent être différentes, elles conservent entre elles des grandes régions identiques et on les qualifie d'isoformes. Les protéines alternatives sont entièrement différentes des protéines de référence et ne sont donc pas des isoformes des protéines de référence[3],[4]
Protéines alternatives détectées
Avant l'utilisation de la protéomique, peu de protéines alternatives humaines ont été détectées[5]. Certaines sont associées à des gènes impliqués dans des maladies neurodégénératives[6],[7]. La protéomique permet désormais de détecter l'expression des protéines alternatives à grande échelle[8],[9],[10].
Notes et références
- B. Vanderperre, J.F. Lucier, C. Bissonnette, J. Motard, G. Tremblay, S. Vanderperre, M. Wisztorski, M. Salzet, F.M. Boisvert et X. Roucou, « Direct detection of alternative open reading frames translation products in human significantly expands the proteome », PLoS One, vol. 8, no 8, (PMID 23950983, DOI 10.1371/journal.pone.0070698., lire en ligne)
- Alberts B, Johnson A, Lewis J, et al., "From RNA to protein", Molecular Biology of the Cell. 4th edition. New York: Garland Science; 2002.
- Vanderperre B, Staskevicius AB, Tremblay G, McCoy M, O'Neill MA, Cashman NR, Roucou X. An overlapping reading frame in the PRNP gene encodes a novel polypeptide distinct from the prion protein, FASEB J. 2011 Jul;25(7):2373-86. doi: 10.1096/fj.10-173815. PMID: 19014979.
- Bergeron D, Lapointe C, Bissonnette C, Tremblay G, Motard J, Roucou X. An out-of-frame overlapping reading frame in the ataxin-1 coding sequence encodes a novel ataxin-1 interacting protein, J Biol Chem. 2013 Jul 26;288(30):21824-35. doi: 10.1074/jbc.M113.472654. PMID: 23760502.
- Abramowitz J, Grenet D, Birnbaumer M, Torres HN, Birnbaumer L. XLalphas, the extra-long form of the alpha-subunit of the Gs G protein, is significantly longer than suspected, and so is its companion Alex, Proc Natl Acad Sci U S A. 2004 Jun 1;101(22):8366-71. PMID: 15148396.
- Vanderperre B, Staskevicius AB, Tremblay G, McCoy M, O'Neill MA, Cashman NR, Roucou X. An overlapping reading frame in the PRNP gene encodes a novel polypeptide distinct from the prion protein, FASEB J. 2011 Jul;25(7):2373-86. doi: 10.1096/fj.10-173815. PMID: 19014979.
- Bergeron D, Lapointe C, Bissonnette C, Tremblay G, Motard J, Roucou X. An out-of-frame overlapping reading frame in the ataxin-1 coding sequence encodes a novel ataxin-1 interacting protein, J Biol Chem. 2013 Jul 26;288(30):21824-35. doi: 10.1074/jbc.M113.472654. PMID: 23760502.
- Vanderperre B, Lucier JF, Bissonnette C, Motard J, Tremblay G, Vanderperre S, Wisztorski M, Salzet M, Boisvert FM, Roucou X. Direct detection of alternative open reading frames translation products in human significantly expands the proteome, PLoS One. 2013 Aug 12;8(8):e70698. doi: 10.1371/journal.pone.0070698. PMID: 23950983
- Oyama M, Kozuka-Hata H, Suzuki Y, Semba K, Yamamoto T, Sugano S. Diversity of translation start sites may define increased complexity of the human short ORFeome, Mol Cell Proteomics. 2007 Jun;6(6):1000-6. PMID: 17317662.
- Slavoff SA, Mitchell AJ, Schwaid AG, Cabili MN, Ma J, Levin JZ, Karger AD, Budnik BA, Rinn JL, Saghatelian A. Peptidomic discovery of short open reading frame-encoded peptides in human cells, Nat Chem Biol. 2013 Jan;9(1):59-64. doi: 10.1038/nchembio.1120. PMID: 23160002.
