Utilisateur:Neanth/Brouillon
Le Remodelage de la chromatine est la modification dynamique de la structure de la chromatine afin de permettre l'accès de l'ADN génomique condensé aux protéines régulatrices de la machinerie transcriptionnelle, et ainsi contrôler l'expression des gènes. Ce remodelage est principalement assuré par 1) La modification covalente des histones grâce à des enzymes spécifiques, par exemple, des histones acétyltransférases (HATs), désacétylases, méthyltransférases, et kinases, et 2) des complexes de remodelage de la chromatine ATP-dépendants qui peuvent soit déplacer, éjecter ou restructurer les nucléosomes.[1] Outre la régulation active de l'expression des gènes, le remodelage dynamique de la chromatine est impliqué dans la régulation épigénétique de plusieurs processus biologiques clés, comme par exemple la réplication et la réparation de l'ADN cellulaire; l'apoptose; la ségrégation des chromosomes ainsi que le développement et la pluripotence. Les aberrations dans les protéines de remodelage de la chromatine sont associées à des maladies humaines, notamment le cancer. Le ciblage des voies de remodelage de la chromatine devient une cible thérapeutique majeure dans le traitement de plusieurs cancers.
Introduction[modifier | modifier le code]
La régulation transcriptionnelle du génome est contrôlée principalement lors de l'étape de pré-initiation via la liaison aux protéines de la machinerie transcriptionnelle (à savoir, l'ARN polymérase, les facteurs de la transcription, et les activateurs et répresseurs) au niveau de la séquence promotrice principale sur la région codante de l'ADN. Cependant, l'ADN est fermement compacté dans le noyau grâce à l'aide de protéines de compaction, principalement des protéines d'histones afin de former des unités répétées de nucléosomes qui se regrouperont par la suite pour former une structure chromatinienne condensée. Une structure aussi condensée occulte plusieurs régions régulatrices de l'ADN, les empêchant alors d'interagir avec les les protéines de la machinerie transcriptionnelle et de réguler l'expression des gènes. Afin de surmonter ce problème et de permettre un accès dynamique à l'ADN condensé, un processus connu sous le nom de remodelage de la chromatine altère l'architecture des nucléosomes afin d'exposer ou de cacher des régions de l'ADN afin de permettre une régulation transcriptionnelle.
Par définition, le remodelage de la chromatine est le processus qui recourt à l'assistance d'enzymes afin de faciliter l'accès à l'ADN nucléosomique via le remodelage de la structure, la composition et le positionnement des nucléosomes.
Classification[modifier | modifier le code]
L'accès à l'ADN nucléosomique est régi par deux classes majeures de complexes protéiques:
- Les complexes modifiant les histones de façon covalente.
- Les complexes remodelant la chromatine dépendants de l'ATP.
Les complexes modifiant les histones de façon covalente[modifier | modifier le code]
Des complexes spécifiques, connus sous le nom de modificateurs d'histones catalysent l'addition ou la suppression de plusieurs groupements chimiques sur les histones. Ces modifications enzymatiques incluent l'acétylation, la méthylation, la phosphorylation, et l'ubiquitination et ont lieu principalement à l'extrémité N-terminale des queues d'histones. Ces modifications affectent l'affinité de liaison entre les histones et l'ADN, et par ce fait relâchent ou resserrent l'ADN condensé qui est enroulé autour des histones, par exemple, la méthylation de certains résidus spécifiques de lysine en H3 et H4 provoquent une augmentation de la condensation de l'ADN autour des histones, et par ce fait empêchent la liaison de facteurs de transcription sur l'ADN ce qui mène à la répression du gène. A l'opposé, l'acétylation des histones relâche la chromatine et expose l'ADN à la liaison des facteurs de transcription, menant alors à une augmentation du niveau d'expression.[4]
Modifications connues[modifier | modifier le code]
Les modification bien connues d'histones incluent:[5]
La lysine et l'arginine sont connus pour être méthylés. Les lysines méthylées sont les marques du code d'histone les mieux comprises, puisqu'une méthylation spécifique de la lysine correspondent assez bien aux statuts de l'expression génique. La méthylation des H3K4 et H3K36 sont corrélées à une activation transcriptionnelles tandis que la déméthylation de H3K4 est corrélée avec le silencing de la région génomique. La méthylation des lysines H3K9 et H3K27 est corrélée à une répression transcriptionnelle.[6] En particulier, H3K9me3 est étroitement corrélée à de l'hétérochromatine constitutive.[7]
- Acétylation - par des HAT (histone acétyl transférase); désacetylation - par des HDAC (histone désacetylase)
L'acétylation tend à caractériser l' 'ouverture' de la chromatine puisque les histones acétylées ne peuvent pas se rassembler aussi bien que les histones désacétylées..
Cependant, il existe beaucoup plus de modifications d'histones, et les approches sensitives comme la spectrométrie de masse ont récemment grandement étendu le catalogue des modifications.[8]
L'hypothèse du code d'histone[modifier | modifier le code]
Le code d'histone est l'hypothèse que la transcription de l'information génétique encodée dans l'ADN est en partie régulée par les modifications chimiques des protéines d'histones, principalement sur leurs extrémités non-structurées. Avec la participation de modifications similaires telles que la méthylation de l'ADN cela fait partie du code épigénétique.
Un ensemble de preuves suggère qu'un tel code est écrit par des enzymes spécifiques qui peuvent (par exemple) méthyler ou acétyler l'ADN ('writers'), effacé par d'autres enzymes qui ont une activité déméthylase ou désacétylase ('erasers') , et finalement identifiées puis lues par des protéines ('readers') qui sont recrutées sur ces modifications d'histones et se lient via des domaines spécifiques, par exemple, bromodomaine, chromodomaine. Cette triple action de ‘writing’(écriture), ‘reading’ (lecture) et ‘erasing’ (effacement) établit un environnement local favorable pour la régulation transcriptionnelle, la réparation des dommages liés à l'ADN, etc.[9]
Le concept critique de l'hypothèse du code d'histone est que les modifications d'histones servent à recruter d'autres protéines par une reconnaissance spécifique des histones modifiées via des domaines protéiques spécialisés dans ce genre de but, plutôt qu'au travers d'une simple stabilisation ou déstabilisation de l'interaction entre l'histone et l'ADN sous-jacent. Ces protéines recrutées agissent alors pour altérer la structure de la chromatine activement ou pour promouvoir la transcription.
Un bref résumé du code d'histone pour le statut d'expression génique est donné ci-dessous (la nomenclature des histones est décrite ici):
| Type de modification |
Histone | ||||||
|---|---|---|---|---|---|---|---|
| H3K4 | H3K9 | H3K14 | H3K27 | H3K79 | H4K20 | H2BK5 | |
| mono-méthylation | activation[10] | activation[11] | activation[11] | activation[11][12] | activation[11] | activation[11] | |
| di-méthylation | repression[6] | répression[6] | activation[12] | ||||
| tri-méthylation | activation[13] | répression[11] | répression[11] | activation,[12] répression[11] |
répression[6] | ||
| acétylation | activation[13] | activation[13] | |||||
Remodelage de la chromatine dépendant de l'ATP[modifier | modifier le code]
Les complexes remodeleurs de la chromatine dépendants de l'ATP régulent l'expression des gènes soit en déplaçant, éjectant ou restructurant les nucléosomes. Ces complexes protéiques ont un domaine ATPase commun, l'énergie issue de l'hydrolyse de l'ATP permet à ces complexes remodeleurs de repositionner les nucléosomes (souvent mentionnés en tant que "nucleosome sliding" en anglais) le long de l'ADN, éjecter ou assembler des histones sur/hors de l'ADN ou facilite l'échange des variants d'histones, et de ce fait créent des régions d'ADN nu afin d'activer les gènes.[14] De plus, plusieurs remodeleurs ont une activité ADN-translocation pour effectuer des tâches de remodelage spécifiques.[15]
Tous les complexes remodeleurs de la chromatine dépendants de l'ATP possèdent une sous-unité ATPase qui appartient à la superfamille des protéines SNF2. En association avec l'identité de la sous-unité, deux groupes principaux ont été classifiés pour ces protéines. Ils sont connus en tant que groupe SWI2/SNF2 et groupe d'imitation SWI (ISWI). La troisième classe de complexe ATP-dépendant qui a été récemment décrite contient une ATPase Snf2-like et exprime également une activité désacétylase.[16]
Remodeleurs de la chromatine connus[modifier | modifier le code]
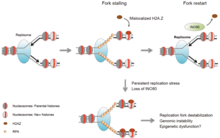
Il y a au moins cinq familles de remodeleurs de la chromatine chez les eucaryotes: SWI/SNF, ISWI, NuRD/Mi-2/CHD, INO80 et SWR1 dont les deux premiers remodeleurs ont été très bien étudiés jusqu'à présent, en particulier chez l'organisme modèle de la levure. Bien que tous les remodeleurs partagent un domaine ATPase commun, leurs fonctions sont spécifiquement basées sur plusieurs processus biologiques (réparation de l'ADN, apoptose, etc.). Ceci est du au fait que chaque complexe remodeleur a des domaines protéiques uniques (Helicase, bromodomaine, etc.) dans leur région catalytique ATPase et a également des sous-unités recrutées différentes.
Fonctions spécifiques[modifier | modifier le code]
- Plusieurs expériences in-vitro suggèrent que les remodeleurs ISWI permettent d'organiser correctement les nucléosomes sous forme de faisceaux et créent un espacement égal entre les nucléosomes, tandis que les remodeleurs SWI / SNF perturbent les nucléosomes.
- Les remodeleurs de la famille ISWI ont montré qu'ils jouent un rôle central dans l'assemblage de la chromatine après la réplication de l'ADN pour permettre le maintien de structures chroatieniennes de haut niveau d'organisation.
- Les familles de remodeleurs INO80 et SWI/SNF participent à la réparation des coupures double brin de l'ADN et à la réparation des nucléotides excisés et de ce fait jouent un rôle crucial dans la réponse aux lésions de l'ADN médiée par TP53.
- Les complexes remodeleurs NuRD/Mi-2/CHD médient principalement la répression transcriptionnelle dans le noyau et sont requis pour le maintient de la pluripotence dans les cellules souches embryonnaires.[14]
Importance[modifier | modifier le code]
Dans les processus biologiques normaux[modifier | modifier le code]
Le remodalge de la chromatine joue un rôle central dans la régulation de l'expression des gènes en apportant la machinerie transcriptionnelle avec un accès dynamique au génome qui serait autrement fermement compacté. De plus, le déplacement des nucléosomes par les remodeleurs de la chromatine est essentiel à plusieurs processus biologiques importants, notamment l'assemblage des chromosomes et la ségrégation, la réplication et la réparation de l'ADN, le développement embryonnaire et la pluripotence, et la progression du cycle cellulaire. La dérégulation du remodelage de la chromatine provoque une perte de la régulation transcriptionnelle à ces check-point critiques qui sont requis pour des fonctions cellulaires normales, et de fait provoque de nombreuses syndromes de pathologies, notamment le cancer.
Réponse aux dommages de l'ADN[modifier | modifier le code]
Le relâchement de la chromatine est l'une des réponses les plus précoces aux lésions de l'ADN.[17]
Le relâchement serait initié par PARP1, dont l'accumulation au niveau de la lésion de l'ADN est à moitié complète au bout de 1.6 secondes après que les dommages aient eu lieu.[18] L'accumulation des remodeleurs de chromatine Alc1 y fait rapidement suite, il possède un domaine de liaison à l'ADP-ribose, lui permettant d'être rapidement attiré au produit de PARP1. Le recrutement maximum de Alc1 a lieu dans les 10 secondes suivant la lésion de l'ADN.[17] A peu près la moitié du maximum de relâchement de la chromatine, supposément grâce à l'action Alc1, a lieu dans les 10 seconds.[17] L'action de PARP1 at au site de la cassure double brin permet le recrutement de deux enzymes de réparation de l'ADN MRE11 et NBS1. La moitié du recrutement maximum de ces deux enzymes de réparation de l'ADN prend 13 secondes pour MRE11 et 28 secondes pour NSB1.[18]
Un autre processus de relâchement de la chromatine, après la formation d'une cassure double brin de l'ADN, recourt à l'utilisation de yH2AX qui est la forme phosphirylée de la protéineH2AX. Les variants d'histone H2AX constituent environ 10% des histones H2A de la chromatine humaine.[19] Lors d'une expérience γH2AX (phosphorylé sur la serine 139 de H2AX) est détecté au bout 20 secondes après l'irradiation des cellules (avec la formation d'une cassure double brin de l'ADN), et la moitié de l'accumulation maximum de γH2AX a eu lieu en une minute.[19] L'étendue de la chromatine associée au γH2AX phosphorylé est d'environ deux millions de paires de bases depuis le site d'une cassure double brin de l'ADN. [19]
γH2AX ne cause pas la décondensation de la chromatine de lui-même, mais dans les quelques secondes qui suivent l'irradiation la protéine “Mediator of the DNA damage checkpoint 1” (MDC1) s'attache spécifiquement à γH2AX.[20][21] Cela s'accompagne simultanément d'une accumulation de la protéine RNF8 et de la protéine NBS1 qui est impliquée dans la réparation de l'ADN et qui se lie à MDC1 puisque MDC1 s'attache à γH2AX.[22] RNF8 médie une décondensation extensive de la chromatine, au travers de son interactions subséquente avec la protéine CHD4,[23] un composant du complexe remodeleur à activité désacétylase NuRD. L'accumulation de CHD4 sur le site de la cassure double brin est rapide, la moitié du maximum de l'accumulation est atteinte environ 40 secondes après l'irradiation.[24]
Le relâchement initial rapide de la chromatine lors d'une lésion de l'ADN (avec une initiation rapide de la réparation de l'ADN) est suivi d'un recondensation lente, avec de la chromatine qui recouvre un état de compaction proche de son niveau avant la lésion en à peu près 20min.[17]
Cancer[modifier | modifier le code]
Le remodelage de la chromatine procure une régulation fine de la croissance cellulaire et des étapes de division, comme la progression du cycle cellulaire, la réparation de l'ADN et la ségrégation des chromosomes, et de ce fait exerce une fonction de répresseur des tumeurs. Les mutations qui ont lieu dans ces remodeleurs de la chromatine et la dérégulation des modifications covalentes d'histones peuvent potentiellement favoriser l'auto-suffisance dans la croissance cellulaire et permettre d’échapper aux signaux régulateurs cellulaires de la croissance - ce sont deux caractéristiques importantes du cancer.[25]
- La désactivation des mutations dans SMARCB1, qui était précédemment connus sous le nom de hSNF5/INI1 et d'un composant du complexe remodeleur SWI/SNF humain ont été trouvés dans un grand nombre de tumeurs rhaboïdes, affectant couramment la population pédiatrique.[26] Des mutations similaires sont aussi présentes dans d'autres cancers juvéniles, comme le carcinome du plexus choroïde, medulloblastome et dans certaines leucémies aiguës. De plus, des études sur des souris KO soutiennent fortement que SMARCB1 est une protéine répressive des tumeurs. Depuis l'observation originelle des mutations de SMARCB1 dans les tumeurs rhabdoïdes, plusieurs sous-unités supplémentaires du complexe remodeleur de la chromatine SWI/SNF humain ont été retrouvées mutées dans une vaste étendue de néoplasmes.[27]
- L'ATPase SWI/SNF ATPase BRG1 (or SMARCA4) du complexe remodeleur est celle qui est mutée le plus fréquement dans le cancer.[28] Les mutations dans ce gène ont été reconnues initialement dans les cellules cancéreuses humaines dérivées de la glande surrénale[29] et du poumon.[30] Dans le cancer, les mutations dans BRG1 montrent une préférence anormalement haute pour les mutations faux-sens qui ciblent le domaine ATPase.[31][28] Les mutations sont enrichies sur les séquences ATPase hautement conservées,[32] qui reposent sur des surfaces fonctionnelles importantes telles que la poche ATP ou la surface de liaison à l'ADN.[31] Ces mutations agissent de façon génétiquement dominante pour altérer les fonctions régulatoires aux enhancers[31] et aux promoteurs.[32]
- La protéine de fusion PML-RAR dans les leucémies aigües myéloïdes recrute des histone désacetylases. Cela mène à la répression des gènes responsables de la différenciation chez les myélocytes et conduit alors à une leucémie.
- La protéine suppressive de tumeurs Rb agit grâce au recrutement des enzymes BRG1 homologues humaines de SWI/SNF, de l'histone désacetylase et de l'ADN méthyltransferase. Les mutations dans BRG1 sont rapportés dans plusieurs cancers, causant la perte de l'action suppressive de tumeurs de Rb.[33]
- Des publications récentes indiquent une hyperméthylation de l'ADN dans la régions promotrice de gènes répresseurs des tumeurs majeurs dans plusieurs cancers. Bien qu'il n'y ait toujours que peu de mutations qui aient été rapportées dans les histones méthylatransférases, la corrélation entre l'hyperméthylation de l'ADN et la méthylation de l'histone H3 lysine-9 a été rapportée dans plusieurs cancers, principalement dans les cancers du sein et colorectaux.
- Les mutations dans l'Histone Acétyl Transférases (HAT) p300 (faux-sens et de type tronqué) sont les les plus couramment rapportées dans les carcinomes colorectaux, pancréatiques, du sein et gastriques. La perte de l'état hétérozygote dans la région codante de p300 (chromosome 22q13) est présente dans un grand nombre de glioblastomes.
- De plus, les HATs ont différents rôles tels qu'être des facteurs de transcription, en plus d''avoir une activité histone acétylase, par exemple, la sous-unité HAT, hADA3 peut agir comme une protéine adaptatrice liant les facteurs de transcription avec d'autres complexes HAT. En l'absence de hADA3, TP53 l'activité transcriptionnelle est significativement réduite, suggérant le rôle de hADA3 dans l'activation la fonction de TP53 en réponse à une lésion de l'ADN.
- De même, TRRAP, l'homologue humain de Tra1 chez la levure, a été montré pour interagir directement avec c-Myc et E2F1 - qui sont des oncoprotéines connues.
Génomique du cancer[modifier | modifier le code]
Des avancées rapides dans l'oncogénétique et des méthodes à haut-débit ChIP-chip, ChIP-Seq et le séquençage au bisulfite fournissent plus d'informations sur le rôle du remodelage de la chromatine dans la régulation transcriptionnelle et son rôle dans le cancer.
Intervention thérapeuthique[modifier | modifier le code]
L'instabilité génétique causée par la dérégulation du remodelage de la chromatine est étudiée dans plusieurs cancers, notamment le cancer du sein, colorectal et pancréatique. Une telle instabilité cause largement une grande étendue du silencing des gènes avec un impact primaire sur les gènes répresseurs des tumeurs. Par conséquent, des stratégies sont maintenant essayées pour surpasser le silencing épigénétique avec une combinaison synergique des HDAC inhibitors ou HDI et agents de déméthylation de l'ADN. Les HDIs sont primairement utilisée en tant que thérapie d'appoint dans plusieurs types de cancers.[34][35] Les inhibiteurs HDAC peuvent induire l'expression de p21 (WAF1), un régulateur de l'activité suppresseur de tumeur de p53. Les HDACs sont impliqués dans la voie par la quelle les protéines du rétinoblastome (pRb) répriment la croissance cellulaire.[36] Les œstrogènes sont des facteur mitogène bien établis, impliqués dans la tumerogénèse et la progression du cancer du sein via sa liaison au estrogen receptor alpha (ERα). Des données récentes indiquent le l'inactivation de la chromatine médiée par HDAC et la méthylation de l'ADN est un composant critique du silencing de ERα silencing dans les cellules du cancer du sein humain.[37]
- Utilisation approuvée:
- Vorinostat a été autorisé par l'U.S. FDA en Octobre 2006 pour le traitement du lymphomes cutanés à cellules T (CTCL).
- Romidepsin (nom commercial Istodax) a été autorisé par l'US FDA en Nov 2009 pour les lymphomes cutanés à cellules T (CTCL).
- Phase III Essais cliniques:
- Panobinostat (LBH589) est en essais clinique pour plusieurs cancers notamment en essai clinique de phase III trial pour les lymphomes cutanés à cellules T (CTCL).
- Acide valproïque (en tant que Mg valproate) est en phase III d'essais cliniques pour le cancer cervical et le cancer ovarien.
- Début des essais cliniques pivots de phase II
- Belinostat (PXD101) a eu une phase II d'essai pour les cancers ovariens récidivants, et a montré de bons résultats pour le lymphomes cutanés à cellules T.
- inhibiteurs de HDAC
Les candidats pionniers actuels pour de nouvelles cibles médicamenteuses sont l'Histone Lysine Methyltransferases (KMT) et la protéine Arginine Méthyltransférases (PRMT).[38]
Autres syndromes pathologiques[modifier | modifier le code]
- Le syndrome ATRX (α-thalassémie/retard mental lié à l'X) et le syndrome d'α-thalassémie/myélodysplasie sont causés par des mutations dans ATRX, une ATPase liée à SNF2 avec un PHD.
- Le syndrome CHARGE, un trouble autosomique dominant, a été récemment lié à une haploinsuffisance de CHD7, qui code pour l'ATPase CHD7 qui appartient à la CHD family.[39]
Senescence[modifier | modifier le code]
Le remodelage architectural de la chromatine est impliqué dans le processus de senescence cellulaire, qui est lié au vieillissement de l'organisme, bien que distinct de celui-ci. La sénescence cellulaire réplicative fait référence à un arrêt permanent du cycle cellulaire où les cellules post-mitotiques continuent d'exister en tant que cellules métaboliquement actives mais échouent à proliférer.[40][41] La senescence peut survenir suite à une dégradation associée à l'âge, une attrition des télomères, des progerias, des pre-malignancies, et d'autres formes de dommages ou pathologies. Les cellules senescentes subissent des changements phénotypiques répressifs distincts, potentiellement pour prévenir la prolifération des cellules endommagées ou cancéreuses, avec une modification de l'organisation de la chromatine, des fluctuations dans l'abdondance des remodeleurs, et des changements dans les modifications épigénétiques.[42][43][40] Les cellules senescentes subissent une modifications des territoires chromatiniens étant donné que l'hétérochromatine migre vers le centre du noyau et déplace l'euchromatine et l'hétérocrhomatine facultative vers des regions en périphérie du noyau. Cela perturbe les intéractions chromatine-lamine et des inversions du motif généralement observé dans une cellule mitotiquement active.[44][42] Les domaines individuels associés à la lamine (Individual Lamina-Associated Domains) (LADs) et les domaines TAD (Topologically Associating Domains) (TADs) sont perturbés par cette migration qui peut affecter les interactions cis le long du génome.[45] De plus, il existe un schéma général de perte d'histones canoniques, particulièrement pour les histones des nucléosomes H3 and H4 et l'histone linker H1.[44] Les variants d'histones avec deux exons sont up-régulés chez les cellules senescentes pour produire un assemblage modifié des nucléosomes qui contribue à la permissivité de la chromatine aux changements liés à la senescence.[45] Bien que la transcription des protéines des variants d'histone peut être élevé, les protéines canoniques d'histones ne sont pas exprimées puisqu'elles sont seulement produite lors de la phase S du cycle cellulaire et les cellules senescentes sont post-mitotiques.[44] Lors de la senescence, des portions des chromosomes peuvent être exportées depuis le noyau pour la dégradation lysosomale qui résulte en une plus grande organisation un plus grand désordre organisationnel et une perturbation des interactions de la chromatine.[43]
L'abondance des remodeleurs de la chromatine peut être impliquée dans le senescence cellulaire puisqu'un knockdown ou un knockout de remodeleurs ATP-dépendant comme NuRD, ACF1, et SWI/SNP peut résulter en dommages de l'ADN et un phénotype senescent chez la levure, C. elegans, la souris, et les cultures de cellules humaines.[46][43][47] ACF1 et NuRD sont downregulés chez les cellules senescentes ce qui suggère que le remodelage de la chromatine est essentielle pour maintenir un phénotype mitotique.[46][47] Les gènes impliqués dans la signalisation de la senescence peuvent être réprimés par conformation de la chromatine et des complexes polycomb répressifs comme vu dans le silencing de PRC1/PCR2 sur p16.[48][49] Une déplétion spécifique des remodeleurs donne lieu à une inactivation des gnes de prolifération au travers d'un échec du maintien du silencing.[43] Certains remodeleurs agissent sur les régions enhancer des genes plutôt que sur les loci spécifiques afin de prévenir la ré-entrée dans le cycle cellulaire en formant des régions d'hétérochromatine dense autour des régions régulatrices.[49]
Les cellules senescentes subissent des fluctuations étendues de modifications épigénétiques dans des régions de chromatine spécifiques par rapport aux cellules mitotiques. Les cellules humaines et murines subissant une senescence réplicative expérimentent une diminution globale globale de la méthylation; cependant, des loci spécifiques peuvent différent de la tendance générale.[50][45][43][48] Des régions spécifiques de chromatine, en particulier celles autour des promoteurs ou des enhancers des loci prolifératifs, peuvent montrer des états de méthylations élevés avec un déséquilibre global des modifications répressives et activatrices des histones.[42] Les gènes prolifératifs peuvent montrer des augmentations de la marque répressive H3K27me3 tandis que les gènes impliqués dans le silencing ou la production d'histones aberrants peut être enrichi en modification activatrice H3K4me3.[45] De plus, l'up-régulation des histone deacetylases, tels que les membres de la famille sirtuin, peut retarder la senescence en supprimant les groupements acétyls qui contribuent à un meilleur assemblage de la chromatine.[51] La perte générale de la méthylation, combinée avec l'addition des groupements acétyls résulte en une conformation de la chromatine plus accessible avec une propension tournée vers la désorganisation lors d'une comparaison avec des cellules mitotiquement actives.[43] La perte générale des histones empêche l'ajout de modifications d'histones et contribue à des changements d'enrichissement dans certaines régions de la chromatine pendant la sénescence..[44].[44]
Voir aussi[modifier | modifier le code]
- Epigénétique
- Histone
- Nucléosomes
- Chromatine
- Histone acetyltransferase
- Transcription factors
- CAF-1 (Chromatin assembly factor-1) - une chaperonne d'histone qui exécute un rôle de coordination dans le remodelage de la chromatine.
Références[modifier | modifier le code]
- « Predicting nucleosome positions on the DNA: combining intrinsic sequence preferences and remodeler activities », Nucleic Acids Research, vol. 37, no 17, , p. 5641–55 (PMID 19625488, PMCID 2761276, DOI 10.1093/nar/gkp610)
- (en) Timothy J. Richmond, Karolin Luger, Armin W. Mäder et Robin K. Richmond, « Crystal structure of the nucleosome core particle at 2.8 A resolution », Nature, vol. 389, no 6648, , p. 251–260 (DOI 10.1038/38444, lire en ligne)
- Boyer, « The chromatin signature of pluripotent cells », Stembook, (PMID 20614601, DOI 10.3824/stembook.1.45.1)
- « Chromatin remodeling and cancer, Part I: Covalent histone modifications », Trends in Molecular Medicine, vol. 13, no 9, , p. 363–72 (PMID 17822958, DOI 10.1016/j.molmed.2007.07.003)
- « The language of covalent histone modifications », Nature, vol. 403, no 6765, , p. 41–5 (PMID 10638745, DOI 10.1038/47412, Bibcode 2000Natur.403...41S)
- « Determination of enriched histone modifications in non-genic portions of the human genome », BMC Genomics, vol. 10, , p. 143 (PMID 19335899, PMCID 2667539, DOI 10.1186/1471-2164-10-143)
- Philip Hublitz, Mareike Albert et Antoine Peters, « Mechanisms of Transcriptional Repression by Histone Lysine Methylation », The International Journal of Developmental Biology, vol. 10, no 1387, , p. 335–354 (ISSN 1696-3547)
- « Identification of 67 histone marks and histone lysine crotonylation as a new type of histone modification », Cell, vol. 146, no 6, , p. 1016–28 (PMID 21925322, PMCID 3176443, DOI 10.1016/j.cell.2011.08.008)
- « Translating the histone code », Science, vol. 293, no 5532, , p. 1074–80 (PMID 11498575, DOI 10.1126/science.1063127, CiteSeerx 10.1.1.453.900)
- « Histone H3K4 demethylases are essential in development and differentiation », Biochemistry and Cell Biology, vol. 85, no 4, , p. 435–43 (PMID 17713579, DOI 10.1139/o07-057)
- « High-resolution profiling of histone methylations in the human genome », Cell, vol. 129, no 4, , p. 823–37 (PMID 17512414, DOI 10.1016/j.cell.2007.05.009)
- « DOT1L/KMT4 recruitment and H3K79 methylation are ubiquitously coupled with gene transcription in mammalian cells », Molecular and Cellular Biology, vol. 28, no 8, , p. 2825–39 (PMID 18285465, PMCID 2293113, DOI 10.1128/MCB.02076-07)
- « The landscape of histone modifications across 1% of the human genome in five human cell lines », Genome Research, vol. 17, no 6, , p. 691–707 (PMID 17567990, PMCID 1891331, DOI 10.1101/gr.5704207)
- « Chromatin remodeling and cancer, Part II: ATP-dependent chromatin remodeling », Trends in Molecular Medicine, vol. 13, no 9, , p. 373–80 (PMID 17822959, PMCID 4337864, DOI 10.1016/j.molmed.2007.07.004)
- « Chromatin remodelling: the industrial revolution of DNA around histones », Nature Reviews Molecular Cell Biology, vol. 7, no 6, , p. 437–47 (PMID 16723979, DOI 10.1038/nrm1945)
- M. Vignali, A. H. Hassan, K. E. Neely et J. L. Workman, « ATP-Dependent Chromatin-Remodeling Complexes », Molecular and Cellular Biology, vol. 20, no 6, , p. 1899–1910 (ISSN 0270-7306, PMID 10688638, PMCID 110808, DOI 10.1128/mcb.20.6.1899-1910.2000, lire en ligne)
- « The poly(ADP-ribose)-dependent chromatin remodeler Alc1 induces local chromatin relaxation upon DNA damage », Mol. Biol. Cell, vol. 27, no 24, , p. 3791–3799 (PMID 27733626, PMCID 5170603, DOI 10.1091/mbc.E16-05-0269)
- « PARP1-dependent kinetics of recruitment of MRE11 and NBS1 proteins to multiple DNA damage sites », J. Biol. Chem., vol. 283, no 2, , p. 1197–208 (PMID 18025084, DOI 10.1074/jbc.M706734200)
- « DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139 », J. Biol. Chem., vol. 273, no 10, , p. 5858–68 (PMID 9488723, DOI 10.1074/jbc.273.10.5858)
- « RNF8 ubiquitylates histones at DNA double-strand breaks and promotes assembly of repair proteins », Cell, vol. 131, no 5, , p. 887–900 (PMID 18001824, DOI 10.1016/j.cell.2007.09.040)
- « MDC1 directly binds phosphorylated histone H2AX to regulate cellular responses to DNA double-strand breaks », Cell, vol. 123, no 7, , p. 1213–26 (PMID 16377563, DOI 10.1016/j.cell.2005.09.038)
- « Phospho-dependent interactions between NBS1 and MDC1 mediate chromatin retention of the MRN complex at sites of DNA damage », EMBO Rep., vol. 9, no 8, , p. 795–801 (PMID 18583988, PMCID 2442910, DOI 10.1038/embor.2008.103)
- « A new non-catalytic role for ubiquitin ligase RNF8 in unfolding higher-order chromatin structure », EMBO J., vol. 31, no 11, , p. 2511–27 (PMID 22531782, PMCID 3365417, DOI 10.1038/emboj.2012.104)
- « The NuRD chromatin-remodeling complex regulates signaling and repair of DNA damage », J. Cell Biol., vol. 190, no 5, , p. 741–9 (PMID 20805320, PMCID 2935570, DOI 10.1083/jcb.201001048)
- « The hallmarks of cancer », Cell, vol. 100, no 1, , p. 57–70 (PMID 10647931, DOI 10.1016/S0092-8674(00)81683-9)
- « Truncating mutations of hSNF5/INI1 in aggressive paediatric cancer », Nature, vol. 394, no 6689, , p. 203–6 (PMID 9671307, DOI 10.1038/28212, Bibcode 1998Natur.394..203V)
- « The spectrum of SWI/SNF mutations, ubiquitous in human cancers », PLOS ONE, vol. 8, no 1, , e55119 (PMID 23355908, PMCID 3552954, DOI 10.1371/journal.pone.0055119, Bibcode 2013PLoSO...855119S)
- « The Many Roles of BAF (mSWI/SNF) and PBAF Complexes in Cancer », Cold Spring Harbor Perspectives in Medicine, vol. 6, no 8, , a026930 (PMID 27413115, PMCID 4968166, DOI 10.1101/cshperspect.a026930)
- « The retinoblastoma protein and BRG1 form a complex and cooperate to induce cell cycle arrest », Cell, vol. 79, no 1, , p. 119–30 (PMID 7923370, DOI 10.1016/0092-8674(94)90405-7)
- « Frequent BRG1/SMARCA4-inactivating mutations in human lung cancer cell lines », Human Mutation, vol. 29, no 5, , p. 617–22 (PMID 18386774, DOI 10.1002/humu.20730)
- « Dominant-negative SMARCA4 mutants alter the accessibility landscape of tissue-unrestricted enhancers », Nature Structural & Molecular Biology, vol. 25, no 1, , p. 61–72 (PMID 29323272, PMCID 5909405, DOI 10.1038/s41594-017-0007-3)
- « Smarca4 ATPase mutations disrupt direct eviction of PRC1 from chromatin », Nature Genetics, vol. 49, no 2, , p. 282–288 (PMID 27941795, PMCID 5373480, DOI 10.1038/ng.3735)
- « Chromatin remodeling: why it is important in cancer », Oncogene, vol. 20, no 24, , p. 2988–90 (PMID 11420713, DOI 10.1038/sj.onc.1204322)
- « Histone deacetylase inhibitors: discovery and development as anticancer agents », Expert Opinion on Investigational Drugs, vol. 14, no 12, , p. 1497–511 (PMID 16307490, DOI 10.1517/13543784.14.12.1497)
- « Histone deacetylase inhibitors: a new class of potential therapeutic agents for cancer treatment », Clinical Cancer Research, vol. 8, no 3, , p. 662–4 (PMID 11895892, lire en ligne)
- « Histone deacetylase inhibitor selectively induces p21WAF1 expression and gene-associated histone acetylation », Proceedings of the National Academy of Sciences of the United States of America, vol. 97, no 18, , p. 10014–9 (PMID 10954755, PMCID 27656, DOI 10.1073/pnas.180316197, Bibcode 2000PNAS...9710014R)
- « Quantitation of HDAC1 mRNA expression in invasive carcinoma of the breast* », Breast Cancer Research and Treatment, vol. 94, no 1, , p. 11–6 (PMID 16172792, DOI 10.1007/s10549-005-6001-1)
- « Toward the development of potent and selective bisubstrate inhibitors of protein arginine methyltransferases », Bioorganic & Medicinal Chemistry Letters, vol. 20, no 7, , p. 2103–5 (PMID 20219369, DOI 10.1016/j.bmcl.2010.02.069)
- « The biology of chromatin remodeling complexes », Annual Review of Biochemistry, vol. 78, , p. 273–304 (PMID 19355820, DOI 10.1146/annurev.biochem.77.062706.153223)
- Aled John Parry et Masashi Narita, « Old cells, new tricks: chromatin structure in senescence », Mammalian Genome, vol. 27, nos 7–8, , p. 320–331 (ISSN 0938-8990, PMID 27021489, PMCID 4935760, DOI 10.1007/s00335-016-9628-9)
- L. Hayflick et P. S. Moorhead, « The serial cultivation of human diploid cell strains », Experimental Cell Research, vol. 25, no 3, , p. 585–621 (ISSN 0014-4827, PMID 13905658, DOI 10.1016/0014-4827(61)90192-6)
- Tamir Chandra, Philip Andrew Ewels, Stefan Schoenfelder, Mayra Furlan-Magaril, Steven William Wingett, Kristina Kirschner, Jean-Yves Thuret, Simon Andrews, Peter Fraser et Wolf Reik, « Global Reorganization of the Nuclear Landscape in Senescent Cells », Cell Reports, vol. 10, no 4, , p. 471–483 (ISSN 2211-1247, PMID 25640177, PMCID 4542308, DOI 10.1016/j.celrep.2014.12.055)
- Luyang Sun, Ruofan Yu et Weiwei Dang, « Chromatin Architectural Changes during Cellular Senescence and Aging », Genes, vol. 9, no 4, , p. 211 (ISSN 2073-4425, PMID 29659513, PMCID 5924553, DOI 10.3390/genes9040211)
- Steven W. Criscione, Yee Voan Teo et Nicola Neretti, « The chromatin landscape of cellular senescence », Trends in Genetics, vol. 32, no 11, , p. 751–761 (ISSN 0168-9525, PMID 27692431, PMCID 5235059, DOI 10.1016/j.tig.2016.09.005)
- Na Yang et Payel Sen, « The senescent cell epigenome », Aging (Albany NY), vol. 10, no 11, , p. 3590–3609 (ISSN 1945-4589, PMID 30391936, PMCID 6286853, DOI 10.18632/aging.101617)
- Jeannine Basta et Michael Rauchman, « The Nucleosome Remodeling and Deacetylase (NuRD) Complex in Development and Disease », Translational Research, vol. 165, no 1, , p. 36–47 (ISSN 1931-5244, PMID 24880148, PMCID 4793962, DOI 10.1016/j.trsl.2014.05.003)
- Xueping Li, Dong Ding, Jun Yao, Bin Zhou, Ting Shen, Yun Qi, Ting Ni et Gang Wei, « Chromatin remodeling factor BAZ1A regulates cellular senescence in both cancer and normal cells », Life Sciences, vol. 229, , p. 225–232 (ISSN 1879-0631, PMID 31085244, DOI 10.1016/j.lfs.2019.05.023)
- Carlos López-Otín, Maria A. Blasco, Linda Partridge, Manuel Serrano et Guido Kroemer, « The Hallmarks of Aging », Cell, vol. 153, no 6, , p. 1194–1217 (ISSN 0092-8674, PMID 23746838, PMCID 3836174, DOI 10.1016/j.cell.2013.05.039)
- Nilgun Tasdemir, Ana Banito, Jae-Seok Roe, Direna Alonso-Curbelo, Matthew Camiolo, Darjus F. Tschaharganeh, Chun-Hao Huang, Ozlem Aksoy, Jessica E. Bolden, Chi-Chao Chen et Myles Fennell, « BRD4 Connects Enhancer Remodeling to Senescence Immune Surveillance », Cancer Discovery, vol. 6, no 6, , p. 612–629 (ISSN 2159-8290, PMID 27099234, PMCID 4893996, DOI 10.1158/2159-8290.CD-16-0217)
- V. L. Wilson et P. A. Jones, « DNA methylation decreases in aging but not in immortal cells », Science, vol. 220, no 4601, , p. 1055–1057 (ISSN 0036-8075, PMID 6844925, DOI 10.1126/science.6844925, Bibcode 1983Sci...220.1055W)
- Matt Kaeberlein, Mitch McVey et Leonard Guarente, « The SIR2/3/4 complex and SIR2 alone promote longevity in Saccharomyces cerevisiae by two different mechanisms », Genes & Development, vol. 13, no 19, , p. 2570–2580 (ISSN 0890-9369, PMID 10521401, PMCID 317077, DOI 10.1101/gad.13.19.2570)
Lectures complémentaires[modifier | modifier le code]
- « Chromatin modifiers and remodellers: regulators of cellular differentiation », Nature Reviews Genetics, vol. 15, no 2, , p. 93–106 (PMID 24366184, PMCID 3999985, DOI 10.1038/nrg3607)
Liens externes[modifier | modifier le code]
- MBInfo - Chromatin
- MBInfo - DNA Packaging
- YouTube - Chromatin, Histones and Modifications
- YouTube - Epigenetics Overview
- (en) MeSH Chromatin+remodeling
Category:Gene expression Category:Cancer Category:Epigenetics Category:Nuclear organization
