Répétition riche en leucine
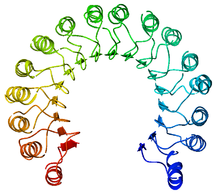


Une répétition riche en leucine, ou leucine-rich repeat (LRR) en anglais, est un motif structurel présent dans certaines protéines où il forme un repliement en fer à cheval α/β[4],[5]. Ce type de repliement est constitué de séquences de 20 à 30 résidus d'acides aminés contenant une forte proportion de leucine, un acide aminé hydrophobe, constituant un domaine en forme de solénoïde. Chaque élément répété est généralement constitué de l'enchaînement feuillet β–coude–hélice α, dont la répétition constitue un domaine qui présente globalement la géométrie d'un fer à cheval dont l'intérieur est formé de feuillets β parallèles et l'extérieur est formé d'un réseau d'hélices α. Une face des feuillets β et un côté des hélices α sont exposés au solvant et sont par conséquent constitués essentiellement de résidus hydrophiles, tandis que la région comprise entre les hélices et les feuillets forme le cœur hydrophobe de la protéine dans lequel sont enfouis les résidus de leucine.
Les répétitions riches en leucine interviennent fréquemment dans l'établissement d'interactions protéine-protéine[6],[7].
Exemples[modifier | modifier le code]
Les motifs LRR ont été identifiés dans un grand nombre de protéines aux fonctions biologiques très diversifiées[8]. L'inhibiteur de ribonucléase en est l'exemple le plus connu, mais d'autres protéines telles que la tropomoduline (en), protéine régulatrice de la tropomyosine, et le récepteur de type Toll possèdent également ce motif structurel. Le récepteur de type Toll possède en fait dix motifs LRR successifs qui interviennent dans la liaison aux motifs moléculaires associés aux agents infectieux.
Si le motif LRR typique contient environ une proportion d'une hélice α pour chaque feuillet β, certaines variantes qui forment des superhélices βα présentent parfois de longues boucles à la place des hélices entre feuillets adjacents. On a également identifié une variante structurelle appelée LRV et constituée d'une alternance d'hélices α et 310 formant une superhélice droite dépourvue des feuillets β habituellement présents dans les répétitions riches en leucines[2].
Domaines associés[modifier | modifier le code]
Les répétitions riches en leucine sont souvent entourés de domaines N-terminaux et C-terminaux riches en cystéine. Ces domaines riches en cystéine existent également en présence de domaines LRR adjacents. Il s'agit de petits domaines entièrement constitués de feuillets β qui ont été structurellement décrits pour les internalines (en) A (InlA), B (InlB), E (InlE) et H (InlH) de la bactérie Listeria monocytogenes. Leur fonction semble être essentiellement structurelle : ils sont fusionnés avec l'extrémité C-terminale des répétitions riches en leucine, ce qui a pour effet de les stabiliser et de les rigidifier. Elles n'interviennent pas directement dans les interactions protéine-protéine mais ajuste le positionnement du domaine LRR adjacent dans ce but. Ces domaines appartiennent à la famille des domaines de type immunoglobuline dans la mesure où ils sont formés de deux feuillets β en sandwich de la même façon que les immunoglobulines ; le brin β de l'un des feuillets est cependant bien plus court que dans les domaines de type immunoglobuline habituels[3],[9],[10].
Un cluster fer-soufre est présent à l'extrémité N-terminale de certaines protéines contenant la variante structurelle LRV riche en leucine. Ces protéines ont une structure en deux domaines : un petit domaine N-terminal contenant un groupe de quatre résidus de cystéine formant un agrégat atomique 4Fe-4S et un grand domaine C-terminal contenant le motif LRV[2]. Des études biochimiques ont montré que l'agrégat 4Fe-4S est sensible à l'oxygène mais ne semble pas porteur d'une activité rédox.
Notes et références[modifier | modifier le code]
- (en) Bostjan Kobef et Johann Deisenhofer, « Mechanism of Ribonuclease Inhibition by Ribonuclease Inhibitor Protein Based on the Crystal Structure of its Complex with Ribonuclease A », Journal of Molecular Biology, vol. 264, no 5, , p. 1028-1043 (PMID 9000628, DOI 10.1006/jmbi.1996.0694, lire en ligne)
- (en) John W. Peters, Michael H.B. Stowell et Douglas C. Rees, « A leucine-rich repeat variant with a novel repetitive protein structural motif », Nature Structural Biology, vol. 3, no 12, , p. 991-994 (PMID 8946850, DOI 10.1038/nsb1296-991, lire en ligne)
- (en) Wolf-Dieter Schubert, Gero Göbel, Meikel Diepholz, Ayub Darji, Daniel Kloer, Torsten Hain, Trinad Chakraborty, Jürgen Wehland, Eugen Domann et Dirk W. Heinz, « Internalins from the human pathogen Listeria monocytogenes combine three distinct folds into a contiguous internalin domain », Journal of Molecular Biology, vol. 312, no 4, , p. 783-794 (PMID 11575932, DOI 10.1006/jmbi.2001.4989, lire en ligne)
- (en) Bostjan Kobe et Johann Deisenhofer, « The leucine-rich repeat: a versatile binding motif », Trends in Biochemical Sciences, vol. 19, no 10, , p. 415-421 (PMID 7817399, DOI 10.1016/0968-0004(94)90090-6, lire en ligne)
- (en) Purevjav Enkhbayar, Masakatsu Kamiya, Mitsuru Osaki, Takeshi Matsumoto et Norio Matsushima, « Structural principles of leucine-rich repeat (LRR) proteins », Proteins: Structure, Function, and Bioinformatics, vol. 54, no 3, , p. 394-403 (PMID 14747988, DOI 10.1002/prot.10605, lire en ligne)
- (en) Bostjan Kobe et Andrey V Kajava, « The leucine-rich repeat as a protein recognition motif », Current Opinion in Structural Biology, vol. 11, no 6, , p. 725-732 (PMID 11751054, DOI 10.1016/S0959-440X(01)00266-4, lire en ligne)
- (en) Nicholas J. Gay, Leonard C. Packman, Michael A. Weldon et Jennifer C.J. Barna, « A leucine-rich repeat peptide derived from the Drosophila Toll receptor forms extended filaments with a β-sheet structure », FEBS Letters, vol. 291, no 1, , p. 87-91 (PMID 1657640, DOI 10.1016/0014-5793(91)81110-T, lire en ligne)
- (en) J. M. Rothberg, J. R. Jacobs, C. S. Goodman et S. Artavanis-Tsakonas, « slit: an extracellular protein necessary for development of midline glia and commissural axon pathways contains both EGF and LRR domains », Genes & Development, vol. 4, no 12A, , p. 2169-2187 (PMID 2176636, DOI 10.1101/gad.4.12a.2169, lire en ligne)
- (en) Wolf-Dieter Schubert, Claus Urbanke, Thilo Ziehm, Viola Beier, Matthias P. Machner, Eugen Domann, Jürgen Wehland, Trinad Chakraborty et Dirk W. Heinz, « Structure of Internalin, a Major Invasion Protein of Listeria monocytogenes, in Complex with Its Human Receptor E-Cadherin », Cell, vol. 111, no 6, , p. 825-836 (PMID 12526809, DOI 10.1016/S0092-8674(02)01136-4, lire en ligne)
- (en) Alexander Freiberg, Matthias P. Machner, Wolfgang Pfeil, Wolf-Dieter Schubert, Dirk W. Heinz et Robert Seckler, « Folding and Stability of the Leucine-rich Repeat Domain of Internalin B from Listeria monocytogenes », Journal of Molecular Biology, vol. 337, no 2, , p. 453-461 (PMID 15003459, DOI 10.1016/j.jmb.2004.01.044, lire en ligne)
