Marqueur fluorescent en PCR
De nombreux marqueurs fluorescents sont utilisés en PCR (réaction en chaîne par polymérase).
Historique[modifier | modifier le code]
Par souci de clarté, les dates correspondent à la première publication sur le domaine et seuls les premiers auteurs sont cités, les références complètes étant dans le chapitre "bibliographie".
- 1880 : Premier milieu de culture synthétique pour micro-organismes fluorescents par Hueppe F.
- 1964 : Détection par fluorescence de l’ADN associé au BET par Lepecq JB.
- 1972 : Première migration d’ADN sur gel d’électrophorèse par Aaij C.
- 1986 : Première publication publique sur la PCR par Kary Mullis (prix Nobel de chimie en 1993).
- 1989 : Première détection de produit de PCR à l’aide du BET (et non par la radioactivité) par Xu ZD.
- 1992 : Invention de la PCR en temps réel par Higuchi R.
- 1994 : Dépôt de brevet du SYBR Green I par Molecular Probes.
- 1995 : Premier emploi du SYBR green pour détecter un produit de PCR par Schneeberger C.
- 1995 : Invention des sondes d’hydrolyses par Livak KJ.
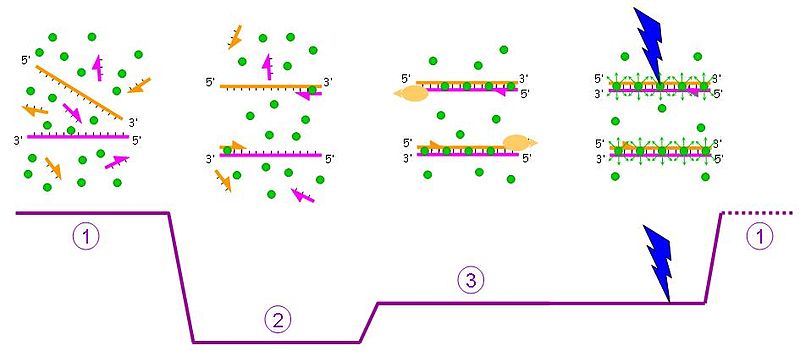
Les marqueurs aspécifiques[modifier | modifier le code]
Les intercalants de l'ADN[modifier | modifier le code]
Les marqueurs du petit sillon[modifier | modifier le code]
Les cyanhydrines asymétriques[modifier | modifier le code]
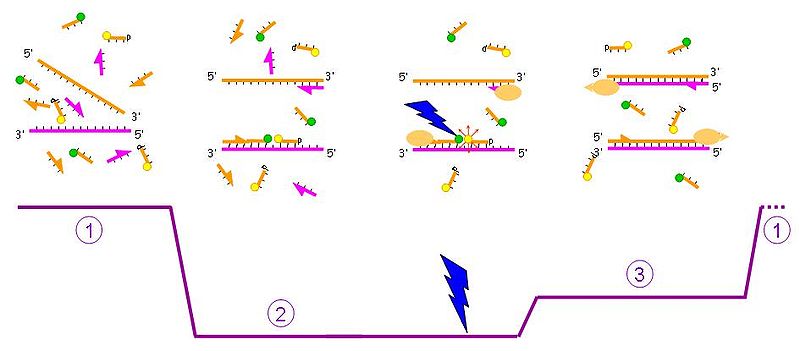
Les marqueurs séquences spécifiques[modifier | modifier le code]
Les sondes d'hybridation[modifier | modifier le code]
Les sondes d'hydrolyses[modifier | modifier le code]
La sonde est construite à l'aide d'un donneur (la fluorescéine dont la longueur d'onde d'excitation est à 530 nm) et d'un "quencheur", greffés sur un polynucléotide complémentaire de la séquence d'ADN d'intérêt.
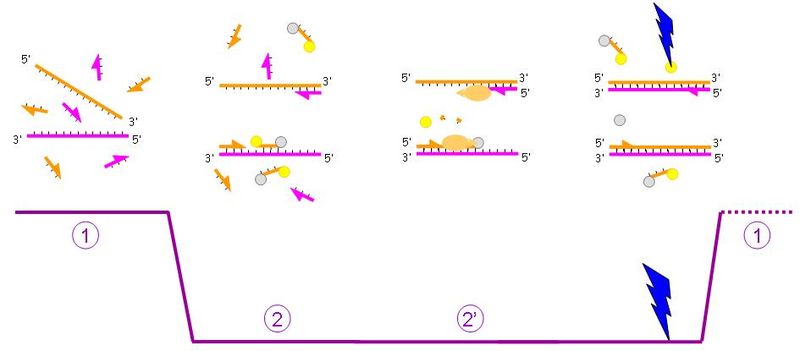
Les sondes Molecular Beacons[modifier | modifier le code]
Les sondes Scorpions[modifier | modifier le code]
Bibliographie[modifier | modifier le code]
Bibliographie francophone[modifier | modifier le code]
- Hueppe F. Etudes sur le lait blue. Cohn's Beiträge zur Biologie der Pflanzen. 1880;3
- Lepecq JB, Yot P, Paoletti C. Interaction entre le bromhydrate d’ethidium (BET) et les acides nucléiques (AN). Etude spectrofluorométrique. [Interaction of ethidium bromhydrate (BET) with nucleic acids (NA). Spectrofluorimetric study.] C R Hebd Seances Acad Sci. 1964;259:1786-9.
- Poitras E, Houde A. La PCR en temps réel: principes et applications. Reviews in Biology and Biotechnology (Canada). 2002;2(2):2-11
- Tsé C, Capeau J. Quantification des acides nucléiques par PCR quantitative en temps réel. Ann Biol Clin (Paris). 2003;61(3):279–93
Bibliographie anglophone[modifier | modifier le code]
- Aaij C, Borst P. The gel electrophoresis of DNA. 1972. Biochim Biophys Acta. May 10;269(2):192-200.
- Higuchi R, Dollinger G, Walsh PS, Griffith R. Simultaneous amplification and detection of specific DNA sequences. 1992. Biotechnology;10:413–7.
- Livak KJ, Flood JA, Marmaro J, Giusti W, Deetz K. Oligonucleotides with fluorescent dyes at opposite ends provide a quenched probe system useful for detecting PCR product and nucleic acid hybridization. 1995. PCR Methods Appl. 4:357–362.
- Mullis K, Faloona F, Scharf S, Saiki R, Horn G, Erlich H. Specific enzymatic amplification of DNA in vitro: the polymerase chain reaction. Cold Spring Harb Symp Quant Biol. 1986;51 Pt 1:263-73.
- Schneeberger C, Speiser P, Kury F, Zeillinger R. Quantitative detection of reverse transcriptase-PCR products by means of a novel and sensitive DNA stain. 1995. PCR Methods Appl. Feb;4(4):234-8.
- Xu ZD, Yu YJ, Hsie AW, Caskey CT, Rossiter B, Gibbs RA. Deletion screening at the hypoxanthine-guanine phosphoribosyltransferase locus in Chinese hamster cells using the polymerase chain reaction. 1989. Teratog Carcinog Mutagen; 9(3):177-87.