Inhibiteur de ribonucléase
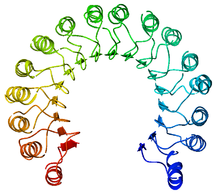

L'inhibiteur de ribonucléase est une protéine d'environ 49 kDa contenant environ 450 résidus d'acides aminés et ayant un point isoélectrique acide voisin de 4,7. C'est une protéine abondante, qui constitue environ 0,1 % de la masse protéique des cellules et qui joue un rôle important dans la régulation de la durée de vie de l'ARN[2]. Elle possède un domaine à répétition riche en leucine (LRR) qui forme des complexes particulièrement étroits avec certaines ribonucléases.
Structure[modifier | modifier le code]
Cette protéine a un taux de cystéine particulièrement élevé d'environ 6,5 %, contre 1,7 % habituellement dans les protéines, et est sensible à l'oxydation. Elle a également un taux élevé en leucine d'environ 21.5 %, au lieu des 9 % constituant généralement les protéines, mais est sensiblement plus pauvre en autres résidus hydrophobes, notamment en valine, isoleucine, méthionine, tyrosine et phénylalanine.
L'inhibiteur de ribonucléase est l'exemple classique des protéines à domaine à répétition riche en leucine dont la structure secondaire alterne hélices α et feuillets β, ce qui l'incurve pour former un solénoïde droit en forme de fer à cheval. Les feuillets β s'organisent parallèlement à l'intérieur de la structure tandis que les hélices α en forment l'extérieur. Cette structure semble stabilisée par des résidus d'asparagine à la base de chaque coude, au niveau de la transition entre hélice α et feuillet β. Ces derniers ont une longueur respective de 28 et 29 résidus, formant une unité structurelle de 57 résidus qui constitue l'unité de base répétée dans l'ensemble du domaine LRR.
Affinité pour les ribonucléases[modifier | modifier le code]
L'affinité de cette protéine pour les ribonucléases est parmi les plus fortes de toutes les interactions protéine-protéine connues. La constante de dissociation du complexe formé par l'inhibiteur de ribonucléase et la ribonucléase pancréatique est d'ordre femtomolaire (1 fM = 10−15 mol L−1) dans les conditions physiologiques, et celle du complexe formé avec l'angiogénine est inférieure à 1 fM.
L'inhibiteur de ribonucléase peut se lier à différents types de ribonucléases pancréatiques malgré la relative variabilité de leur séquence peptidique. Les études biochimiques et cristallographiques suggèrent que les interactions protéine-protéine des complexes formés par l'inhibiteur de ribonucléase sont principalement de nature électrostatique mais font également intervenir de grandes régions profondes[3],[4].
L'affinité de cette protéine avec les ribonucléases joue un rôle physiologique important dans la mesure où de nombreuses ribonucléases ont un effet cytotoxique et cytostatique fortement corrélé avec leur capacité à se lier à l'inhibiteur de ribonucléase[5].
Les inhibiteurs de ribonucléase de mammifères n'ont pas d'affinité pour certaines ribonucléases pancréatiques d'autres espèces. C'est en particulier le cas pour certaines ribonucléases d'amphibiens telles que la ranpirnase/onconase et l'amphinase de la grenouille léopard, ce qui confère à ces ribonucléases une certaine cytotoxicité contre les cellules cancéreuses[6].
Notes et références[modifier | modifier le code]
- (en) Bostjan Kobef et Johann Deisenhofer, « Mechanism of Ribonuclease Inhibition by Ribonuclease Inhibitor Protein Based on the Crystal Structure of its Complex with Ribonuclease A », Journal of Molecular Biology, vol. 264, no 5, , p. 1028-1043 (PMID 9000628, DOI 10.1006/jmbi.1996.0694, lire en ligne)
- (en) Cytoplasmic Ribonuclease Inhibitor, « Robert Shapiro », Methods in Enzymology, vol. 341, , p. 611-628 (PMID 11582809, DOI 10.1016/S0076-6879(01)41180-3, lire en ligne)
- (en) Frank S. Lee, Robert Shapiro, Bert L. Vallee, « Tight-binding inhibition of angiogenin and ribonuclease A by placental ribonuclease inhibitor », Biochemistry, vol. 28, no 1, , p. 225-230 (PMID 2706246, DOI 10.1021/bi00427a031, lire en ligne)
- (en) Anastassios C. Papageorgiou, Robert Shapiro et K.Ravi Acharya, « Molecular recognition of human angiogenin by placental ribonuclease inhibitor—an X‐ray crystallographic study at 2.0 Å resolution », EMBO Journal, vol. 16, no 17, , p. 5162-5177 (PMID 9311977, DOI 10.1093/emboj/16.17.5162, lire en ligne)
- (en) Alexander A. Makarov et Olga N. Ilinskaya, « Cytotoxic ribonucleases: molecular weapons and their targets », FEBS Letters, vol. 540, nos 1-3, , p. 15-20 (PMID 12681476, DOI 10.1016/S0014-5793(03)00225-4, lire en ligne)
- (en) W. Ardelt, K. Shogen et Z. Darzynkiewicz, « Onconase and Amphinase, the Antitumor Ribonucleases from Rana pipiens Oocytes », Current Pharmaceutical Biotechnology, vol. 9, no 3, , p. 215-225 (PMID 18673287, PMCID 2586917, DOI 10.2174/138920108784567245, lire en ligne)
