Jonction de Holliday

Une jonction de Holliday est une jonction mobile entre quatre brins d'ADN. La structure tire son nom de Robin Holliday, qui l'a proposé en 1964[1],[2],[3] pour expliquer un type particulier d'échange d'information génétique qu'il a observé chez Ustilago maydis, la recombinaison homologue. Elles sont en fait un intermédiaire lors du processus de recombinaison génétique, très important au maintien de l'intégrité du génome[4],[1].
La jonction de Holliday est généralement symétrique, ce qui permet un déplacement spécifique selon un motif préservant l'appariement des bases. Ceci signifie que les 4 bras individuels peuvent glisser les uns sur les autres tout en conservant la structure.
Structure[modifier | modifier le code]

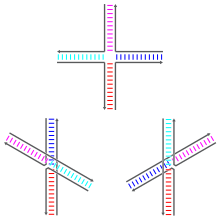
Les jonctions de Holliday peuvent exister sous différentes conformations avec différents patrons de superposition coaxiale entre les 4 bras de la double hélice d'ADN. La superposition coaxiale est la tendance des bouts francs des acides nucléiques de se lier entre eux via des interactions entre les bases exposées. Il existe trois conformations possibles : une forme sans superposition des brins et deux formes avec superposition des brins d'ADN. En l'absence de cations divalents tels que le Mg2+, la forme sans superposition est priorisée dû à la répulsion électrostatique entre les charges négatives des brins d'ADN. En présence d'un minimum de 0,1 mM Mg2+, la répulsion électrostatique est diminuée et les formes avec superposition prédominent.
Les jonctions de Holliday dans la recombinaison homologue sont composées de séquences identiques ou presque identiques, impliquant alors que la jonction centrale possède un arrangement symétrique. Ceci permet la migration des brins. Le clivage, ou la résolution de la jonction de Holliday, peut s’effectuer selon deux méthodes. Dans la première méthode, il y a un clivage double brin du brin receveur ou du brin envahissant. On observe dans le produit final une conversion génique au niveau du brin receveur provenant du brin envahissant sans que ce dernier ne soit modifié ou vis-versa. Dans la deuxième méthode, il y a un clivage double brin au niveau des deux chromatides. On observe un enjambement entre les chromatides, ce qui permet un échange de matériels génétiques entre les régions homologues. Peu importe la méthode effectuée, on obtient des régions hétéroduplexes dans la région où il y a eu la formation de la jonction de Holliday.
Fonction[modifier | modifier le code]
La jonction de Holliday est un intermédiaire dans la recombinaison homologue, un processus biologique qui augmente la diversité génétique en recombinant les gènes entre deux chromosomes. La jonction de Holliday est également présente dans la recombinaison site specifique nécessitant des intégrases en plus des évènements de réparation de bris double brins de l'ADN[5]. Par ailleurs, des jonctions de Holliday peuvent apparaitre dans des structures cruciformes d'ADN non B dans le but de réduire le stress sur les hélices induit par des séquences symétriques d'ADN surenroulé[6].
Résolution[modifier | modifier le code]
Dans une jonction de Holliday, deux duplex homologues sont liés via un échange de nucléotides entre deux brins d’ADN doubles brins. La jonction implique donc la présence de 4 brins d’ADN. La formation de la jonction de Holliday commence par la formation d’une boucle D d’ADN dans laquelle un brin d’un duplex envahit le deuxième duplex. Ceci crée des régions hétéroduplexes d’ADN, c’est-à-dire des régions où l’ADN des deux brins est complémentaire. Le clivage de la boucle D suivi par une ligation crée la jonction de Holliday. Les duplex ainsi liés vont ensuite adopter une forme planaire à la suite d'interactions avec des protéines recombinases. À ce stade, on observe deux régions homoduplexes et deux régions hétéroduplexes créant ainsi deux axes de symétries, un axe vertical et un axe horizontal. Une protéine recombinase est alors en mesure de cliver les brins selon ces axes de symétrie. Dépendamment de l’axe qui est clivé, les produits vont varier. Si le clivage s’effectue selon l’axe horizontal, les deux brins filles vont différer des brins parentaux seulement d’une partie de leur séquence, un échange génétique entre les régions homologues va s’être effectué entre les brins parentaux. On obtient un produit sans recombinaison génétique, c’est-à-dire que les brins parentaux sont encore liés ensemble. Cependant, si le clivage s’effectue selon l’axe vertical, il va y avoir un enjambement entre les chromatides. On obtient un produit avec recombinaison génétique où les brins parentaux ont été échangés.
Voir aussi[modifier | modifier le code]
Références[modifier | modifier le code]
- (en) Stahl FW, « The Holliday junction on its thirtieth anniversary », Genetics, vol. 138, no 2, , p. 241–246 (PMID 7828807, lire en ligne [PDF ])
- (en) Liu Y, West S, « Happy Hollidays: 40th anniversary of the Holliday junction », Nat Rev Mol Cell Biol, vol. 5, no 11, , p. 937–44 (PMID 15520813, DOI 10.1038/nrm1502)
- (en) Hays FA, Watson J, Ho PS, « Caution! DNA Crossing: Crystal Structures of Holliday Junctions », J Biol Chem, vol. 278, no 50, , p. 49663–49666 (DOI 10.1074/jbc.R300033200)
- (en) Fu TJ, Tse-Dinh YC, Seeman NC, « Holliday junction crossover topology », J. Mol. Biol., vol. 236, no 1, , p. 91–105 (PMID 8107128, DOI 10.1006/jmbi.1994.1121)
- (en) David M. J. Lilley, « Structures of helical junctions in nucleic acids », Quarterly Reviews of Biophysics, vol. 33, no 2, , p. 109–159 (ISSN 0033-5835 et 1469-8994, DOI 10.1017/s0033583500003590, lire en ligne, consulté le )
- (en) Bloomfield, Victor A. et Tinoco, Ignacio., Nucleic acids : structures, properties, and functions, Sausalito, Calif., University Science Books, , 794 p. (ISBN 0-935702-49-0 et 9780935702491, OCLC 39936769, lire en ligne)
Liens externes[modifier | modifier le code]
Tous ces liens sont en anglais


