Robustesse (évolution)

En biologie de l'évolution, la robustesse d'un système biologique (également appelée robustesse biologique ou génétique[1], robustness en anglais) correspond à la persistance d'une certaine caractéristique ou trait de ce système subissant des perturbations ou des conditions incertaines[2],[3]. En biologie du développement, la robustesse est connue sous le nom de canalisation[4],[5]. Selon le type de perturbation concerné, la robustesse peut être classifiée comme mutationnelle, environnementale, recombinatoire, comportementale, etc[6],[7],[8]. La robustesse est obtenue par la combinaison de plusieurs mécanismes génétiques et moléculaires et peut évoluer aussi bien par sélection directe que par sélection indirecte. Plusieurs systèmes d'étude d'organismes modèles ont été développés afin d'étudier expérimentalement la robustesse et ses conséquences sur le plan de l'évolution.
Classification[modifier | modifier le code]
Robustesse mutationnelle[modifier | modifier le code]
La robustesse mutationnelle (également appelée tolérance aux mutations) décrit l'étendue jusqu'à laquelle le phénotype d'un organisme reste constant malgré une ou plusieurs mutations affectant son génotype[9]. Cette robustesse peut être quantifiée empiriquement dans plusieurs génomes[10],[11] et gènes individuels[12] en induisant des mutations et en mesurant la proportion de mutants qui conservent le même phénotype, la même fonction ou la même valeur sélective. De façon plus générale, ce mécanisme est connu comme la distribution des effets sur la fitness due aux mutations (autrement dit, les fréquences des différentes valeurs sélectives des mutants). Les protéines étudiées jusqu'alors ont montré une tolérance aux mutations d'environ 66 % (autrement dit, deux tiers des mutations sont neutres)[13].
Inversement, la robustesse mutationnelle mesurée chez les organismes varie largement d'une espèce à l'autre. Par exemple, plus de 95 % des mutations ponctuelles chez C. elegans n'ont aucun effet détectable[14], et jusqu'à 90 % des knockouts de gènes individuels chez E. coli ne sont pas létaux[15]. Cependant, les virus tolèrent seulement 20 à 40 % de mutations et sont de fait plus sensibles à la mutagénèse[10].
Robustesse environnementale[modifier | modifier le code]
Dans des environnements qui changent, l'adaptation parfaite à une condition peut survenir aux dépens de l'adaptation à une autre. En conséquence, la pression de sélection totale d'un organisme donné correspond à la sélection moyenne sur tous les environnements possibles pondérée par le pourcentage de temps passé dans l'environnement considéré. Un environnement variable peut ainsi sélectionner une robustesse environnementale dans laquelle les organismes peuvent prospérer dans une large gamme de conditions avec seulement un léger changement dans le phénotype ou la valeur sélective. Certains organismes montrent des adaptations permettant de tolérer des changements conséquents dans la température, la disponibilité en eau, la salinité ou la disponibilité en nourriture. Les plantes en particulier sont incapables de se déplacer lorsque l'environnement change et possèdent ainsi, pour compenser, divers mécanismes leur permettant d'obtenir une certaine robustesse environnementale. De façon similaire, on peut retrouver cela chez des protéines possédant une tolérance à une large gamme de solvants, de concentrations ioniques ou de températures.
Causes génétiques et moléculaires[modifier | modifier le code]

Bien que les génomes mutent à cause de facteurs environnementaux et d'imperfections de réplication, ils montrent néanmoins une tolérance remarquable à ces mutations. Cela provient de la robustesse à la fois au niveau du génome et du protéome.
Robustesse mutationnelle au niveau de l'organisme[modifier | modifier le code]
Il existe de nombreux mécanismes conférant une robustesse à un génome. Par exemple, la redondance génétique réduit l'effet des mutations dans n'importe quelle copie d'un gène multi-copies[16]. De plus, le flux au sein d'une voie métabolique est typiquement limité seulement par quelques-unes des étapes qui le constituent, ce qui signifie que des changements dans la fonction de la plupart des enzymes n'ont qu'un faible effet sur la valeur sélective[17],[18]. De façon analogue, les réseaux métaboliques possèdent de multiples voies alternatives pour produire la plupart de leurs métabolites essentiels[19].
Robustesse mutationnelle au niveau protéique[modifier | modifier le code]
La tolérance d'une mutation au niveau d'une protéine est le résultat de deux caractéristiques principales : la structure du code génétique et la robustesse structurelle de cette protéine[20],[21]. Les protéines résistent à la plupart des mutations parce que la plupart de leurs séquences peuvent se replier pour obtenir des conformations structurelles fortement similaires[22]. Une protéine adopte un ensemble limité de conformations de base parce que ces formes possèdent une énergie plus faible qu'à l'état déplié ou semi-replié (voir les ΔG de repliement)[23],[24]. Cela est accompli par un réseau interne décentralisé d'interactions coopératives (hydrophobes, polaires et covalentes)[25]. La robustesse structurelle protéique résulte de quelques mutations individuelles qui sont suffisamment perturbatrices pour compromettre la fonction de la protéine. Les protéines ont également évolué afin d'éviter l'agrégation[26], qui correspond au fait que les protéines partiellement repliées peuvent se combiner entre elles pour former des masses et des amyloïdes de grande taille, répétées et insolubles[27]. Il existe des preuves montrant que les protéines présentent des caractéristiques de conception négatives afin de réduire l'exposition aux motifs en feuillets bêta qui favorisent l'agrégation au sein de leurs structures[28]. De plus, des preuves complémentaires montrent que le code génétique en lui-même peut être optimisé de telle façon que la majorité des mutations ponctuelles résultent en des acides aminés similaires (conservatifs)[29],[30]. Ces facteurs pris ensemble créent une distribution des effets sur la fitness due aux mutations, qui contiennent une forte proportion de mutations neutres et quasi-neutres[12].
Conséquences sur le plan de l'évolution[modifier | modifier le code]
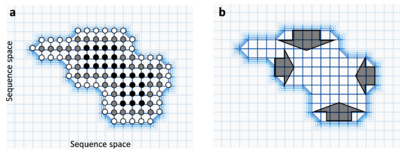
Comme les organismes sont constamment exposés aux perturbations génétiques et non génétiques, la robustesse est importante pour assurer la stabilité des phénotypes. De plus, sous l'équilibre de sélection de mutations, la robustesse mutationnelle peut permettre à une variation génétique cryptique de s'accumuler dans une population. Alors que ces différences génétiques sont phénotypiquement neutres dans un environnement stable, elles peuvent se révéler conférer des différences dans un environnement changeant (notion de capacité évolutive), permettant ainsi l'expression d'un plus grand nombre de phénotypes héritables dans des populations exposées à un environnement variable[31].
Le fait d'être robuste peut également être favorisé aux dépens de la valeur sélective totale dans une stratégie évolutivement stable (également appelée "survie du plus plat")[32]. Un pic fort mais étroit du paysage adaptatif[A 1] confère une valeur sélective forte mais une robustesse moindre car la plupart des mutations conduisent à une perte massive de valeur sélective. De forts taux de mutations peuvent favoriser une population avec des pics de fitness moins hauts mais plus larges. Des systèmes biologiques plus essentiels (organes ou tissus par exemple) peuvent aussi avoir une meilleure sélection de robustesse car les réductions de fonction sont plus dommageables pour la fitness[32]. On suppose que la robustesse mutationnelle est un moteur de la formation théorique des quasi-espèces virales.
Robustesse mutationnelle émergente[modifier | modifier le code]
La sélection naturelle peut directement ou indirectement inactiver la robustesse. Lorsque les taux de mutations sont forts et que les tailles des populations sont importantes, on prédit que ces dernières auront tendance à aller vers les parties plus densément connectées au sein d'un réseau neutre[A 2] car les variants moins robustes auront moins de descendants mutants qui survivront[33]. Les conditions selon lesquelles la sélection peut directement augmenter la robustesse mutationnelle dans ce sens sont restreintes, et donc on pense qu'une telle sélection est limitée à quelques virus[34] et microbes[35] ayant de grandes tailles de populations et de forts taux de mutations. Une robustesse émergente de ce type a été observée dans l'évolution expérimentale du cytochrome P450[36] et de la β-lactamase[37]. Inversement, la robustesse mutationnelle peut évoluer comme un sous-produit de la sélection naturelle de la robustesse face aux perturbations environnementales[38],[39],[40],[41],[42].
Robustesse et évolvabilité[modifier | modifier le code]
On a supposé que la robustesse mutationnelle avait un impact négatif sur l'évolvabilité parce qu'elle réduit l'accessibilité mutationnelle de phénotypes héritables distincts liés à un même génotype, et qu'elle réduit les différences sélectives au sein d'une population génétiquement diversifiée. De façon contre-intuitive cependant, les chercheurs ont supposé que la robustesse phénotypique face aux mutations pourrait en réalité augmenter le rythme d'adaptation phénotypique héritable lorsque l'on se place sur de longues échelles de temps[43],[44],[45],[46].
L'une des hypothèses expliquant la façon dont la robustesse favorise l'évolutivité dans les populations asexuées propose que les réseaux connectés de génotypes neutres pour la fitness résultent en une robustesse mutationnelle qui réduit l'accessibilité à de nouveaux phénotypes héritables sur de courtes échelles de temps. Sur de longues échelles de temps, les mutations neutres et la dérive génétique poussent la population à se disperser sur un plus grand réseau neutre dans l'espace génotypique. Cette diversité génétique donne à la population un accès à un plus grand nombre de phénotypes héritables distincts qui peuvent être obtenus à partir de différents points du réseau neutre[43],[44],[46],[47],[48],[49],[50]. Cependant, ce mécanisme peut être limité aux phénotypes dépendants d'un seul locus ; pour les caractères polygéniques, la diversité génétique dans les populations asexuées n'augmente pas l'évolvabilité de façon significative[51].
Dans le cas des protéines, la robustesse favorise l'évolvabilité sous la forme d'un excès d'énergie libre de repliement des protéines[52]. Comme la plupart des mutations réduisent la stabilité, un excès de cette énergie libre permet de tolérer des mutations qui sont bénéfiques pour l'activité de la protéine mais déstabiliseraient autrement la protéine.
Dans les populations sexuées, la robustesse conduit à l'accumulation de variations génétiques cryptiques à fort potentiel évolutif[53],[54].
L'évolvabilité peut être forte lorsque la robustesse est réversible, avec une capacité évolutive (stockage et relargage de variations) permettant de passer d'une robustesse forte dans la majorité des cas à une robustesse plus faible dans des situations de stress[55].
Méthodes et systèmes modèles[modifier | modifier le code]
Plusieurs systèmes ont été utilisés pour étudier la robustesse. Des modèles in silico ont été utilisés pour modéliser la structure secondaire de l'ARN, les treillis protéiques[A 3], ou encore les réseaux géniques de Wagner[A 4]. Dans les systèmes expérimentaux pour gènes individuels, les chercheurs ont utilisé l'activité enzymatique du cytochrome P450[36], la β-lactamase[37], l'ARN polymérase[13] et les répresseurs de l'opéron lac[13]. La robustesse à l'échelle de l'organisme entier a été étudiée au niveau de la fitness des virus à ARN[10], de la chimiotaxie bactérienne, de la fitness chez la drosophile[15], des réseaux de polarité des segments, des réseaux neurogéniques et du gradient protéique des BMPs, de la fitness chez C. elegans[14] et du développement vulval, et de l'horloge circadienne chez les mammifères[9].
Voir aussi[modifier | modifier le code]
Notes[modifier | modifier le code]
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Robustness (evolution) » (voir la liste des auteurs).
- Schéma permettant de visualiser la relation entre des génotypes et le succès reproductif.
- Groupe de gènes liés par des mutations ponctuelles ayant une fonction ou fitness équivalente.
- Modèles permettant de modéliser le repliement protéique.
- Modèles permettant de modéliser le processus évolutif et développemental des réseaux génétiques régulateurs.
Articles connexes[modifier | modifier le code]
Références[modifier | modifier le code]
- Hiroaki Kitano, « Biological robustness », Nature Reviews. Genetics, vol. 5, no 11, , p. 826–837 (ISSN 1471-0056, PMID 15520792, DOI 10.1038/nrg1471, lire en ligne, consulté le )
- Jörg Stelling, Uwe Sauer, Zoltan Szallasi et Francis J. Doyle, « Robustness of cellular functions », Cell, vol. 118, no 6, , p. 675–685 (ISSN 0092-8674, PMID 15369668, DOI 10.1016/j.cell.2004.09.008, lire en ligne, consulté le )
- M.-A. Félix et A. Wagner, « Robustness and evolution: concepts, insights and challenges from a developmental model system », Heredity, vol. 100, no 2, , p. 132–140 (ISSN 1365-2540, PMID 17167519, DOI 10.1038/sj.hdy.6800915, lire en ligne, consulté le )
- C. H. WADDINGTON, « Canalization of Development and the Inheritance of Acquired Characters », Nature, vol. 150, no 3811, , p. 563–565 (DOI 10.1038/150563a0, lire en ligne)
- J. Arjan G. M. de Visser, Joachim Hermisson, Günter P. Wagner et Lauren Ancel Meyers, « Perspective: Evolution and detection of genetic robustness », Evolution; International Journal of Organic Evolution, vol. 57, no 9, , p. 1959–1972 (ISSN 0014-3820, PMID 14575319, lire en ligne, consulté le )
- Jose A. Fernandez-Leon, « Evolving cognitive-behavioural dependencies in situated agents for behavioural robustness », Bio Systems, vol. 106, nos 2-3, , p. 94–110 (ISSN 1872-8324, PMID 21840371, DOI 10.1016/j.biosystems.2011.07.003, lire en ligne, consulté le )
- Jose A. Fernandez-Leon, « Behavioural robustness: a link between distributed mechanisms and coupled transient dynamics », Bio Systems, vol. 105, no 1, , p. 49–61 (ISSN 1872-8324, PMID 21466836, DOI 10.1016/j.biosystems.2011.03.006, lire en ligne, consulté le )
- Jose A. Fernandez-Leon, « Evolving experience-dependent robust behaviour in embodied agents », Bio Systems, vol. 103, no 1, , p. 45–56 (ISSN 1872-8324, PMID 20932875, DOI 10.1016/j.biosystems.2010.09.010, lire en ligne, consulté le )
- (en) Andreas Wagner, Robustness and Evolvability in Living Systems, Princeton University Press, , 368 p. (ISBN 978-1-4008-4938-3, lire en ligne)
- Rafael Sanjuán, « Mutational fitness effects in RNA and single-stranded DNA viruses: common patterns revealed by site-directed mutagenesis studies », Philosophical Transactions of the Royal Society B: Biological Sciences, vol. 365, no 1548, , p. 1975–1982 (ISSN 0962-8436, PMID 20478892, PMCID PMC2880115, DOI 10.1098/rstb.2010.0063, lire en ligne, consulté le )
- Adam Eyre-Walker et Peter D. Keightley, « The distribution of fitness effects of new mutations », Nature Reviews. Genetics, vol. 8, no 8, , p. 610–618 (ISSN 1471-0056, PMID 17637733, DOI 10.1038/nrg2146, lire en ligne, consulté le )
- Ryan T. Hietpas, Jeffrey D. Jensen et Daniel N. A. Bolon, « Experimental illumination of a fitness landscape », Proceedings of the National Academy of Sciences of the United States of America, vol. 108, no 19, , p. 7896–7901 (ISSN 1091-6490, PMID 21464309, PMCID PMC3093508, DOI 10.1073/pnas.1016024108, lire en ligne, consulté le )
- Haiwei H. Guo, Juno Choe et Lawrence A. Loeb, « Protein tolerance to random amino acid change », Proceedings of the National Academy of Sciences of the United States of America, vol. 101, no 25, , p. 9205–9210 (ISSN 0027-8424, PMID 15197260, DOI 10.1073/pnas.0403255101, lire en ligne, consulté le )
- E. K. Davies, A. D. Peters et P. D. Keightley, « High frequency of cryptic deleterious mutations in Caenorhabditis elegans », Science (New York, N.Y.), vol. 285, no 5434, , p. 1748–1751 (ISSN 0036-8075, PMID 10481013, lire en ligne, consulté le )
- Tomoya Baba, Takeshi Ara, Miki Hasegawa et Yuki Takai, « Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection », Molecular Systems Biology, vol. 2, , p. 2006.0008 (ISSN 1744-4292, PMID 16738554, PMCID PMC1681482, DOI 10.1038/msb4100050, lire en ligne, consulté le )
- Zhenglong Gu, Lars M. Steinmetz, Xun Gu et Curt Scharfe, « Role of duplicate genes in genetic robustness against null mutations », Nature, vol. 421, no 6918, , p. 63–66 (ISSN 0028-0836, PMID 12511954, DOI 10.1038/nature01198, lire en ligne, consulté le )
- Kenneth J. Kauffman, Purusharth Prakash et Jeremy S. Edwards, « Advances in flux balance analysis », Current Opinion in Biotechnology, vol. 14, no 5, , p. 491–496 (ISSN 0958-1669, PMID 14580578, lire en ligne, consulté le )
- Hojung Nam, Nathan E. Lewis, Joshua A. Lerman et Dae-Hee Lee, « Network context and selection in the evolution to enzyme specificity », Science (New York, N.Y.), vol. 337, no 6098, , p. 1101–1104 (ISSN 0036-8075, PMID 22936779, PMCID PMC3536066, DOI 10.1126/science.1216861, lire en ligne, consulté le )
- David C. Krakauer et Joshua B. Plotkin, « Redundancy, antiredundancy, and the robustness of genomes », Proceedings of the National Academy of Sciences of the United States of America, vol. 99, no 3, , p. 1405–1409 (ISSN 0027-8424, PMID 11818563, DOI 10.1073/pnas.032668599, lire en ligne, consulté le )
- Darin M. Taverna et Richard A. Goldstein, « Why are proteins so robust to site mutations? », Journal of Molecular Biology, vol. 315, no 3, , p. 479–484 (ISSN 0022-2836, PMID 11786027, DOI 10.1006/jmbi.2001.5226, lire en ligne, consulté le )
- Nobuhiko Tokuriki et Dan S. Tawfik, « Stability effects of mutations and protein evolvability », Current Opinion in Structural Biology, vol. 19, no 5, , p. 596–604 (ISSN 1879-033X, PMID 19765975, DOI 10.1016/j.sbi.2009.08.003, lire en ligne, consulté le )
- Leonid Meyerguz, Jon Kleinberg et Ron Elber, « The network of sequence flow between protein structures », Proceedings of the National Academy of Sciences of the United States of America, vol. 104, no 28, , p. 11627–11632 (ISSN 0027-8424, PMID 17596339, PMCID PMC1913895, DOI 10.1073/pnas.0701393104, lire en ligne, consulté le )
- Martin Karplus, « Behind the folding funnel diagram », Nature Chemical Biology, vol. 7, no 7, , p. 401–404 (ISSN 1552-4469, PMID 21685880, DOI 10.1038/nchembio.565, lire en ligne, consulté le )
- Nobuhiko Tokuriki, Francois Stricher, Joost Schymkowitz et Luis Serrano, « The stability effects of protein mutations appear to be universally distributed », Journal of Molecular Biology, vol. 369, no 5, , p. 1318–1332 (ISSN 0022-2836, PMID 17482644, DOI 10.1016/j.jmb.2007.03.069, lire en ligne, consulté le )
- Boris E. Shakhnovich, Eric Deeds, Charles Delisi et Eugene Shakhnovich, « Protein structure and evolutionary history determine sequence space topology », Genome Research, vol. 15, no 3, , p. 385–392 (ISSN 1088-9051, PMID 15741509, DOI 10.1101/gr.3133605, lire en ligne, consulté le )
- Elodie Monsellier et Fabrizio Chiti, « Prevention of amyloid-like aggregation as a driving force of protein evolution », EMBO reports, vol. 8, no 8, , p. 737–742 (ISSN 1469-221X, PMID 17668004, PMCID PMC1978086, DOI 10.1038/sj.embor.7401034, lire en ligne, consulté le )
- A. L. Fink, « Protein aggregation: folding aggregates, inclusion bodies and amyloid », Folding & Design, vol. 3, no 1, , R9–23 (ISSN 1359-0278, PMID 9502314, DOI 10.1016/S1359-0278(98)00002-9, lire en ligne, consulté le )
- Jane Shelby Richardson et David C. Richardson, « Natural beta-sheet proteins use negative design to avoid edge-to-edge aggregation », Proceedings of the National Academy of Sciences of the United States of America, vol. 99, no 5, , p. 2754–2759 (ISSN 0027-8424, PMID 11880627, DOI 10.1073/pnas.052706099, lire en ligne, consulté le )
- Manuel M. Müller, Jane R. Allison, Narupat Hongdilokkul et Laurent Gaillon, « Directed Evolution of a Model Primordial Enzyme Provides Insights into the Development of the Genetic Code », PLoS Genetics, vol. 9, no 1, (ISSN 1553-7390, PMID 23300488, PMCID PMC3536711, DOI 10.1371/journal.pgen.1003187, lire en ligne, consulté le )
- Elad Firnberg et Marc Ostermeier, « The genetic code constrains yet facilitates Darwinian evolution », Nucleic Acids Research, vol. 41, no 15, , p. 7420–7428 (ISSN 0305-1048, PMID 23754851, PMCID PMC3753648, DOI 10.1093/nar/gkt536, lire en ligne, consulté le )
- Joanna Masel et Mark L. Siegal, « Robustness: mechanisms and consequences », Trends in genetics: TIG, vol. 25, no 9, , p. 395–403 (ISSN 0168-9525, PMID 19717203, PMCID PMC2770586, DOI 10.1016/j.tig.2009.07.005, lire en ligne, consulté le )
- C. O. Wilke, J. L. Wang, C. Ofria et R. E. Lenski, « Evolution of digital organisms at high mutation rates leads to survival of the flattest », Nature, vol. 412, no 6844, , p. 331–333 (ISSN 0028-0836, PMID 11460163, DOI 10.1038/35085569, lire en ligne, consulté le )
- Erik van Nimwegen, James P. Crutchfield et Martijn Huynen, « Neutral evolution of mutational robustness », Proceedings of the National Academy of Sciences of the United States of America, vol. 96, no 17, , p. 9716–9720 (ISSN 0027-8424, PMID 10449760, lire en ligne, consulté le )
- Rebecca Montville, Remy Froissart, Susanna K. Remold et Olivier Tenaillon, « Evolution of Mutational Robustness in an RNA Virus », PLOS Biology, vol. 3, no 11, , e381 (ISSN 1545-7885, DOI 10.1371/journal.pbio.0030381, lire en ligne, consulté le )
- Joanna Masel et Heather Maughan, « Mutations Leading to Loss of Sporulation Ability in Bacillus subtilis Are Sufficiently Frequent to Favor Genetic Canalization », Genetics, vol. 175, no 1, , p. 453–457 (ISSN 0016-6731, PMID 17110488, PMCID PMC1775008, DOI 10.1534/genetics.106.065201, lire en ligne, consulté le )
- Jesse D Bloom, Zhongyi Lu, David Chen et Alpan Raval, « Evolution favors protein mutational robustness in sufficiently large populations », BMC Biology, vol. 5, , p. 29 (ISSN 1741-7007, PMID 17640347, PMCID PMC1995189, DOI 10.1186/1741-7007-5-29, lire en ligne, consulté le )
- Shimon Bershtein, Korina Goldin et Dan S. Tawfik, « Intense neutral drifts yield robust and evolvable consensus proteins », Journal of Molecular Biology, vol. 379, no 5, , p. 1029–1044 (ISSN 1089-8638, PMID 18495157, DOI 10.1016/j.jmb.2008.04.024, lire en ligne, consulté le )
- Colin D. Meiklejohn et Daniel L. Hartl, « A single mode of canalization », Trends in Ecology & Evolution, vol. 17, no 10, , p. 468–473 (DOI 10.1016/S0169-5347(02)02596-X, lire en ligne, consulté le )
- L. W. Ancel et W. Fontana, « Plasticity, evolvability, and modularity in RNA », The Journal of Experimental Zoology, vol. 288, no 3, , p. 242–283 (ISSN 0022-104X, PMID 11069142, lire en ligne, consulté le )
- Gergely J. Szöllosi et Imre Derényi, « Congruent evolution of genetic and environmental robustness in micro-RNA », Molecular Biology and Evolution, vol. 26, no 4, , p. 867–874 (ISSN 1537-1719, PMID 19168567, DOI 10.1093/molbev/msp008, lire en ligne, consulté le )
- Günter P. Wagner, Ginger Booth et Homayoun Bagheri-Chaichian, « A Population Genetic Theory of Canalization », Evolution, vol. 51, no 2, , p. 329–347 (DOI 10.2307/2411105, lire en ligne, consulté le )
- Ben Lehner, « Genes confer similar robustness to environmental, stochastic, and genetic perturbations in yeast », PloS One, vol. 5, no 2, , e9035 (ISSN 1932-6203, PMID 20140261, PMCID PMC2815791, DOI 10.1371/journal.pone.0009035, lire en ligne, consulté le )
- Jeremy A. Draghi, Todd L. Parsons, Günter P. Wagner et Joshua B. Plotkin, « Mutational robustness can facilitate adaptation », Nature, vol. 463, no 7279, , p. 353–355 (ISSN 0028-0836, PMID 20090752, PMCID PMC3071712, DOI 10.1038/nature08694, lire en ligne, consulté le )
- Andreas Wagner, « Robustness and evolvability: a paradox resolved », Proceedings of the Royal Society B: Biological Sciences, vol. 275, no 1630, , p. 91–100 (ISSN 0962-8452, PMID 17971325, PMCID PMC2562401, DOI 10.1098/rspb.2007.1137, lire en ligne, consulté le )
- Joanna Masel et Meredith V. Trotter, « Robustness and evolvability », Trends in genetics: TIG, vol. 26, no 9, , p. 406–414 (ISSN 0168-9525, PMID 20598394, PMCID PMC3198833, DOI 10.1016/j.tig.2010.06.002, lire en ligne, consulté le )
- Maximino Aldana, Enrique Balleza, Stuart Kauffman et Osbaldo Resendiz, « Robustness and evolvability in genetic regulatory networks », Journal of Theoretical Biology, vol. 245, no 3, , p. 433–448 (ISSN 0022-5193, PMID 17188715, DOI 10.1016/j.jtbi.2006.10.027, lire en ligne, consulté le )
- A. Babajide, I. L. Hofacker, M. J. Sippl et P. F. Stadler, « Neutral networks in protein space: a computational study based on knowledge-based potentials of mean force », Folding & Design, vol. 2, no 5, , p. 261–269 (ISSN 1359-0278, PMID 9261065, DOI 10.1016/S1359-0278(97)00037-0, lire en ligne, consulté le )
- E. van Nimwegen et J. P. Crutchfield, « Metastable evolutionary dynamics: crossing fitness barriers or escaping via neutral paths? », Bulletin of Mathematical Biology, vol. 62, no 5, , p. 799–848 (ISSN 0092-8240, PMID 11016086, DOI 10.1006/bulm.2000.0180, lire en ligne, consulté le )
- (en) S. Ciliberti, O. C. Martin et A. Wagner, « Innovation and robustness in complex regulatory gene networks », Proceedings of the National Academy of Sciences, vol. 104, no 34, , p. 13591–13596 (ISSN 0027-8424 et 1091-6490, PMID 17690244, DOI 10.1073/pnas.0705396104, lire en ligne, consulté le )
- Andreas Wagner, « Neutralism and selectionism: a network-based reconciliation », Nature Reviews. Genetics, vol. 9, no 12, , p. 965–974 (ISSN 1471-0064, PMID 18957969, DOI 10.1038/nrg2473, lire en ligne, consulté le )
- Etienne Rajon et Joanna Masel, « Compensatory evolution and the origins of innovations », Genetics, vol. 193, no 4, , p. 1209–1220 (ISSN 1943-2631, PMID 23335336, PMCID PMC3606098, DOI 10.1534/genetics.112.148627, lire en ligne, consulté le )
- Jesse D. Bloom, Sy T. Labthavikul, Christopher R. Otey et Frances H. Arnold, « Protein stability promotes evolvability », Proceedings of the National Academy of Sciences of the United States of America, vol. 103, no 15, , p. 5869–5874 (ISSN 0027-8424, PMID 16581913, PMCID PMC1458665, DOI 10.1073/pnas.0510098103, lire en ligne, consulté le )
- (en) C. H. Waddington, The strategy of the genes. A discussion of some aspects of theoretical biology. With an appendix by H. Kacser., Londres, George Allen & Unwin, Ltd., , 262 p. (lire en ligne)
- Joanna Masel, « Cryptic Genetic Variation Is Enriched for Potential Adaptations », Genetics, vol. 172, no 3, , p. 1985–1991 (ISSN 0016-6731, PMID 16387877, PMCID PMC1456269, DOI 10.1534/genetics.105.051649, lire en ligne, consulté le )
- Joanna Masel, « Q&A: Evolutionary capacitance », BMC biology, vol. 11, , p. 103 (ISSN 1741-7007, PMID 24228631, PMCID PMC3849687, DOI 10.1186/1741-7007-11-103, lire en ligne, consulté le )
