Protéine fer-soufre
Une protéine fer-soufre, abrégée en protéine Fe-S, est une protéine non héminique possédant dans sa structure un cluster fer-soufre consistant en des groupes de deux, trois ou quatre atomes de fer — chacun dans un état d'oxydation propre — liés à des anions sulfure S2−. De tels clusters se trouvent dans diverses métalloprotéines telles que les ferrédoxines, la NADH déshydrogénase, les hydrogénases, la coenzyme Q-cytochrome c réductase, la succinate déshydrogénase et la nitrogénase[1].
Les clusters fer-soufre sont bien connus pour leur participation dans les réactions d'oxydo-réduction dans la chaîne respiratoire mitochondriale, en particulier dans la NADH déshydrogénase (complexe I) et la succinate déshydrogénase (complexe II). De tels clusters interviennent également dans d'autres processus : comme catalyseurs dans le cas de l'aconitase, comme générateurs de radicaux dans le cas des enzymes à SAM, comme donneurs de soufre dans les biosynthèses de l'acide lipoïque et de la biotine, et même dans certains cas comme régulateurs de l'expression génétique[2].
Les protéines fer-soufre sont sensibles au monoxyde de carbone biogénique. Leur prévalence dans les réactions du métabolisme cellulaire a conduit certains scientifiques à supposer un rôle déterminant pour les clusters fer-soufre dans l'origine de la vie, dans le cadre de scénarios d'abiogenèse appelés « hypothèse du monde fer-soufre » inspirés des observations réalisées autour des cheminées hydrothermales.
Motifs structuraux[modifier | modifier le code]
Dans presque toutes les protéines Fe-S, les centres Fe sont tétraédriques et les ligands terminaux sont des groupes thiols de résidus cystéine. Les groupes sulfure sont bi- ou tri-coordonnés. Les atomes de Soufre servent à stabiliser le Fer par des liaisons de coordination. Il y a autant de soufre inorganique qu'il y a de Fer. La plupart des clusters Fe-S ayant ces caractéristiques appartiennent aux quatre types ci-dessous.
Clusters 1Fe-0S[modifier | modifier le code]
Le centre Fe-S le plus simple est constitué d'un seul atome de fer [1Fe-0S] coordinné par quatre atomes de soufre organique de la chaîne latérale de cystéines de la protéine comme la rubrédoxine par exemple.
Clusters 2Fe-2S[modifier | modifier le code]

Le cluster [2Fe-2S], le plus simple des systèmes polymétalliques, est constitué de deux atomes de fer reliés par deux atomes de soufre inorganique et coordinnés à quatre résidus cystéine dans les ferrédoxines et mitoNEET ou à deux résidus cystéine et deux résidus histidine dans les protéines de Rieske. Les atomes de fer sont deux ions ferriques Fe3+ dans le cas d'une protéine oxydée, ou un ion ferreux Fe2+ et un ion ferrique Fe3+ dans le cas d'une protéine réduite.
Clusters 3Fe-4S[modifier | modifier le code]

Le cluster [3Fe-4S] est constitué de trois atomes de fer reliés deux par deux par trois atomes de soufre, le quatrième atome de soufre reliant les trois atomes de fer. Les états d'oxydation varient de [3Fe-4S]+ (tous les cations à l'état Fe3+) à [3Fe-4S]2− (tous les cations à l'état Fe2+).
Certaines protéines Fe-S peuvent acquérir un ion ferreux pour convertir leur cluster [3Fe-4S] en cluster [4Fe-4S] sous l'action d'un agent réducteur : c'est notamment le cas de l'aconitase, inactive lorsque son cluster n'a que trois cations de fer, et activée lorsqu'il acquiert le quatrième.
Clusters 4Fe-4S[modifier | modifier le code]

Le cluster [Fe4S4] à géométrie de type cubane est la structure fer-soufre la plus courante. Les cations de fer sont typiquement coordonnés par des résidus cystéine. Les ferrédoxines [Fe4S4], des protéines intervenant dans les transferts d'électrons, se rangent en deux catégories dites à bas potentiel (de type bactérien) et à haut potentiel (HiPIP) selon l'état d'oxydation du cluster fer-soufre ; le passage d'une ferrédoxine bas potentiel à une ferrédoxine haut potentiel est illustré par le diagramme suivant :
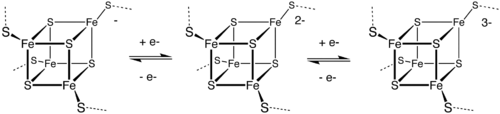
Le cluster des ferrédoxines bactériennes oscille entre les états :
- [ Fe2+3 Fe3+ S2−4 ] e− + [ Fe2+2 Fe3+2 S2−4 ], avec un potentiel d'oxydo-réduction de −0,7 à −0,3 V.
Celui des ferrédoxines HiPIP oscille entre les états :
Ces deux types de ferrédoxines partagent l'état d'oxydation [ Fe2+2 Fe3+2 S2−4 ]. La différence dans les potentiels redox est attribuée à la différence dans les liaisons hydrogène, qui modifient fortement le caractère basique du groupe thiol des résidus cystéine.
Un autre couple redox est mis en œuvre par la nitrogénase, restant plus réducteur que la ferrédoxine bactérienne.
Certaines protéines Fe-S à cluster [Fe4S4] se lient à des substrats et sont donc des enzymes. C'est le cas de l'aconitase, qui se lie au cis-aconitate sur l'un de ces cations de fer dépourvus de ligand à thiol. Le cluster ne déclenche pas de réaction d'oxydo-réduction mais agit comme un acide de Lewis pour catalyser la conversion du cis-aconitate en isocitrate. Dans les enzymes à SAM, le cluster réduit la SAM pour produire un radical permettant de réaliser de nombreuses biosynthèses[3].
Autres clusters Fe-S[modifier | modifier le code]
Les systèmes polymétalliques plus complexes ne sont pas rares. Ainsi, la nitrogénase possède des clusters à sept et à huit cations de fer.
Fonctions et localisations de protéines à centre Fe-S.[modifier | modifier le code]
Les centres Fe-S peuvent avoir des fonctions très diverses. Ils sont très souvent impliqués dans des transferts d’électrons en passant d'une structure de fer ferrique à ferreux (ferrédoxine, complexes de la chaîne respiratoire mitochondriale…) mais ils peuvent être aussi impliqués directement dans une fonction enzymatique (aconitase…), la régulation de l’expression de gène (IRP1, SoxR…) tandis que dans certains cas, on leur attribue une simple fonction structurale (endonucléase III, MutY…)[4].
La plupart des protéines à centre Fe-S sont localisées dans la mitochondrie, le cytosol et le noyau. Une exception est la protéine miner-1 (nommée aussi ERIS) localisée au niveau du réticulum endoplasmique (figure ci-dessous)[5].
Exemples de fonctions et de localisations de protéines à centre Fe-S. Dans la mitochondrie : aconitase (cycle de Krebs) ; complexes de la chaîne respiratoire et biotine synthase (biosynthèse de cofacteur). Dans le noyau : primase (synthèse de l’ADN) ; MYH (réparation de l’ADN) et ADN polymérase. Dans le réticulum endoplasmique : miner-1 (fonction inconnue). Dans le cytosol : IRP1 (homéostasie du fer).
Biogénèse des protéines à centre Fe-S.[modifier | modifier le code]
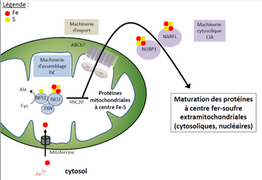
La biogenèse des centres Fe-S commence dans la mitochondrie. Les ions ferreux pénètrent dans la mitochondrie par un transporteur, la mitoferrine. L’assemblage du centre Fe-S est effectué par la machinerie ISC (Iron-Sulfur Cluster). Le soufre inorganique est tout d’abord produit par une cystéine désulfurase, NFS1, à partir de la cystéine. Puis le centre Fe-S est formé sur la protéine ISCU (protéine plateforme) avec l’aide de la frataxine (FXN). Avec l’aide de la protéine chaperone HSC20, le centre Fe-S est soit transféré à des protéines Fe-S mitochondriales soit exporté hors de la mitochondrie grâce à une machinerie d’export constituée notamment d’ABCb7 qui est une protéine ABC (ou MDR, Multi-Drug Resistant). Une fois dans le cytosol, les centres Fe-S sont pris en charge par une machinerie cytosolique d’assemblage des centres Fe-S (CIA) composée notamment de deux protéines à centre Fe-S. NUBP1 et NARFL, qui vont permettre la maturation des protéines à centre Fe-S extramitochondriales (cytosoliques, nucléaires…) (figure ci-dessous)[5],[6],[7].
Biogenèse des centres Fe-S. Le fer ferreux entre par la mitoferrine puis est pris en charge par le complexe ISC qui l’accouple à un atome de soufre inorganique pour former des centres Fe-S. HSC20 aide ces nouveaux centres Fe-S à s’intégrer dans des protéines mitochondriales à centre Fe-S. Cependant, ce dernier peut aussi sortir de la mitochondrie par ABCb7 pour être pris en charge par la CIA permettant la maturation des protéines à centre Fe-S.
Notes et références[modifier | modifier le code]
- (en) S. J. Lippard et J. M. Berg, Principles of Bioinorganic Chemistry, Mill Valley, Californie, États-Unis, University Science Books, , 411 p. (ISBN 0-935702-73-3)
- (en) D. Johnson, D. R. Dean, A. D. Smith et M. K. Johnson, « Structure, function and formation of biological iron–sulfur clusters », Annual Review of Biochemistry, vol. 74, , p. 247–281 (PMID 15952888, DOI 10.1146/annurev.biochem.74.082803.133518)
- (en) S. C. Wang et P. A. Frey, « S-adenosylmethionine as an oxidant: the radical SAM superfamily », Trends Biochem. Sci., vol. 32, no 3, , p. 101-110 (ISSN 0968-0004, PMID 17291766, DOI 10.1016/j.tibs.2007.01.002)
- (en) Romano CA, « Mutants of the base excision repair glycosylase, endonuclease III: DNA charge transport as a first step in lesion detection », Biochemistry, no 50, , p. 6133-6145
- (en) Lill R, « Maturation of iron-sulfur proteins in eukaryotes : mechanisms, connected processes, and diseases », Annual Review of Biochemistry, no 77, , p. 669-700
- (en) Lill R, « The role of mitochondria in cellular iron-sulfur protein biogenesis and iron metabolism. », Biochimica et Biophysica Acta, no 1823, , p. 1491-1508
- (en) Stehling O, « The role of mitochondria in cellular iron-sulfur protein biogenesis : mechanisms, connected processes, and diseases. », Cold Spring Harb Perspect Biol, no 5, , p. 1-17