Enzyme de débranchement du glycogène
| N° EC | EC |
|---|---|
| N° CAS |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
| N° EC | EC |
|---|---|
| N° CAS |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
L'enzyme de débranchement du glycogène (GDE) est un système enzymatique regroupant une activité glycosyltransférase et une activité glycoside hydrolase qui facilite la dégradation du glycogène stocké dans le foie et les muscles en libérant du glucose avec l'aide de phosphorylases. Il s'agit d'une source d'énergie essentielle chez de nombreux êtres vivants. Cette voie métabolique est régulée dans l'organisme, particulièrement dans le foie, sous l'action de diverses hormones telles que l'insuline et le glucagon[1]. Cette régulation très étroite assure l'homéostasie de la glycémie. Les désordres métaboliques résultant de mutations affectant l'enzyme de débranchement du glycogène peuvent conduire par exemple à une glycogénose type 3[2],[3].
Les deux activités enzymatiques constituant l'enzyme de débranchement du glycogène sont réalisées par une protéine unique chez les mammifères, les levures et certaines bactéries, mais par deux protéines distinctes chez E. coli et d'autres bactéries, ce qui complique la nomenclature. Les protéines qui possèdent les deux activités enzymatiques sont appelées enzymes de débranchement du glycogène (GDE), tandis que cette appellation est généralement réservée à la glycoside transférase dans les systèmes à deux enzymes distinctes. Chez certains auteurs, une enzyme qui ne possède qu'une activité glycoside transférase est appelée enzyme de débranchement[4].
Rôle métabolique[modifier | modifier le code]
La glycogène phosphorylase (EC ) est la principale enzyme impliquée dans la dégradation du glycogène, dont elle détache du glucose-1-phosphate lié par une liaison osidique (1→4) :
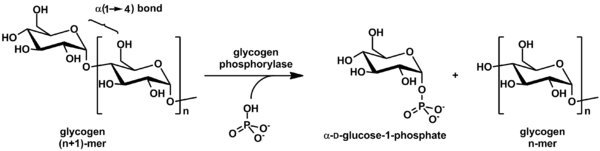
Cette enzyme est en revanche bloquée à hauteur de quatre résidus de glucose en amont des liaisons osidiques (1→6), qui sont celle des ramifications du glycogène ; il faut alors l'intervention d'une enzyme de débranchement du glycogène pour permettre à la glycogène phosphorylase de poursuivre le clivage des liaisons osidiques (1→4) sans être arrêtée par la présence des liaisons osidiques (1→6)[5] :
- l'activité 4-α-glucanotransférase (EC ) déplace trois résidus de glucose de la branche du glycogène vers une autre branche de la molécule, ce qui laisse un unique résidu de glucose lié au glycogène par une liaison osidique (1→6) ;
- l'activité amylo-α-1,6-glucosidase (EC ) clive la liaison osidique (1→6) pour laisser un brin de glycogène constitué de résidus de glucose liés uniquement par des liaisons osidiques (1→4).
Structure[modifier | modifier le code]
Selon les espèces, l'enzyme de débranchement du glycogène est constituée de deux enzymes distinctes ou d'une seule protéine possédant deux sites actifs distincts.
Deux enzymes distinctes[modifier | modifier le code]
Chez E. coli et d'autres bactéries, les activités glycosyltransférase et glycoside hydrolase sont portées par deux enzymes distinctes. Le transfert du glucose est assuré chez E. coli par la 4-α-glucanotransférase, une protéine de 78,5 kDa codée par le gène malQ. Une seconde protéine de 73,6 kDa codée par le gène glgX, appelée enzyme de débranchement, réalise le clivage des liaisons osidiques (1→6).
L'activité de ces deux enzymes n'est pas nécessairement couplée : l'enzyme de débranchement d'E. coli catalyse le clivage de branches de glycogène en fragments de quatre résidus de glucose sans activité glycosyltransférase, fragments qui sont ensuite dégradés par la maltodextrine phosphorylase[2],[6]. La structure de cette dernière enzyme est semblable à celle de l'isoamylase, protéine monomérique possédant un domaine central contenant huit brins β et entouré par huit feuillets α avec une fosse longue de 26 Å et large de 9 Å riche en résidus d'acides aminés aromatiques dont on pense qu'ils stabilisent le brin de glycogène à quatre résidus de glucose avant clivage[2].
Protéine unique avec deux sites actifs[modifier | modifier le code]
Chez les levures et les mammifères, une protéine unique possède les deux activités enzymatiques de débranchement[7]. L'enzyme de débranchement du glycogène humaine, codée par le gène AGL, est un monomère de masse moléculaire 175 kDa. On a pu montrer que les deux fonctions catalytiques de cette protéine peuvent fonctionner indépendamment l'une de l'autre, ce qui démontre la présence de plusieurs sites actifs distincts. Ce résultat a été confirmé par l'existence d'inhibiteurs spécifiques à l'une des deux activités enzymatiques, tels que la polyhydroxylamine qui inhibe sélectivement l'activité glycoside hydrolase sans affecter l'activité glycosyltransférase de manière mesurable[8]. L'enzyme de débranchement du glycogène est la seule enzyme d'eucaryote connue possédant plusieurs sites actifs distincts et active comme monomère[9].
Certaines études ont montré que le côté C-terminal de l'enzyme de levure est associée à l'activité glycoside hydrolase tandis que le côté N-terminal est associée à l'activité glycosyltransférase[7]. Outre ces deux sites actifs, l'enzyme paraît également posséder un troisième site lui permettant de se lier au glycogène[8].
Notes et références[modifier | modifier le code]
- (en) H. G. Hers, W. Verhue et F. van Hoof, « The Determination of Amylo-1,6-Glucosidase », European Journal of Biochemistry, vol. 2, no 3, , p. 257-264 (PMID 6078537, DOI 10.1111/j.1432-1033.1967.tb00133.x, lire en ligne)
- (en) Hyung-Nam Song, Tae-Yang Jung, Jong-Tae Park, Byung-Chul Park, Pyung Keun Myung, Winfried Boos, Eui-Jeon Woo et Kwan-Hwa Park, « Structural rationale for the short branched substrate specificity of the glycogen debranching enzyme GlgX », Proteins: Structure, Function, and Bioinformatics, vol. 78, no 8, , p. 1847-1855 (PMID 20187119, DOI 10.1002/prot.22697, lire en ligne)
- (en) Yong Bao, Thomas L. Dawson, Jr. et Yuan-Tsong Chen, « Human Glycogen Debranching Enzyme Gene (AGL): Complete Structural Organization and Characterization of the 5′ Flanking Region », Genomics, vol. 38, no 2, , p. 155-165 (PMID 8954797, DOI 10.1006/geno.1996.0611, lire en ligne)
- (en) Eui-Jeon Woo, Seungjae Lee, Hyunju Cha, Jong-Tae Park, Sei-Mee Yoon, Hyung-Nam Song et Kwan-Hwa Park, « Structural Insight into the Bifunctional Mechanism of the Glycogen-debranching Enzyme TreX from the Archaeon Sulfolobus solfataricus », Journal of Biochemical Chemistry, vol. 283, no 42, , p. 28641-28648 (PMID 18703518, PMCID 2661413, DOI 10.1074/jbc.M802560200, lire en ligne)
- (en) Hironori Hondoh, Wataru Saburi, Haruhide Mori, Masayuki Okuyama, Toshitaka Nakada, Yoshiki Matsuura et Atsuo Kimura, « Substrate Recognition Mechanism of α-1,6-Glucosidic Linkage Hydrolyzing Enzyme, Dextran Glucosidase from Streptococcus mutans », Journal of Molecular Biology, vol. 378, no 4, , p. 913-922 (PMID 18395742, DOI 10.1016/j.jmb.2008.03.016, lire en ligne)
- (en) David Dauvillée, Isabelle S. Kinderf, Zhongyi Li, Behjat Kosar-Hashemi, Michael S. Samuel, Lynette Rampling, Steven Ball et Matthew K. Morell, « Role of the Escherichia coli glgX Gene in Glycogen Metabolism », Journal of Bacteriology, vol. 187, no 4, , p. 1465_1473 (PMID 15687211, PMCID 545640, DOI 10.1128/JB.187.4.1465-1473.2005, lire en ligne)
- (en) Akifumi Nakayama, Keizo Yamamoto et Shiro Tabata, « Identification of the catalytic residues of bifunctional glycogen debranching enzyme », Journal of Biological Chemistry, vol. 276, no 31, , p. 28824-28828 (PMID 11375985, DOI 10.1074/jbc.M102192200, lire en ligne)
- (en) Baiba K. Gillard, Randle C. White, Ralph A. Zingaro et Thomas E. Nelson, « Amylo-1,6-glucosidase/4-alpha-glucanotransferase. Reaction of rabbit muscle debranching enzyme with an active site-directed irreversible inhibitor, 1-S-dimethylarsino-1-thio-β-D-glucopyranoside », Journal of Biological Chemistry, vol. 255, no 18, , p. 8451-8457 (PMID 6447697, lire en ligne)
- (en) Y. T. Chen, J. K. He, J. H. Ding et B. I. Brown, « Glycogen debranching enzyme: purification, antibody characterization, and immunoblot analyses of type III glycogen storage disease », American Journal of Human Genetics, vol. 41, no 6, , p. 1002-1015 (PMID 2961257, PMCID 1684360)

